Практикум №11
Попытка понять взаимосвязь между генами №1: GO-analysis.
Ради формализма (и ''хронометража'') я решил для начала перечислить все гены, которые были мне даны для анализа: ACOT2, ACOT4, ACSBG1, ACSBG2, ACSL1, ACSL3, ACSL4, ACSL5, ACSL6, MEIKIN, SLC27A2.
Копируя наименования генов, я пришёл к осознанию, что большинство из них начинается на AC(''что-то''). Как позже выяснится, речь идёт об Acyl-CoA (ацил-коэнзим А).
Собственно, это была последняя попытка понять какую-то схожесть на уровне ''морфологии'' этих аббревиатур. В дальнейшем эти слова в моей ментальной активности не участвовали и представляли из себя некую зловещую, уходящую корнями вглубь моей любимой, чёрт возьми, лабной рутины символику.
На странице GO-анализа я запустил с опциями ''biological process'' и ''Homo sapiens'' интригующий всех учёных нашей комнаты в ДСЛ анализ.
Результаты смотрите после небольшого рабочего перерыва...
Ой! Мы же не перед кабинетом врача-офтальмолога в 202 поликлинике! Продолжим сейчас же =)
Выдача произведена с учётом поправки на множественное тестирование (включена опция ''Use the Bonferroni correction for multiple testing'').
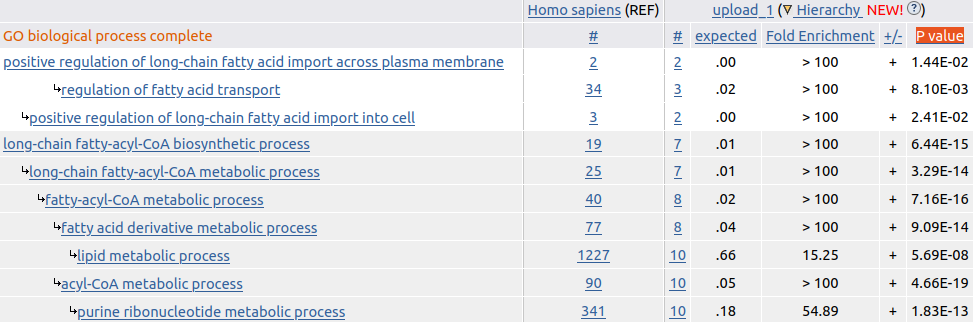
В таблице 1 представлены описания основных колонок выдачи GO-анализа. На рисунке 1 изображена сама выдача:
Название колонки (номер в скобках).
Описание
GO biological process complete (1)
Название группы генов, вовлечённых в некий биологический процесс.
# (2)
Количество всех генов из референсного списка генов (все гены в выбранном организме), участвующих в данном процессе.
# (3)
Количество генов, участвующих в этой категории аннотации данных, из загруженного списка генов.
expected (4)
Количество генов, которое можно было бы ожидать в рамках данной категории из загруженного списка. Считается на основе референсного набора генов.
Fold Enrichment (5)
Отношение числа генов из списка, встретившихся в группе, к ожидаемому числу генов.
+/- (6)
Перепредставленность (+, Fold Enrichment > 1), недопредставленность (-, Fold Enrichment < 1).
P value (7)
P-value, определённое точным тестом Фишера (в моём случае) или биномиальным распределением (при выбранной другой опции).
Если нажать на определённую категорию аннотации (каждый раз думаю, как же это обозвать покрасивее), можно попасть в райский уголок (по мнению многих биоинформатиков), состоящий из десятков тысяч каких-то страшных непонятных определённых к данной группе генов.
А можно не нажимать. Мы ведь и так счастливые люди, особенно под конец семестра =) Зачем нам так много и без того навалом подкатившего после досрока по химии райского наслаждения?
Да, пожалуй мы обойдёмся табличкой в выдаче...
Как можно увидеть, большинство генов в списке так или иначе связаны с биосинтезом ацил-КоА длинноцепочечных жирных кислот (ах, как же скрипит моя голова в попытках вспомнить биохимию с всероса...). Некоторые гены связаны с транспортом (точнее: импортом) длинноцепочечных (C13 - C22, как старательно отмечено в описании группы) жирных кислот через плазматическую мембрану.
Таким образом, можно сказать, что GO-анализ - довольно полезный метод, который незаменим в том случае, когда генов очень-очень много.
Попытка понять взаимосвязь между генами №2: STRING.
Отправив гены в анализ STRING (и выбрав органим Homo sapiens), я столкнулся с тем, что ген (и белок) MEIKIN не встречается в базе данных Homo sapiens. А жаль...
Остальные (найденные) гены приводились с описанием функций кодируемых ими продуктов.
Надо сказать, что мои отношения со STRING не задались были сомнительными с самого начала использования: всегда получались какие-то кривые сети, в которых кроме совместного упоминания практически никогда ничего не встречалось.
В данном же случае даже при minimum required interaction score, равном 0.7 (high confidence: это фактически порог вероятности существования данной связи) получилась высокосвязная сеть, представленная на рисунке 2 сверху. На том же рисунке снизу представлена так называемая физическая подсеть, на которой рёбра изображаются только в случае принадлежности белков к одному и тому же физическому комплексу.
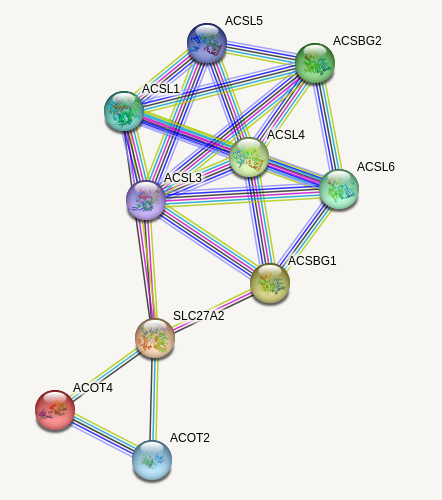
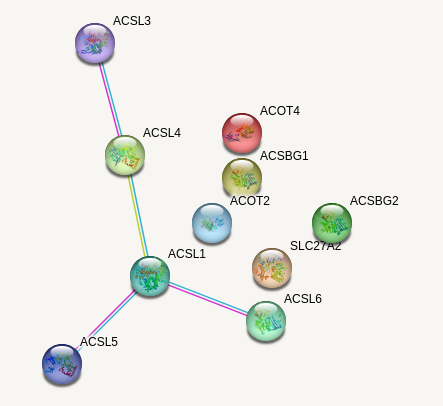
Как и ожидалось (после GO-анализа), сеть, рождённая сервисом STRING, будет не такой посредственной, как это обычно бывает. Рёбра окрашены в зависимости от типа ассоциации (функциональной или физической). Самые ценные связи - синие и фиолетовые - взаимодействия из курируемых баз даных и экспериментально подтверждённые соответственно. Салатовый, например, означает встречаемость в одной статье, что мне всегда казалось не самой значительной ассоциацией =)
В разделе ''Analysis'' приведены:
1) PPI enrichment p-value, указывающий то, насколько сеть случайна (в данном случае p-value довольно низкий, что обозначает крайне низкую вероятность встретить такую же или более... кхм... ''извращённую'' сеть при условии полной случайности генерации сети, что есть нулевая гипотеза).
2) Анализ различных функциональных взаимодействий, в том число и GO анализ обогащения, который подтвердил GO анализ из первого пункта: все белки действительно связаны с метаболизмом длинноцепочечных жирных кислот. GO анализ принадлежности к конкретному клеточному компартменту показал, что 7 белков связаны с эндоплазматическим ретикулумом, 6 - с митохондриями, а 5 - с пероксисомами... Если бы я ещё помнил, что там происходит биохимического с этими жирными кислотами =)
Есть у STRING и другие возможности помимо построения таких сетей: визуализация Cooccurrence (совместного появления) белков среди различных таксонов, Coexpression (коэкспрессия) и другие. Это довольно богатый функционал с красивыми и понятными визуализациями, простота которых не оставит никого равнодушным...
Попытка понять взаимосвязь между генами №3: Reactom.
В результате работы сервиса Reactom получилась следующая карта (рисунок 3):
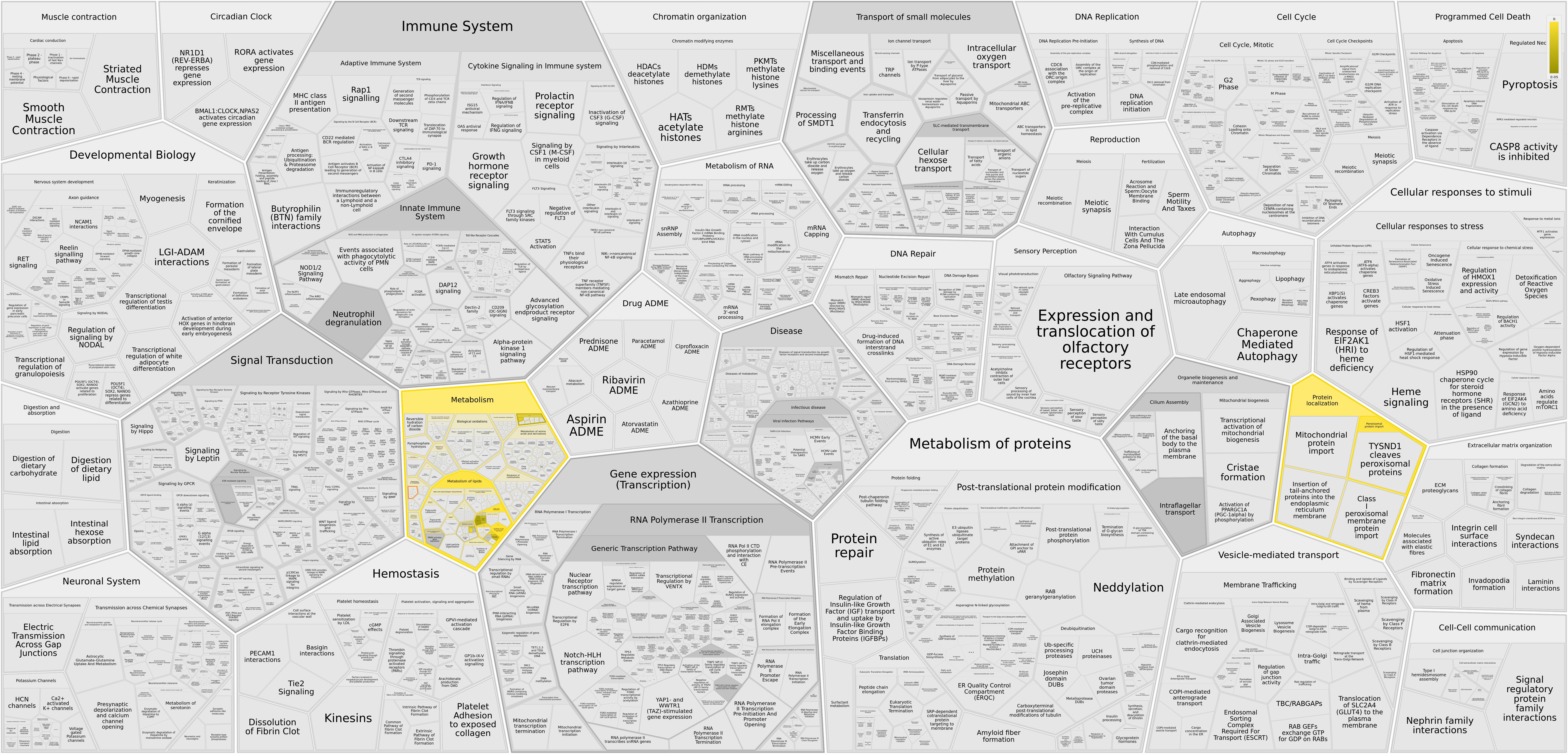
Из неё мы ничего нового не узнаем, поскольку нам уже в третий раз говорят о метаболизме липидов и транспорте жирных кислот внутрь клетки.
Интерфейс Reactom - это что-то с чем-то. Такого обилия кнопок не наблюдал ни на одном из ранее увиденных сайтов. Так или иначе после была получена схема метаболизма жирных кислот, поскольку именно этот путь был в очередной раз перепредставлен входными генами. Таким образом также в очередной раз была подтверждена локализация продуктов данных генов: они все так или иначе связаны с ЭПР, митохондриями и пероксисомами.
Было показано также, что один из генов связан неким образом с вирусными заболеваниями, а именно с вирусом иммунодефицита человека.
Наконец, в карте биосинтеза ацил-КоА я, наконец, нашёл конкретный ген и посмотрел его экспрессию в различных тканях.
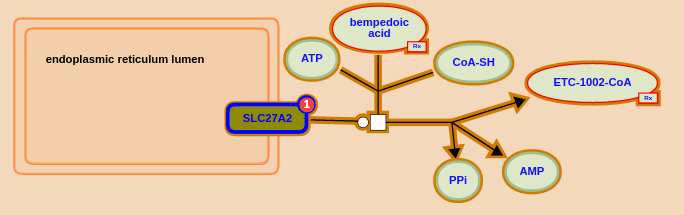
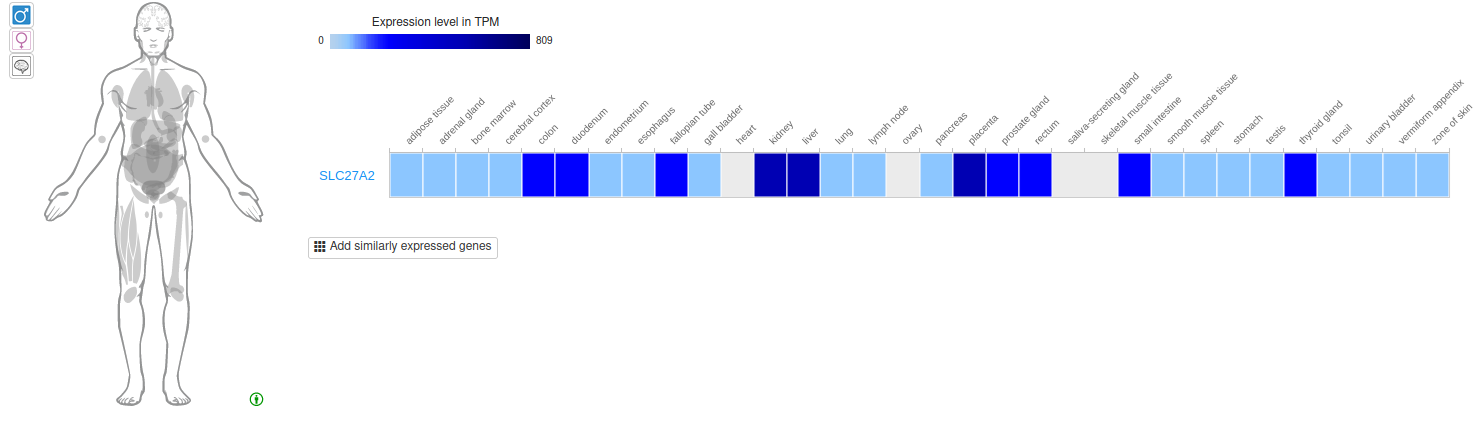
Как оказалось, этот ген, SLC27A2, более всего экспрессируется в почках, лёгких и плаценте, что, наверное, логично для фермента, связанного с биосинтезом жирных кислот =)
Таким образом, Reactom - это ещё один сервис, которые позволяет многое узнать о данном наборе геном. При этом можно сказать, что он предоставляет самые красивые визуализации найденной информации. К сожалению, правда, сайт имеет довольно перегруженый интерфейс, в котором довольно тяжко разбираться. Но это нисколько не принижает ценности этого ресурса.
Кирилл Кузенков, студент второго курса ФББ