Практикум №12
Задание №1: Знакомство с базой данных OPM.
Характеристики данного белка.
Для выполнения практикума мне был предложен белок с идентификатором MEPRM_MYCTU. Это белок, который, вероятно, входит в структуру транспортного комплекса ABC и каким-то образом участвует в формировании резистентности к некоторым лекарствам.
Последовательность белка в fasta-формате: 005318.fasta.
PDB-файл с предсказанной с помощью AlphaFold структурой: AF-O05318-F1-model_v4.pdb.
База данных OPM.
В базе данных OPM был найден белок, являющийся рецептором специфического сидерофора энтеробактина, улавливающего трёхвалентные ионы железа. Белок содержит β-бочонок из 22 трансмембранных β-листов. Рецептор локализован в наружной мембране отрицательной по Граму бактерии Pseudomonas aeruginosa.
Идентификатор PDB: 5NC4
Идентификатор Uniprot: PFEA_PSEAE
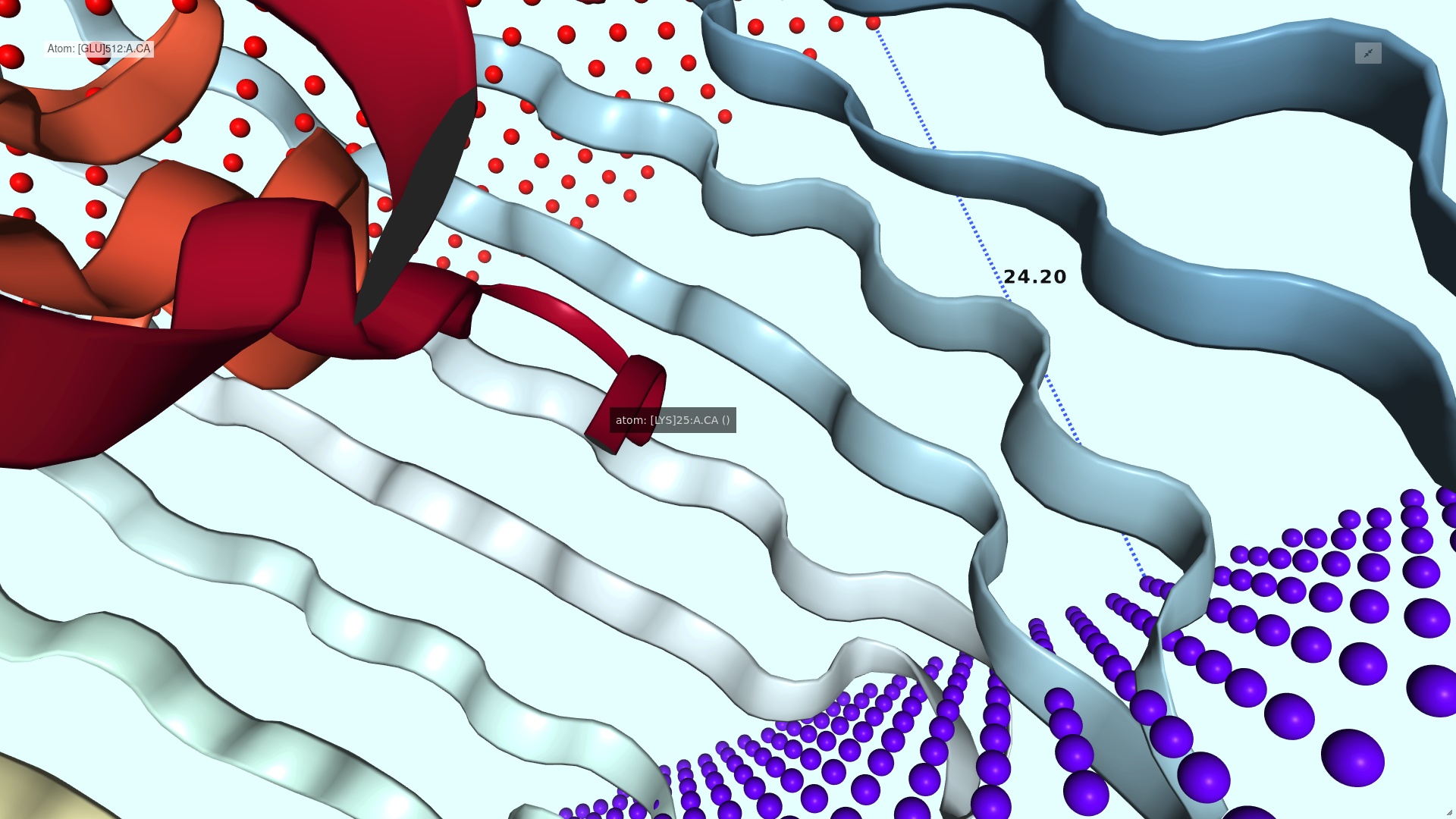
На рисунке 1 представлена трёхмерная визуализации единственное субъединицы белка с помощью пакета nglview в Python. Красным цветом в стиле ''ball+stick'' отмечена положительно заряженная сторона мембраны, синим - отрицательно заряженная. В стиле ''cartoon'' отмечена поверхность белка, встроенного в мембрану. Разными цветами отмечены разные β-листы.
Толщина гидрофобной части белка в мембране.
24.2 Å
Координаты трансмембранных участков.
1( 155- 164), 2( 174- 184), 3( 191- 198), 4( 233- 242), 5( 249- 255), 6( 290- 297), 7( 304- 310), 8( 346- 355), 9( 361- 369),10( 407- 416),11( 421- 429),12( 439- 447),13( 455- 461),14( 506- 514),15( 517- 525),16( 568- 575),17( 583- 589),18( 609- 616),19( 624- 630),20( 659- 667),21( 674- 678),22( 711- 719)
Среднее количество остатков в одном β-тяже белка.
8
Мембрана, в которую встроен белок.
Внешняя мембрана Грам-отрицательной бактерии.
Задание №2: DeepTMHMM. Предсказание трансмембранных элементов по последовательности белка.
Текстовая выдача предсказания трансмембранных элементов альфа-спирального белка: MEPRM_MYCTU.md
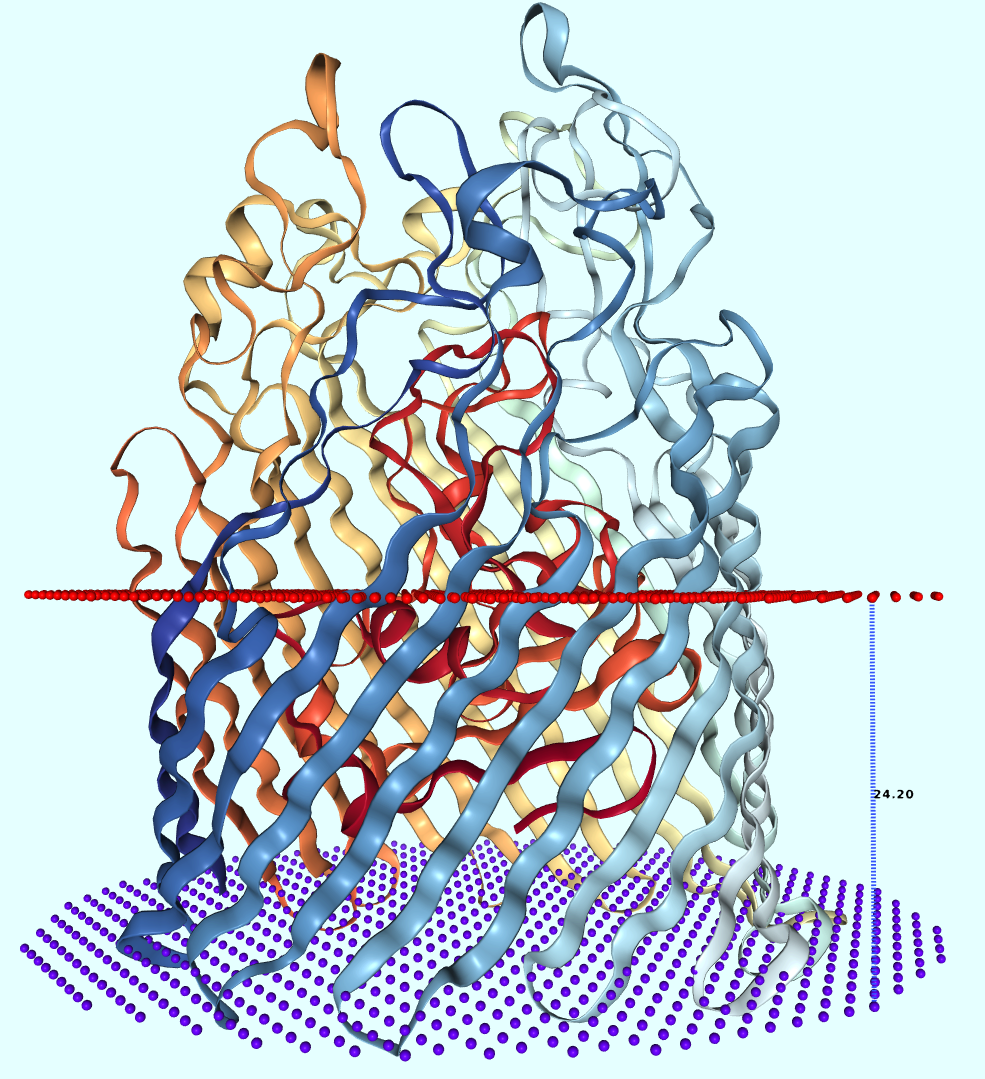
На рисунке 2 представлен график предсказания трансмембранных элементов белка MEPRM_MYCTU: по оси ''X'' отмечены координаты - номера аминокислотных остатков, а по оси ''Y'' отмечена вероятность предсказания локализации. На небольшом горизонтальном рисунке сверху изображён профиль предсказаний: красным отмечены вероятные трансмембранные элементы, розовым - участки, располагающиеся вне мембраны с внутренней её стороны, а синим - с внешней её стороны. Можно заметить, что и N-конец, и C-конец располагаются с внутренней относительно мембраны стороны.
Текстовая выдача предсказания трансмембранных элементов белка β-бочонка: PFEA_PSEAE.md
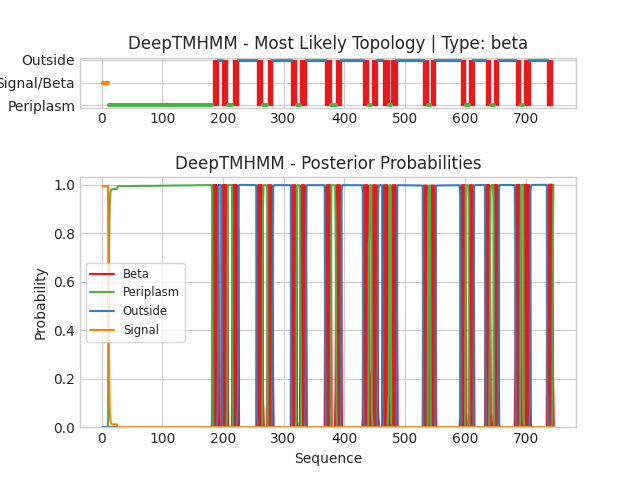
На рисунке 3 представлен график предсказания трансмембранных структур β-бочоночного белка PFEA_PSEAE. На вероятностном профиле локализации сверху красным, синим и зелёным цветами отмечены те же локализации, что и в случае альфа-спирального белка. Оранжевым отмечен сигнал локализации (во внешней мембране Грам-отрицательной бактерии?). Таким образом, было предсказано 22 трансмембранных бета-листа, что соответствует PDB-визуализации.
Предсказание локализации структур β-бочоночного белка довольно точно соответствует реальной PDB-визуализации. N-конец белка, судя по профилю локализации, должен располагаться в периплазме (с отрицательной стороны мембраны). C-конец должен входить в трансмембранный участок.
Найдя N-конец на структуре PDB, я заметил, что он находится в трансмембранном участке, а не в периплазме, как было предсказано.
C-конец входит в трансмембранный участок, как и было предсказано (изображение не приведено).
Задание №3: PPM. Предсказание положения выданного белка в мембране.
С помощью сервиса PPM (версия PPM 3.0) по pdb-файлу белка MEPRM_MYCTU было получено предсказание положения данного в мембране.
Были выбраны следующие настройки алгоритма:
Type of membrane.
Gram-positive bacteria inner membrane
Allow curvature.
no
Topology (N-ter).
in
Тип мембраны выбран в соответствии с таксономическим положением данной бактерии. Mycobacterium tuberculosis - Грам-положительная бактерия (формально) с одной мембраной.
Предположение о локализации N-конца было взято из предсказания, полученного с помощью DeepTMHMM во втором задании.
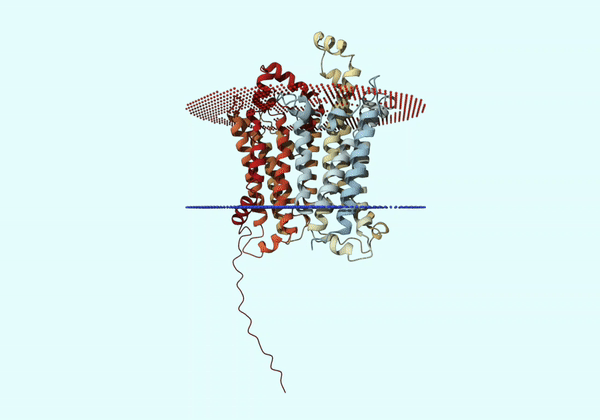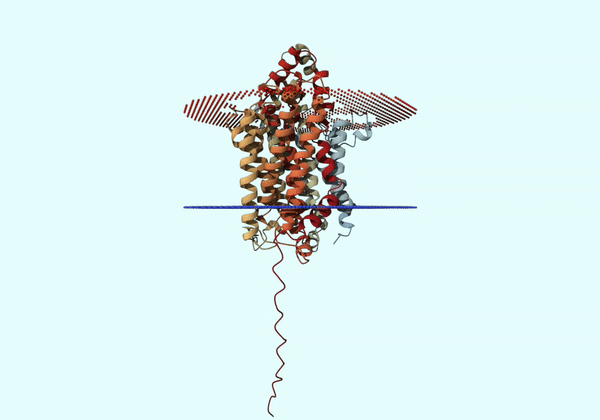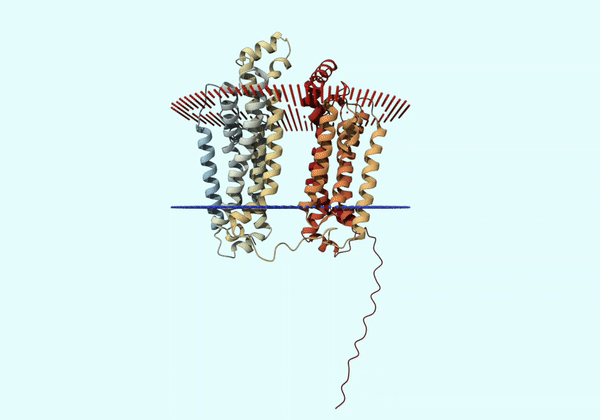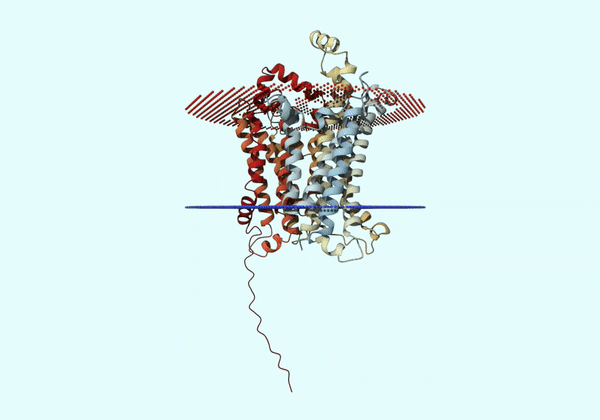
Толщина гидрофобной части белка в мембране.
30.5 ± 0.8 Å
Координаты трансмембранных участков.
1( 33- 56), 2( 102- 121), 3( 147- 169), 4( 175- 198), 5( 206- 228), 6( 253- 270), 7( 307- 327), 8( 362- 383), 9( 406- 431),10( 443- 471),11( 474- 492),12( 521- 539)
Среднее количество остатков в одной трансмембранной альфа-спирали белка.
22
Мембрана, в которую встроен белок.
Внутренняя мембрана Грам-положительной бактерии.
Задание №4: Сравнение алгоритмов предсказания трансмембранных спиралей.
Сравним результаты двух предсказаний (DeepTMHMM и PPM) локализации белка MEPRM_MYCTU:
В целом результаты очень схожи. И то, и другое предсказания говорят о том, что в выданном белке 12 трансмембранных альфа-спиралей, а N- и C-концы имеют внутреннюю (относительно мембраны) локализацию. Особенно хорошо это видно для N-конца на рисунке 5 (он довольно значительно выступает из мембраны, как и показано на вероятностном профиле, полученном во втором задании). Тем не менее стоит отметить, что N-конец (вплоть до 19 аминокислотного остатка) имеет низкую (''Low'') достоверность в предсказании модели AlphaFold (''Model Confidence''), и это могло бы повлиять на результаты предскания PPM.
Можно заметить, что сервис PPM расположил две небольшие альфа-спирали с внешней (относительно мембраны) стороны, и ещё две - с внутренней. Эти альфа-спирали совсем не видны в предсказании, полученном с помощью DeepTMHMM, и являются единственным отличием в работе двух сервисов. При этом эти ''мини-спирали'' имеют довольно высокую достоверность предсказания (По большей части ''Confident'' и ''Very high'') AlphaFold, и это подчёркивает данное несоответствие в двух предсказаниях.
В свою очередь, сравнение pdb-структуры белка PFEA_PSEAE с предсказанием, полученным с помощью DeepTMHMM, говорит о том, что по большей части предсказание достоверно описывает реальность: оно предсказало 22 трансмембранных β-листа.
В то же время предсказание включало в себя длинный N-конец, локализованный в периплазме, несмотря на то, что на самом деле он практически не выступает из мембраны (было показано на рисунке 4).
Исходя из полученных результатов, можно сделать выводы, что сервисы делают довольно хорошие предсказания, но в деталях могут ошибаться.
Кирилл Кузенков, студент второго курса ФББ