Практикум №4
Задание №1. Составление списка гомологичных белков, включающих паралоги.
Поиск гомологов.
Скачав из соответствующего репозитория протеомы выбранных ранее бактерий, я объединил их в единый файл database.fasta, после чего проиндексировал его с помощью следующей команды:
makeblastdb -in database.fasta -dbtype prot -parse_seqids
Далее я с помощью локального запуска blastp нашёл гомологи выданного белка CLPX_ECOLI в сформированной базе данных из протеомов, поставив порог на e-value в 1e-3:
blastp -query P0A6H1.fasta -db database.fasta -out final.tab -evalue 0.001 -outfmt 7
При вожу выдачу программы без заголовка и выравниваний: final.tab.
Задание №2 Реконструкция и визуализация.
После этого я объединил все находки в единый fasta-файл, сохранив при этом в заголовках только идентификаторы.
На онлайн-ресурсе NGPhylogeny я запустил филогенетическую реконструкию дерева, использовав в качестве программы множественного выравнивания MAFFT, а в качестве алгоритма построения дерева - FastME.
При этом алгоритм FastME использовался без опции ''Gamma distributed rates across sites'' (моделирование скорости замен в различных сайтах), поскольку она часто замедляет работу программы и не улучшает её эффективности. В качестве начального дерево было использовано дерево, сконструированное посредством алгоритма BIONJ. Также был использован bootstrap с количеством реплик, равным 100.
Полученное дерево в формате Newick сохранено в файле Output_Tree_finds.nhx.
Данные реконструкции.
Основываясь на предположении, что филогенетическая реконструкция верна, можно найти некоторую информацию о парах ортологов и паралогов.
Ортологи
Паралоги
CLPX RHOJR и CLPX MYCTU
Q0S8C7 RHOJR и Q0S6Y7 RHOJR
FTSH MYCTU и Q47KU4 THEFY
Q1AY82 RUBXD и Q1AU05 RUBXD
Q0S8C7 RHOJR и Q82EB8 STRAW
Q82QV8 STRAW и Q82EE9 STRAW
Далее я укоренил дерево в среднюю точку и покрасил в различные цвета получившиеся ортологичные группы белков (Рис. 1). Группы, состоящие из небольшого количества ортологов, покрашены в чёрный цвет. Также было добавлено отображение поддержек bootstrap (цифры, приведённые под ветвями).
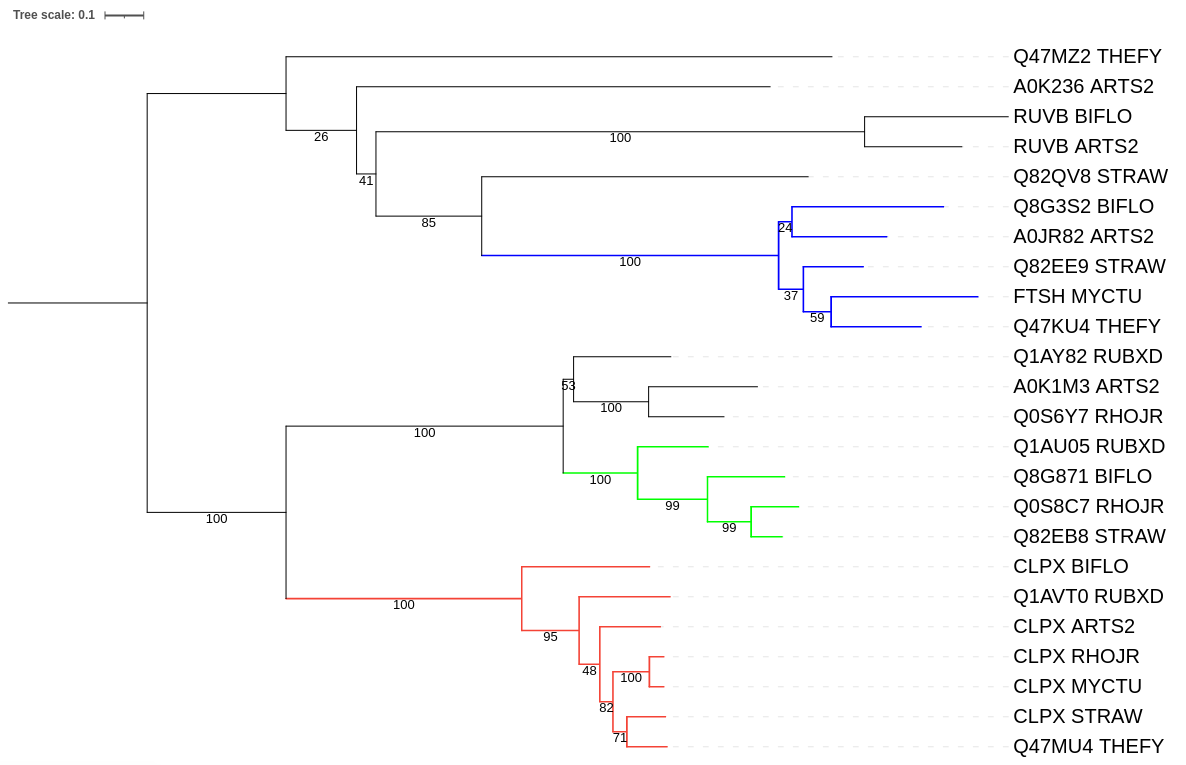
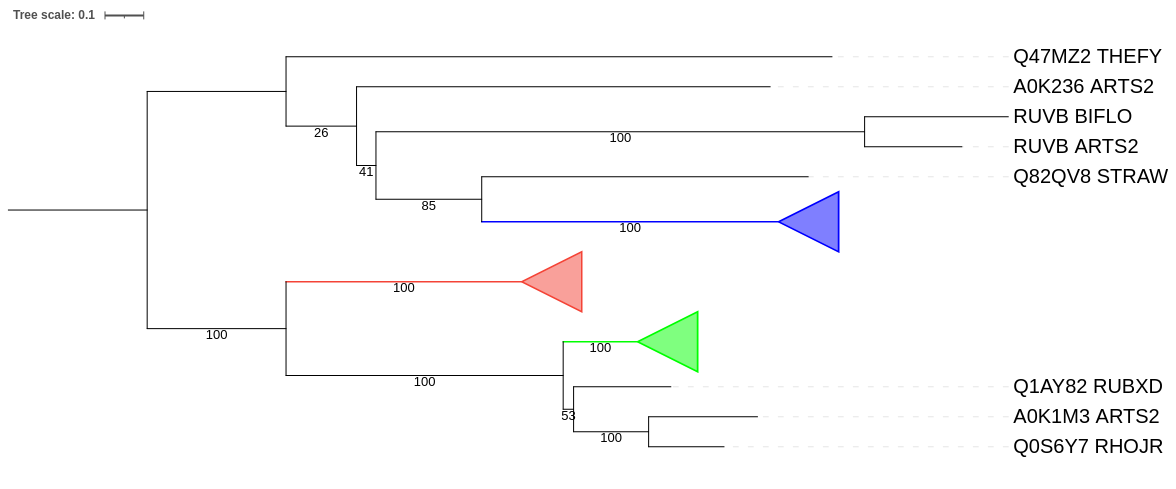
Далее ортологичные группы были ''схлопнуты'' (Рис. 2).
Кирилл Кузенков, студент второго курса ФББ