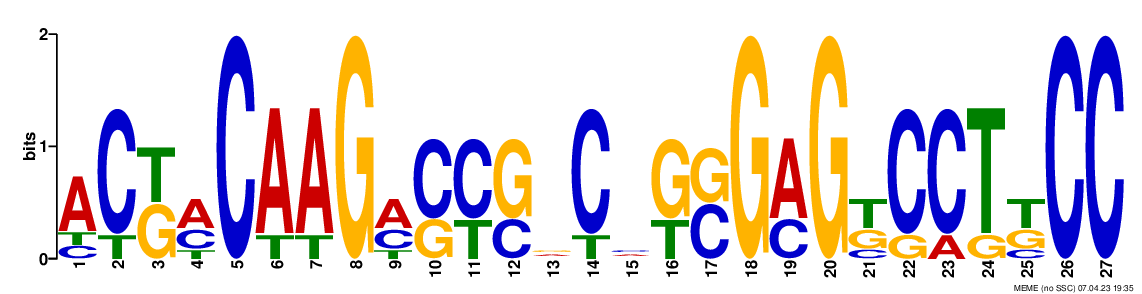Практикум №7
Поиск оперонов в геноме P. aeruginosa.
Сервис Operon-mapper.
Для поиска оперонов в геноме я выбрал штамм PAO1 бактерии Pseudomonas aeruginosa. Сборку можно скачать здесь: sequence.fasta.
Непосредственно для поиска оперонов использовался web-сервис Operon-mapper. На вход подавались:
1) Геном Pseudomonas aeruginosa в формате fasta: sequence.fasta.
2) Файл с координатами ORF (open reading frame) в формате gff: sequence.gff3.
Файл, полученный на выходе, содержит список найденных оперонов: List_of_operons. При этом в выдачу также были включены ID кластеров ортологичных генов (COG-и) и функциональная аннотация.
Дальнейшие действия подробно описаны в коде. Из списка оперонов была получена таблица с характеристиками первых CDS всех оперонов (ведь именно перед первым CDS непосредственно находится промотор), а далее - таблица со всеми функционально аннотированными первыми CDS-ами (поскольку необходимо было отбирать housekeeping гены). Затем из последней таблицы были найдены те CDS, которые входят в группу housekeeping (получилось 257 оперонов). Далее были извлечены следующие данные:
1) Тренировочный набор (Train.fasta), составленный из 60 последовательностей промоторов оперонов. Последовательности представляли из себя фрагменты длиной в 150 пар оснований, предшествующие первым CDS оперонов: то есть имели координаты (для + цепи) start = начало первого CDS - 150, end = начало перого CDS). При этом гены из группы housekeeping:
A) Транскрипционные факторы,
B) Принимающие участие в трансляции, поддерживающие структуру рибосом и т.д. (категория [J] COGs),
C) ДНК и РНК полимеразы,
D) Связанные так или иначе с биоэнергетикой (дыхательной цепью),
E) P450,
F) АТФ-азы.
2) Тестовый набор (Test.fasta), сформированный из всех остальных последовательностей найденных промоторов в геноме бактерии (получилось 2947 последовательностей).
3) Негативный контроль (Negative.fasta), который был создан путён вырезания случайной последовательности длины 150 в геноме бактерии таким образом, чтобы эта последовательность не пересекалась со всеми найденными промоторами. Выборка была взята размером, равным размеру тестового набора (то есть из 2947 последовательностей).
Запуск MEME.
Затем был произведён локальный запуск программы MEME на kodomo с помощью следующей команды:
meme Train.fasta -dna -nmotifs 3 -minw 6
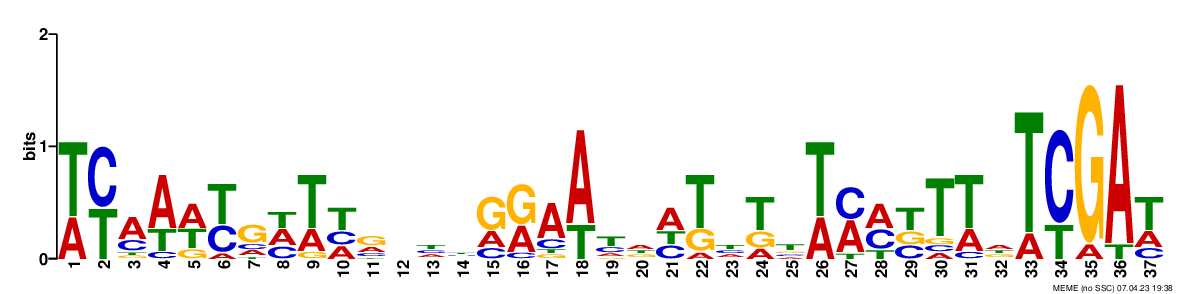
Выход программы: meme.txt. Было найдено три мотива (в соответствии с запросом): logo1 (E-value = 1.7e-003), logo2 (E-value = 1.6e+002) и logo3 (E-value = 6.1e+002). Они представлены, соответственно, на рисунках 1, 2 и 3. Только первый мотив проходит общепринятый порог значимости, равный 0.05 (соответствующее E-value меньше 0.05), поэтому решено было далее работать с ним. Остальные мотивы имеют гигантские E-value и слишком маленькую длину, которая не характерна боксу Прибнова.
Запуск FIMO.
Запуск FIMO осуществлялся по следующей команде:
fimo -thresh 0.001 meme.txt Test.fasta
При пороге в 0.01 нашлось 1708 находок в 859 последовательностях (оперонов). При этом в опероне №2137 встретилось целых 16 искомых мотивов, хотя должен быть один. Это говорит о том, что порог следовало понизить.
При пороге в 0.001 нашлось 130 находок в 148 последовательностях, что говорит о малом числе ложных находок. Этот порог и был выбран в качестве основного.
Далее я проверил при выбранном пороге количество находок, которое будет найдено в негативном контроле, с помощью следующей команды:
fimo -thresh 0.001 meme.txt Negative.fasta
При этом нашлось 40 мотивов в 33 последовательностях, что значимо меньше, чем в тестовом наборе.
Кирилл Кузенков, студент второго курса ФББ