Практикум №9
Задание №1
Поиск домена и доменной архитектуры.
Для того, чтобы грамотно произвести отбор домена, я написал код (используемый и в последующих этапах задания).
Для построения HMM профиля был выбран домен FN3_7 (PF18447) со следующими необходимыми данными:
Number in seed
Number in full
Number in UniProt
HMM length
15
220
493
97
Далее была отобрана архитектура IL7RA_MACFA (Q38IC7), состоящая из двух доменов: FN3_7, fn3 (порядок сохранён). Архитектура включает в себя 102. последовательности.
Далее были скачаны полные последовательности из выборки full в формате fasta: FN3_7_full-120.fasta. Параллельно была создана таблица с AC всех последовательностей из full со столбцом "Belonging to architecture" из boolean-значений: Table_scores.csv. True означает, что соответствующий белок входит в выбранную архитектуру. False, соответственно, означает обратное.
Позже в ходе выполнения задания в таблицу добавлялись новые столбцы: 1) Belonging to alignment for HMM - boolean-значения: True, если соответствующая последовательность входит в выравнивание для построение HMM-профиля. 2) score - вес, выдаваемый алгоритмом HMM. 3) E-value: E-value, выдаваемое алгоритмом HMM, 4) Sensitivity, 1 - Specificity и F1-score - значения соответствующих метрик для каждого значения веса последовательности.
Был создан отдельный файл в fasta-формате из последовательностей с выбранной архитектурой: arc.fasta. Эти последовательности были выровнены в Jalview с помощью Muscle с параметрами по умолчанию. Промежуточный файл с выравниванием: arc_aligned.fasta. После этого была произведена ревизия полученного выравнивания: удаление N-концевого участка, исключение из выравнивания некоторых наиболее отличающихся последовательностей. Также была проведена процедура удаление избыточных последовательностей на уровне идентичности 90%. В итоге получилось готовое для подачи в алгоритм выравнивание: arc_aligned2.fasta, состоящее из 12 последовательностей.
Создание HMM-профиля.
Для создания профиля, калибровки и поиска по профилю (по умолчанию стоит порог на E-value, равный 10) использовались следующие команды:
hmm2build hmm_profile.out arc_aligned2.fasta
hmm2calibrate hmm_profile.out
hmm2search --cpu 1 hmm_profile.out FN3_7_full-102.fasta > output.txt
Полученный профиль после калибровки: hmm_profile.txt.
Файл с результатами работы HMMsearch: HMMsearch_output.txt.
Поиск порога.
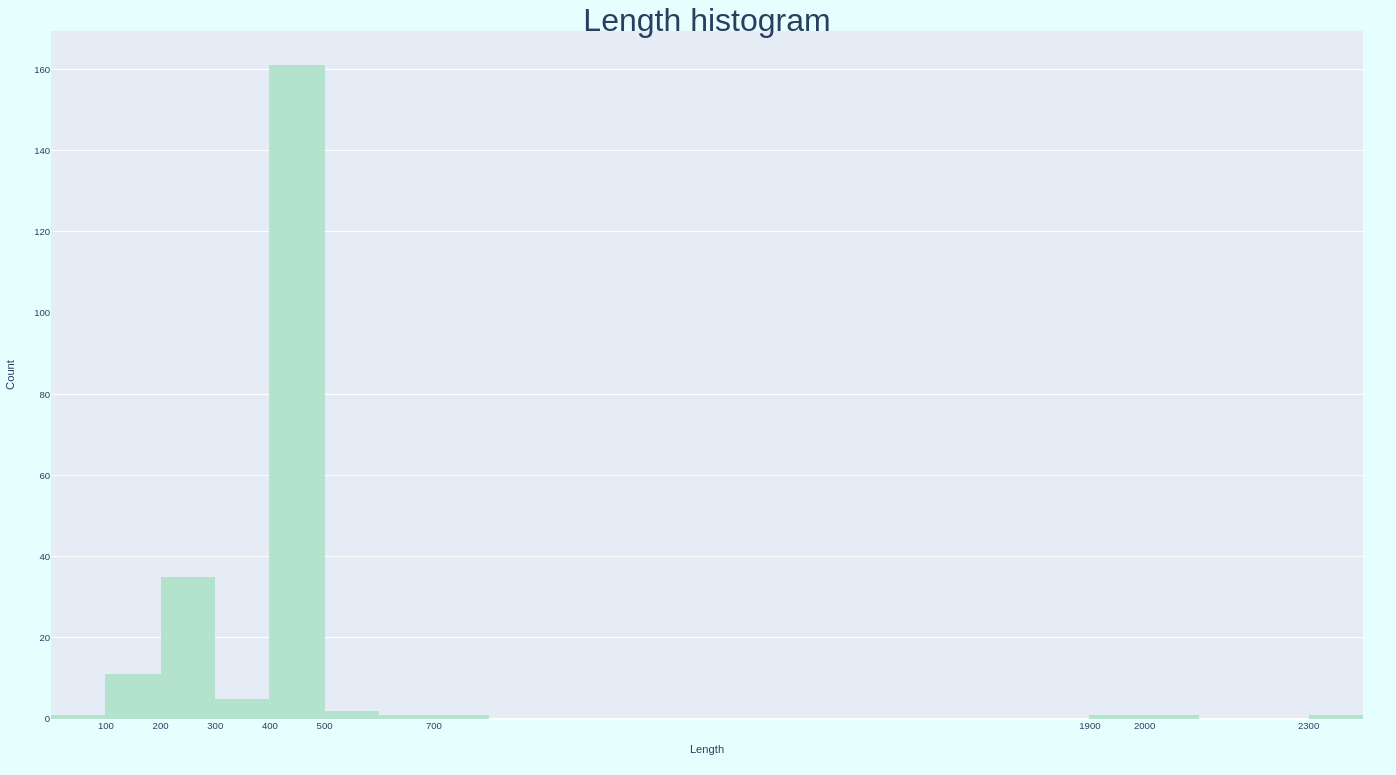
Далее была построена гистограмма распределения длин белков, входящих в выбранное семейство (Рис. 1). Можно сделать вывод, что характерный интервал длин для данного семейства: 100-500.
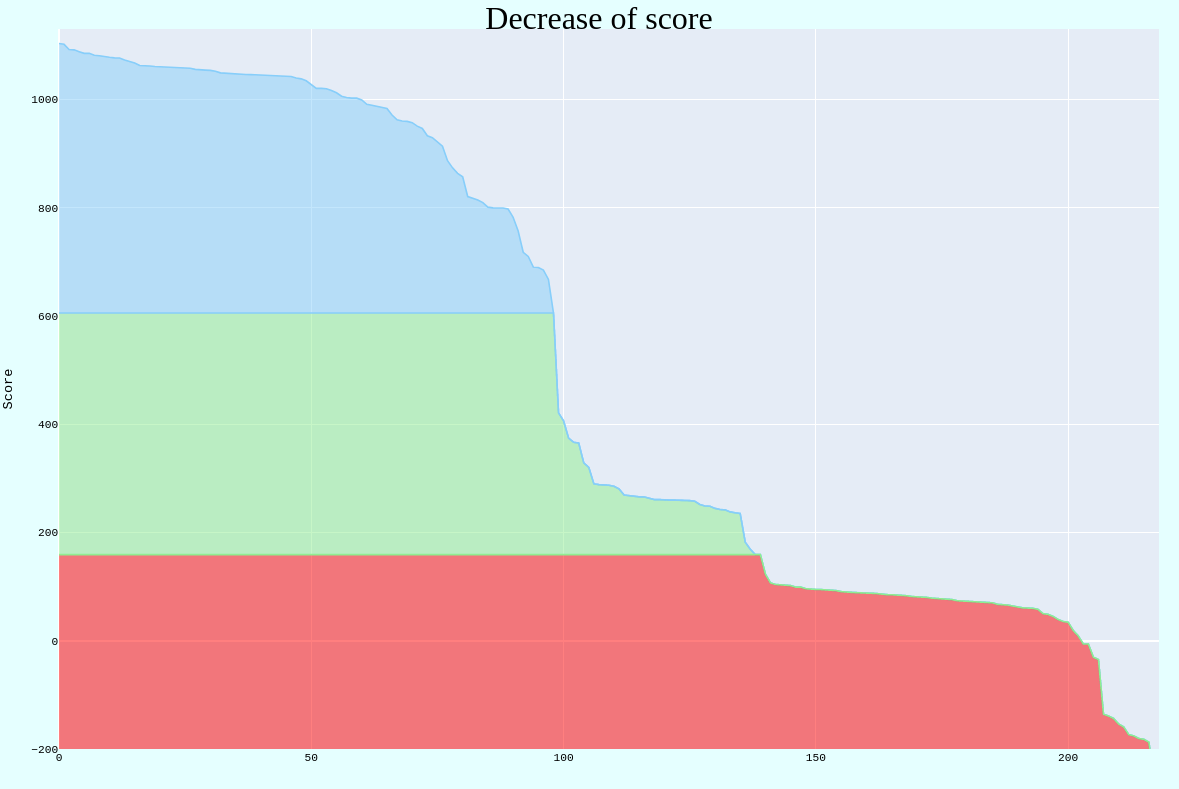
После был построен график падения весов последовательностей (Рис. 2) Разными цветами отмечены разные границы, разделяющие последовательности на те, которые содержат выбранный домен, и те, которые не содержат.
При этом красной цветом отмечена граница весов, отобранная с помощью F1-scores: вес, равный 159, соответствовал минимальному значению F1-score. Зелёным отмечен порог, которая я ранее хотел выделять на основе взгляда на гистограму.
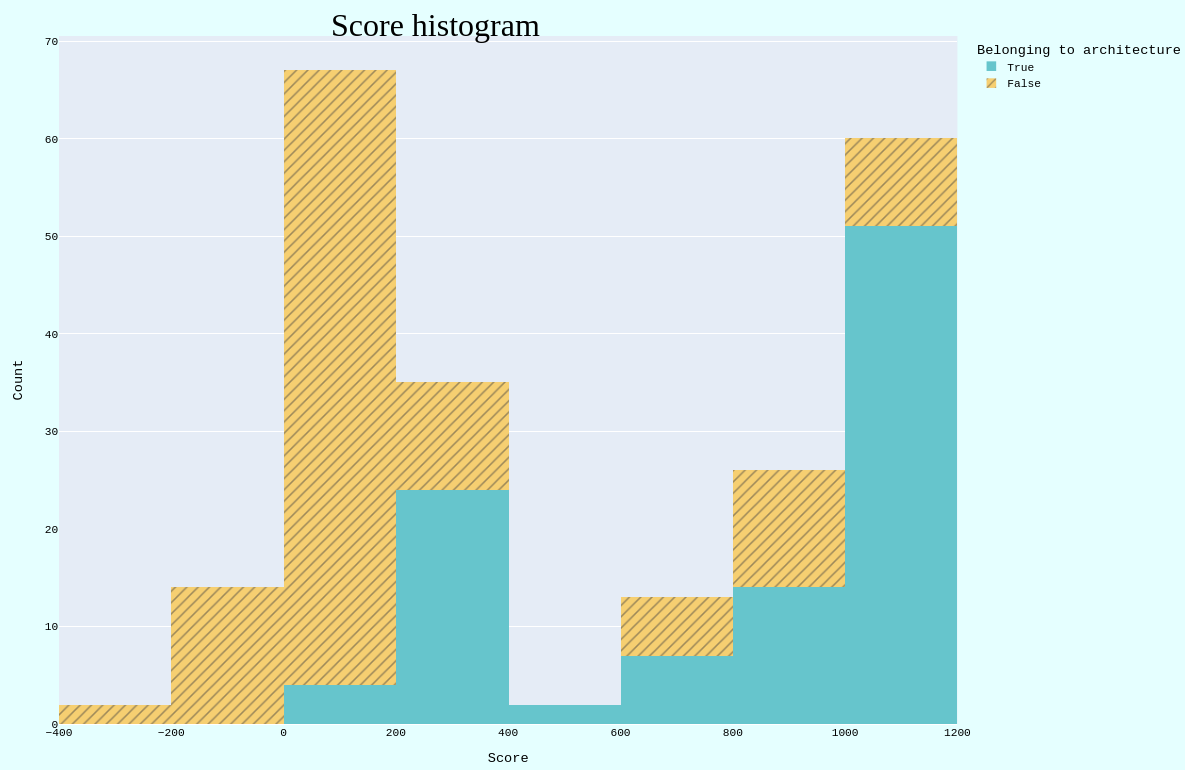
Вдобавок была построена гистрограма распределения весов (Рис. 3) для того, чтобы можно было определить пороговый вес легче. Бирюзовым цветом выделена группа последовательностей, содержащих выбранную архитектуру. Жёлтым, соответственно, выделены остальные последовательности.
При этом красной цветом отмечена граница весов, отобранная с помощью F1-scores: вес, равный 159, соответствовал минимальному значению F1-score. Зелёным отмечен порог, которая я ранее хотел выделять на основе взгляда на гистограму.
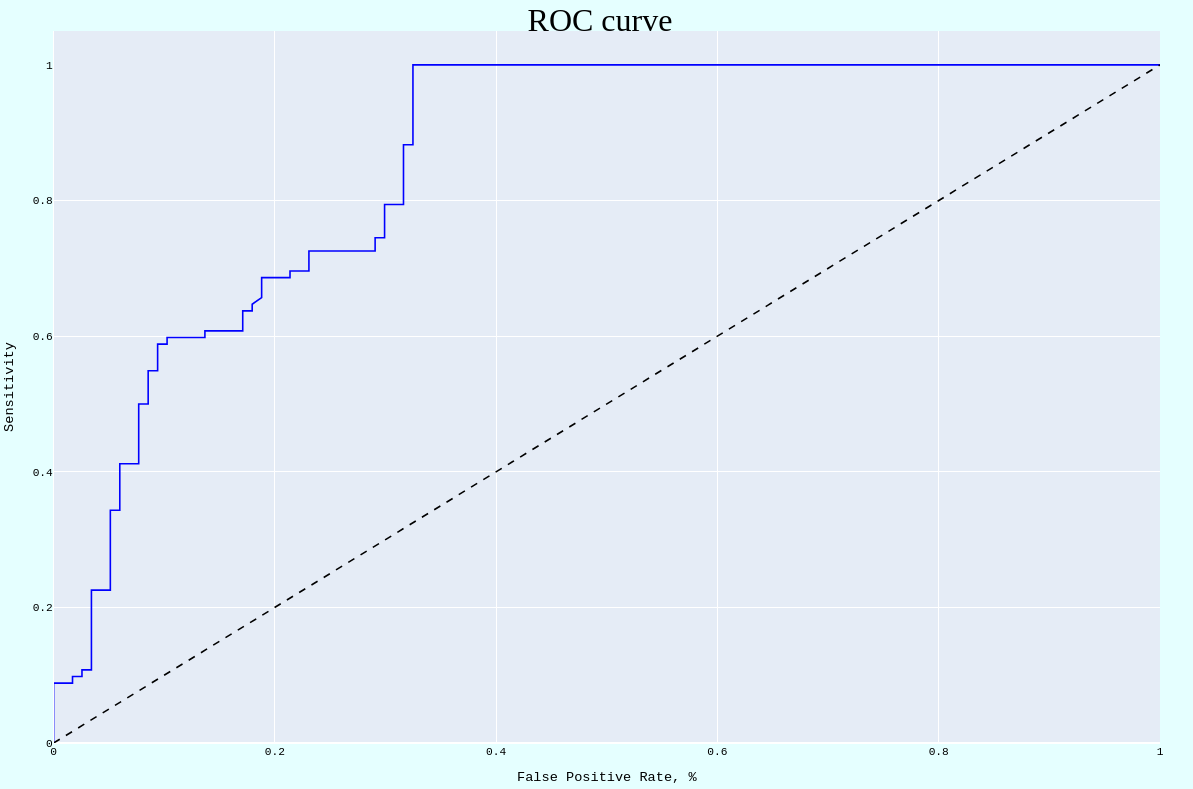
После была построена ROC-кривая (Рис. 4). Площадь под кривой оказалась равно 0.86, что свидетельствует о неплохом качестве классификации.
Порог, полученный при минимизации F1-score: 159.
Кирилл Кузенков, студент второго курса ФББ