Практикум №2
Задание №1: ЭП: хорошая и плохая расшифровки.
В первом задании предстоит оценить качество двух выданных структур (PDB1: 4X5P; PDB2: 5L4V) лектинового домена двухдоменного белка FimH, использующегося патогенными штамами E. coli для адгезии к эпителии мочевыводящих путей [1].
Открыв соответствующие структуры в PyMol, я заметил, что структура с PDB 5L4V в пределах кристаллической ячейки содержит 3 полипептидные цепи, несмотря на то, что биологическая сборка представляет из себя мономер (и это уже может натолкнуть на мысли о качестве...)
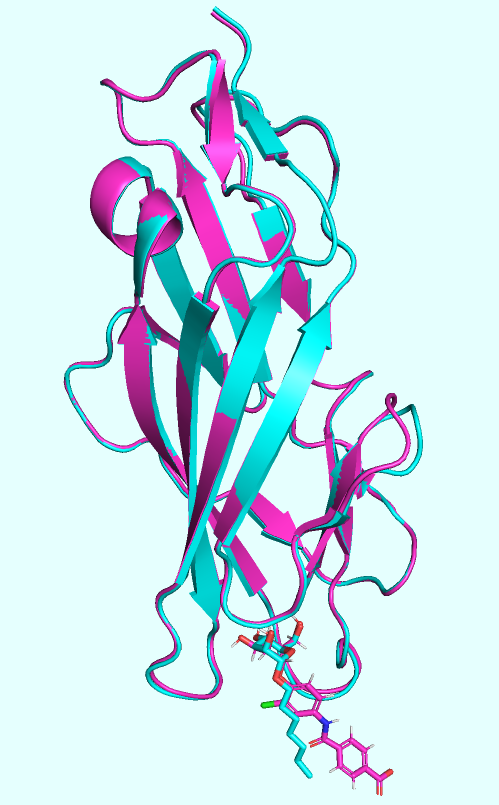
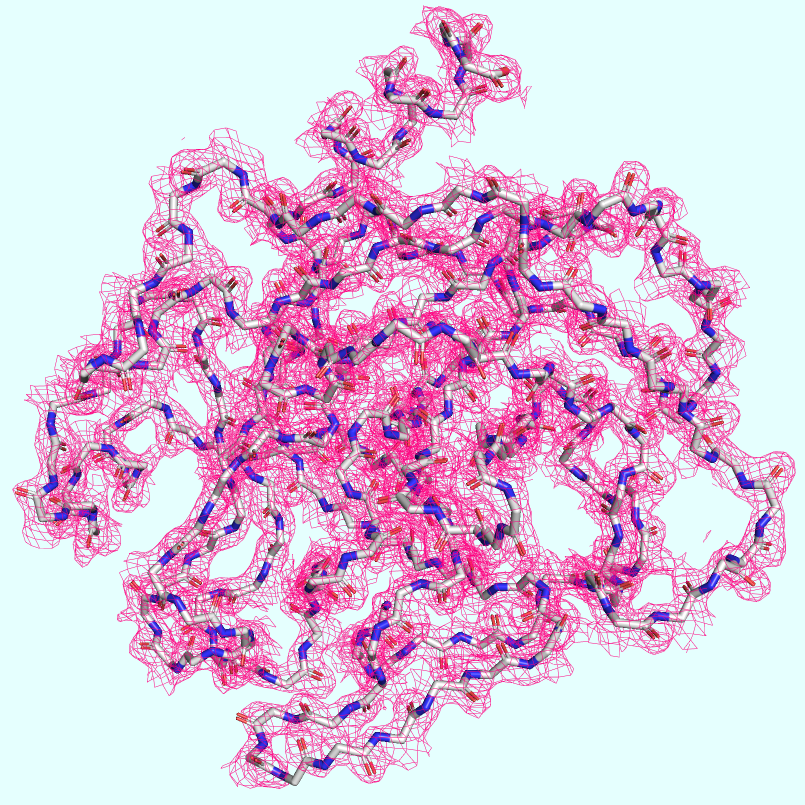
Для удобства предварительного сравнения (до рассмотрения ЭП) пептидных цепей, визуализированных в виде "cartoon", была использована функция структурного выравнивания (align) с 5 циклами уточнения (Рис. 1).
Результат проделанной процедуры наводит на мысли о том, что структуры доменов практически идентичны (исключая некоторые детали расположения неструктурированных петель). Отличия имеются лишь в том, какие лиганды связывают эти домены, что, однако, ничего не говорит о соответствующих качествах.
Таким образом, для того, чтобы сделать более весомые выводы о качестве структур, необходимо рассмотреть их ЭП.
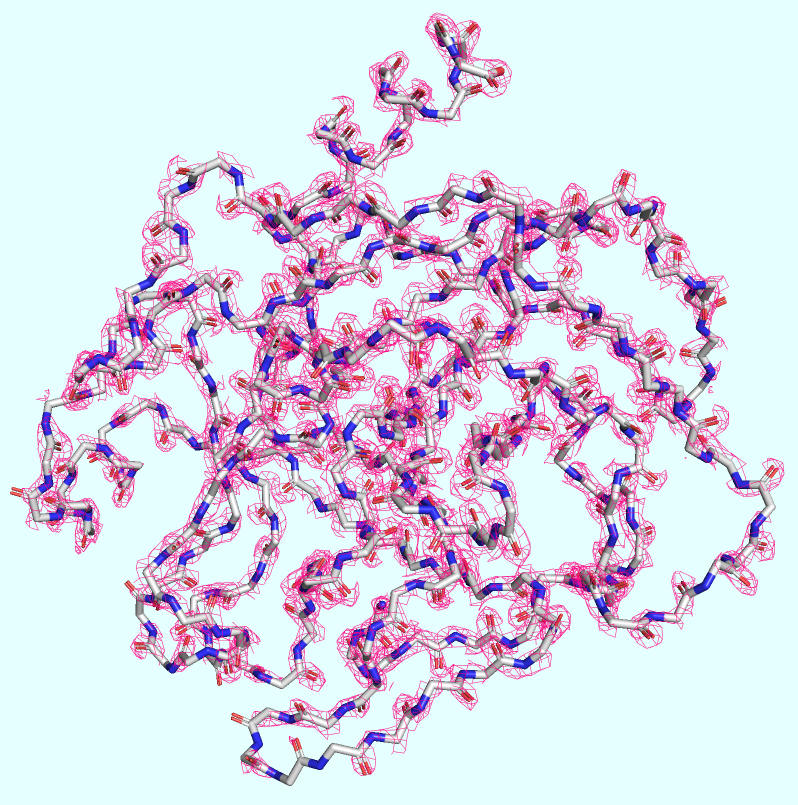
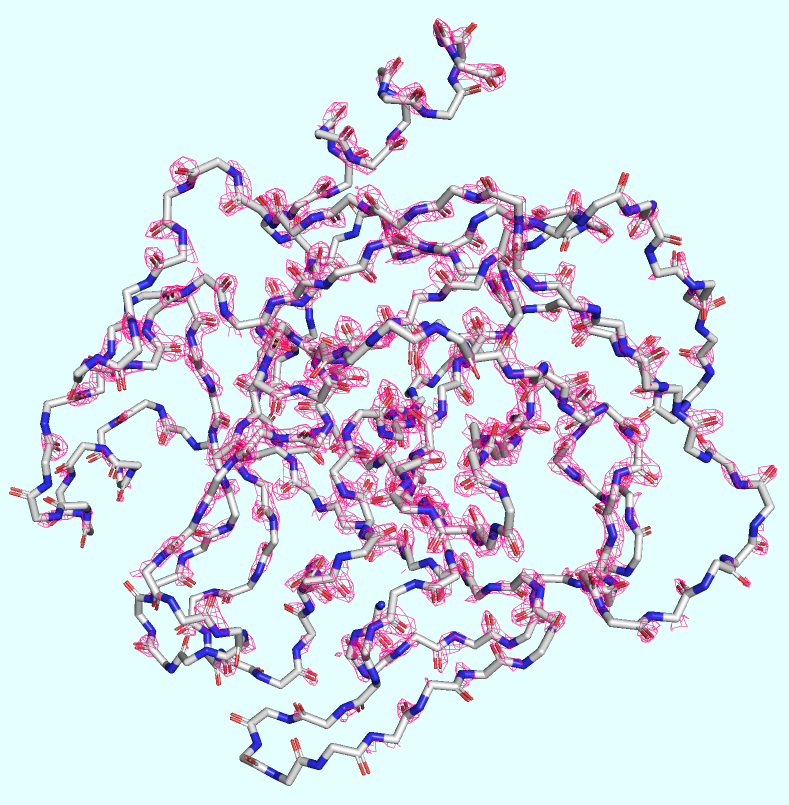
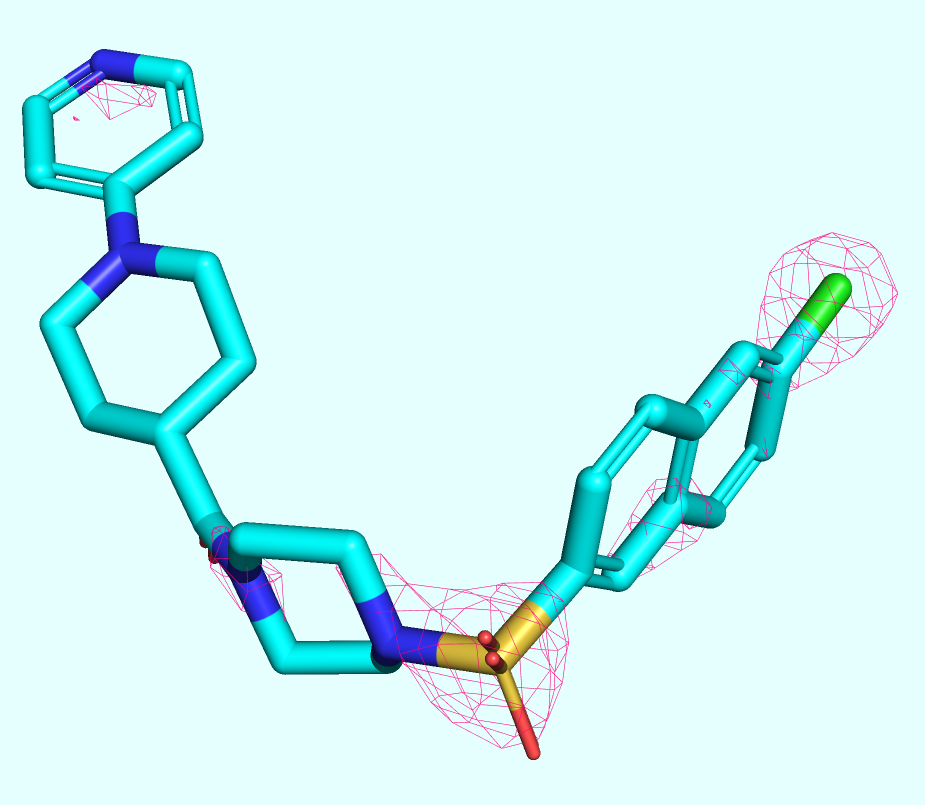
Для того, чтобы явно продемонстрировать отличия, был выбран участок полипептидного остова в районе сайта связывания лиганда (лиганды разные, но связываются в одном и том же сайте связывания). ЭП была визуализирована в виде mesh с уровнем подрезки, равным 1 std. Значения параметра carve были выбраны: 1 Å для 4X5P и 2 Å для 5L4V (для максимизации качества покрытия ЭП выбранных участков и минимизации попадания ЭП боковых радикалов).
В случае модели 4X5P (Рис. 2) наблюдается качественное покрытие ЭП, отдельные облака которой не только описывают форму и направление движения остова белка, но и достаточно чётко соответствуют отдельным атомам, которые в них вписаны. Можно обратить внимание на то, что, по-видимому, точности покрытия ЭП было достаточно для того, чтобы отобразить водороды, связанные с азотами пептидных связей.
Напротив, ЭП плотность модели 5L4V (Рис. 3), корректно описывая общий ход пептидного остова, всё же предоставляет мало информации о деталях положения атомов в пространстве. В каких-то небольших участках эта ЭП и вовсе прерывается и часто выглядит рвано.

Подводя итог, можно сказать, что у первой структуры (4X5P) разрешение должно быть очевидным образом ниже, чем у второй структуры (5L4V). Эти рассуждения подверждают данные с сайта PDB: структура 4X5P имеет разрешение 1.00 Å, а структура 5L4V - 2.99 Å.
Ссылка на сессию pymol: 2pr_1.pse.
Задание №2: ЭП и положение в структуре.
Далее предложено было работать со структурой 1V2K, представляющей из себя белок трипсин, связанный со специфическим ингибитором фактора Xa. Все связанные лиганды были удалены со структуры в PyMol для того, чтобы они не мешали визуализации ЭП самого полипептида.
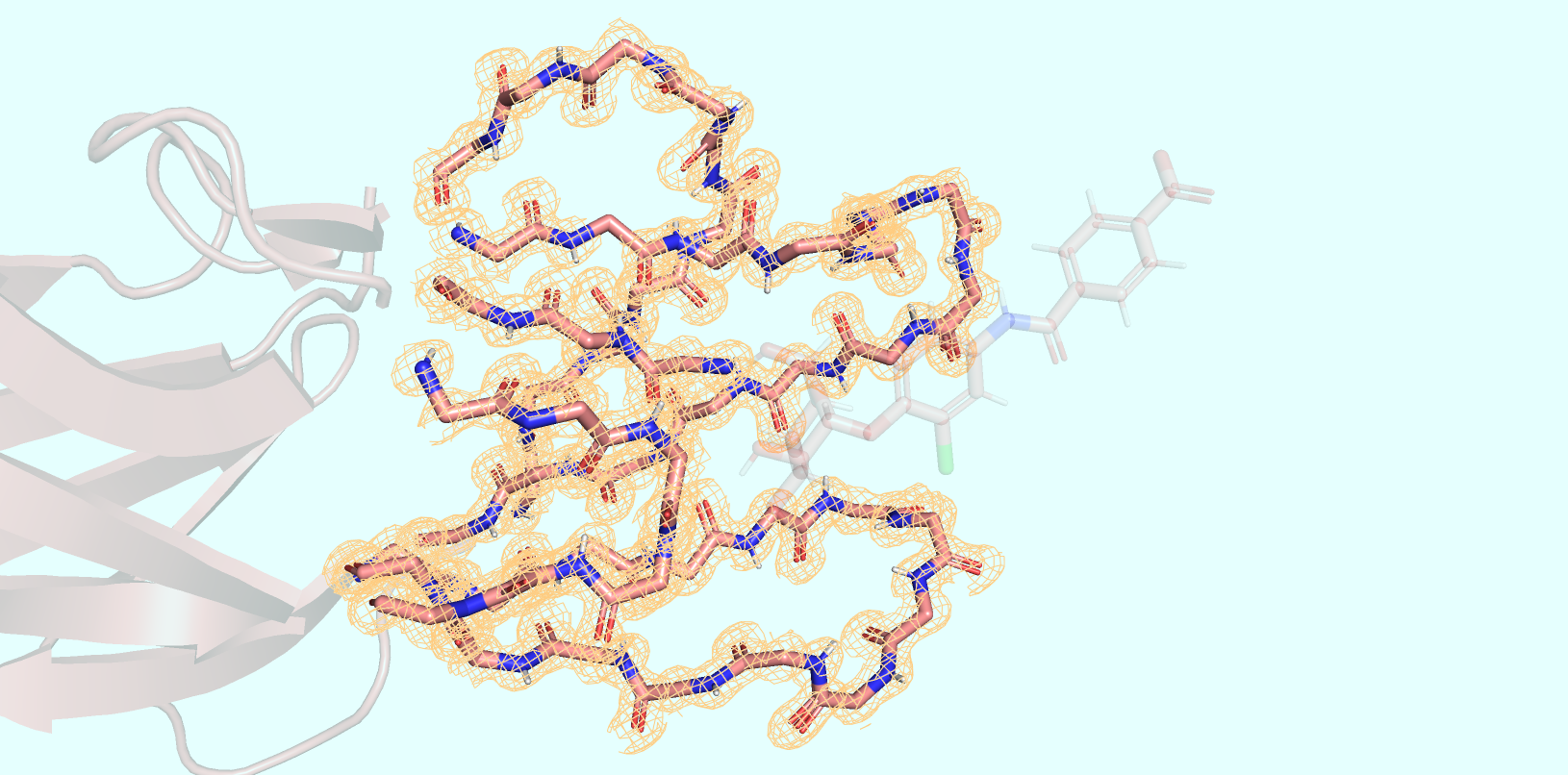
Электронная плотность визуализировалась в виде mesh для всего остова белка с разными уровнями подрезки: 1, 2 и 3 σ (Рис. 4). При этом в первом случае был выбран carve, равный 2 Å, а во всех последующих - 1 Å (параметры выбраны в целях минимизации визуализации лишней ЭП).
Совершенно предсказуемо, что с увеличением стандартного отклонения мы берём всё меньший процент облаков ЭП. Однако же хорошо заметно, что ЭП быстрее убывает в участках, соответствующих краям белковой молекулы (наиболее удалённых от ядра глобулы белка). Это связано с тем, что по краям располагаются наиболее слабо структурированные участки полипептида, которые являются наиболее подвижными и, как следствие, реконструированными с наименьшей уверенностью.
Ссылка на сессию PyMol: 2pr_2.pse.
Задание №3: ЭП и типы атомов.
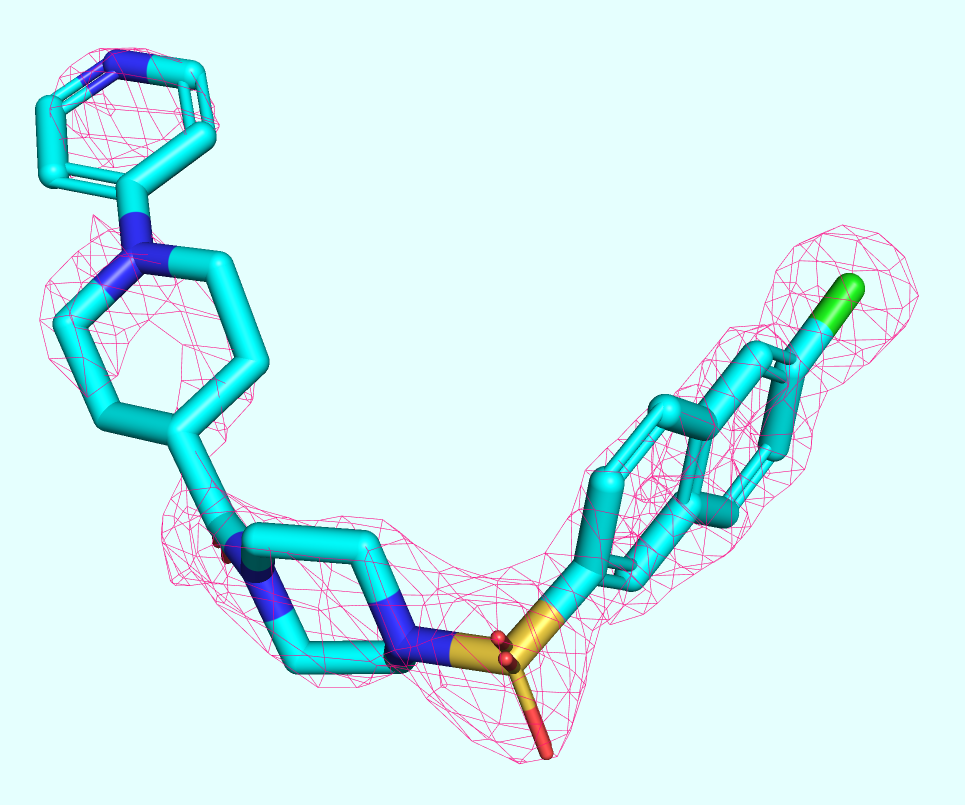
Далее рассматривалась ЭП лиганда в составе той же структуры, что и в задании №2. Аналогично использовались уровни подрезки, равные 1, 2 и 3 σ (Рис. 5). Во всех случаях был выбран carve, равный 2 Å.
Видно, что на уровне подрезки 1 все атомы лиганды покрыты электронной плотностью. Но с увеличением порога (Z-score) ЭП остаётся лишь в районе наиболее тяжёлых атомов: S, Cl, N (хоть и не на всех).
Это связано с физическими особенностями метода РСА: интенсивность рефлекса (отражённой рассеянной волны) равна квадрату модуля структурного фактора, который который можно представить в виде суммы атомных формфакторов, представляющих из себя некоторый интеграл от произведения, включающего в себя функцию ЭП, максимум которой совпадает с количеством электронов в атоме. Иными словами, атомы с наибольшем количеством электронов будут давать наиболее значимый сигнал. Это мы и видим при увеличении уровня подрезки.
Ссылка на сессию PyMol: 2pr_3.pse.
Кирилл Кузенков, студент четвёртого курса ФББ