term3 🕒
Нуклеотидные банки данных
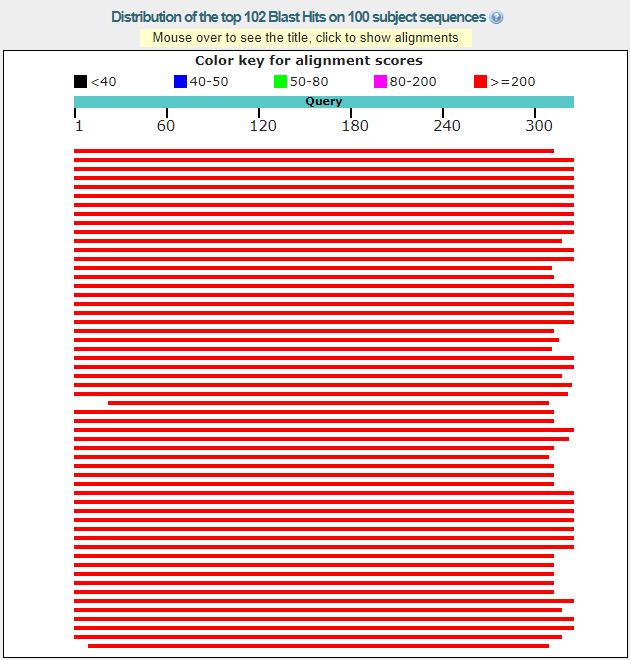
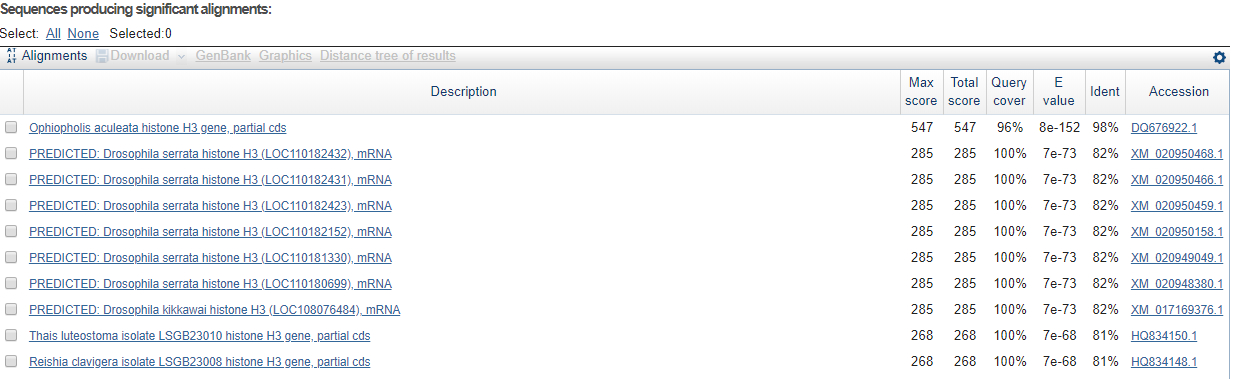
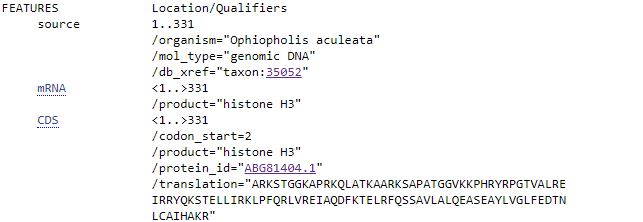
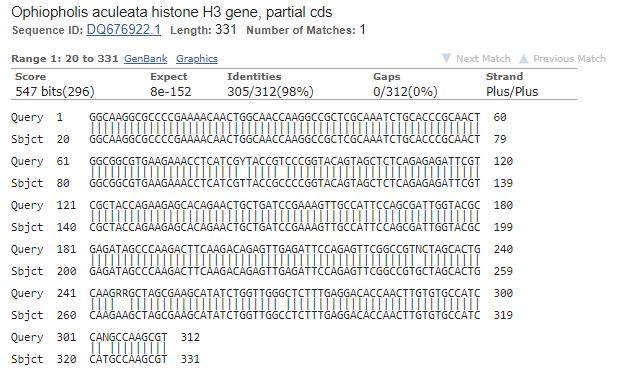
TASK1. Определение таксономии и функции прочтённой нуклеотидной последовательности (из практикума 6)
|
>EMBOSS_001 GGCAAGGCGCCCCGAAAACAACTGGCAACCAAGGCCGCTCGCAAATCTGCACCCGCAACT GGCGGCGTGAAGAAACCTCATCGYTACCGTCCCGGTACAGTAGCTCTCAGAGAGATTCGT CGCTACCAGAAGAGCACAGAACTGCTGATCCGAAAGTTGCCATTCCAGCGATTGGTACGC GAGATAGCCCAAGACTTCAAGACAGAGTTGAGATTCCAGAGTTCGGCCGTNCTAGCACTG CAAGRRGCTAGCGAAGCATATCTGGTTGGGCTCTTTGAGGACACCAACTTGTGTGCCatc cangccaagcgtgtcacnattatgc |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
TASK2. Сравнение списков находок нуклеотидных последовательностей тремя разными вариантами blast
| Параметры запуска BLAST | |||||||
|---|---|---|---|---|---|---|---|
| Algorithm | Database | Max Target Sequences | Expect Threshold | Word Size | Max matches | Match/Mismatch Scores | Gap Costs |
| megablast | Nucleotide collection (nr/nt) | 500 | 0.001 | 28 | 0 | 1,-2 | Linear |
| discontiguous megablast | Nucleotide collection (nr/nt) | 500 | 0.001 | 11 | 0 | 1,-2 | Existence:5, Extention: 2 |
| blastn | Nucleotide collection (nr/nt) | 500 | 0.001 | 11 | 0 | 2,-3 | Existence:5, Extention: 2 |
| Сравнение алгоритмов | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Алгоритм | Score лучшей находки | Score худшей находки | E-value лучшей находки | E-value худшей находки | Ident лучшей находки | Ident худшей находки | Query cover лучшей находки | Query cover худшей находки | |
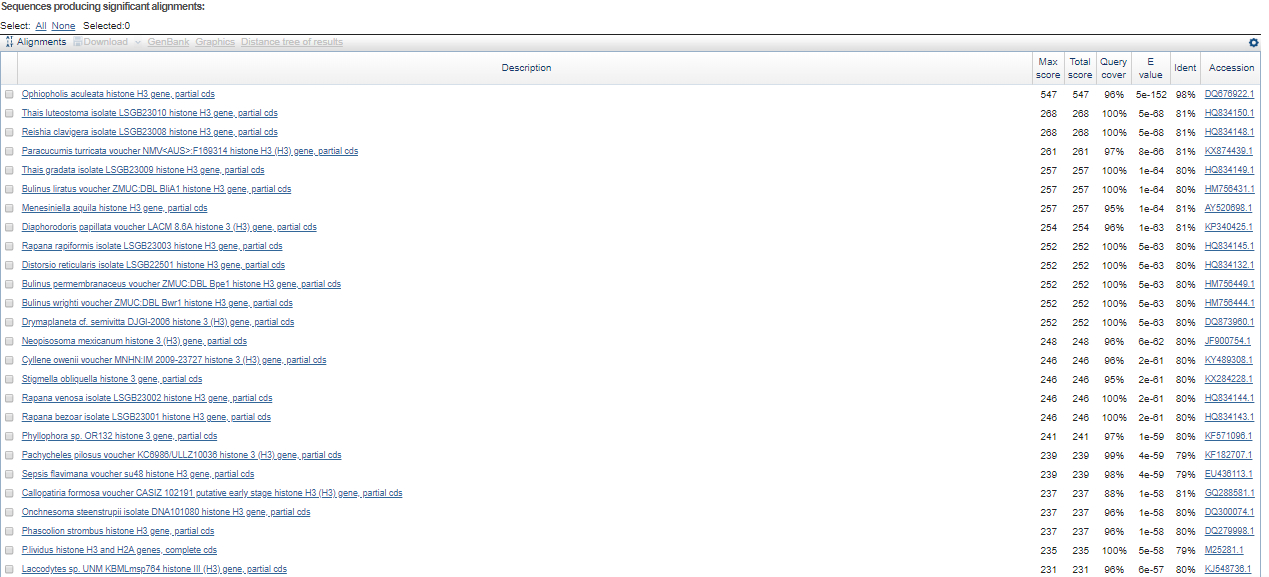
| megablast | 547 | 196 | 5е-152 | 2е-46 | 98 | 78 | 96 | 93 | |
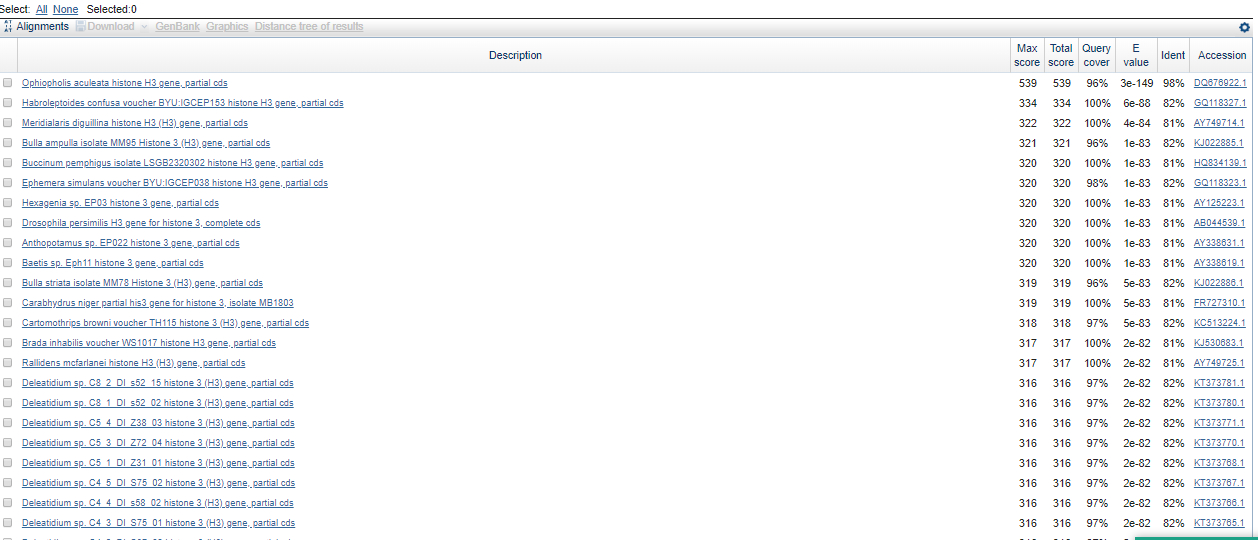
| discontiguous megablast | 539 | 306 | 3e-149 | 3e-79 | 98 | 80 | 96 | 99 | |
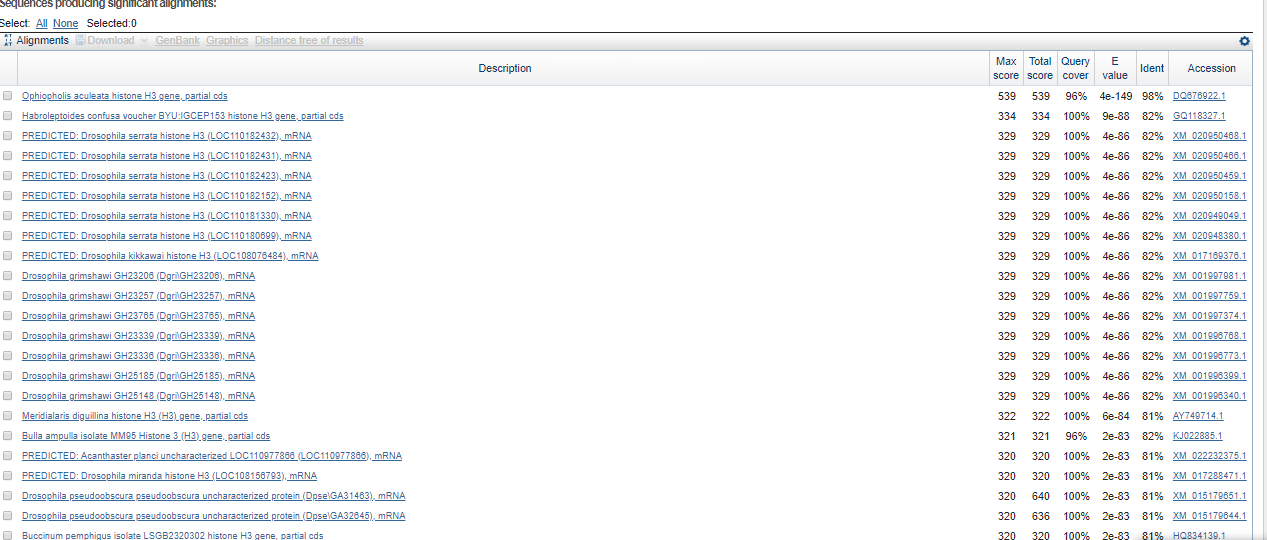
| blastn | 539 | 286 | 4e-149 | 4e-73 | 98 | 79 | 96 | 96 | |
Данные результаты не подтверждают идею того, что blastn рассчитан для поиска находок среди большей области поиска по сравнению с двумя первыми алгоритмами,это видно из значений Score худшей находки. Причиной несоответствия возможно являются параметры запуска BLAST
|
|
|
|
|
|
Алгоритмы нашли разные находки, причем megablast нашел находки от разных организмов, в то время как последние два предоставили набор из нескольких находок,принадлежащих одному виду (Deleatidium sp. - в случае discontiguous megablast; Drosophila grimshawi - в случае blastn)TASK3. Проверка наличия гомологов трех белков в неаннотированном геноме Amoeboaphelidium protococcarum
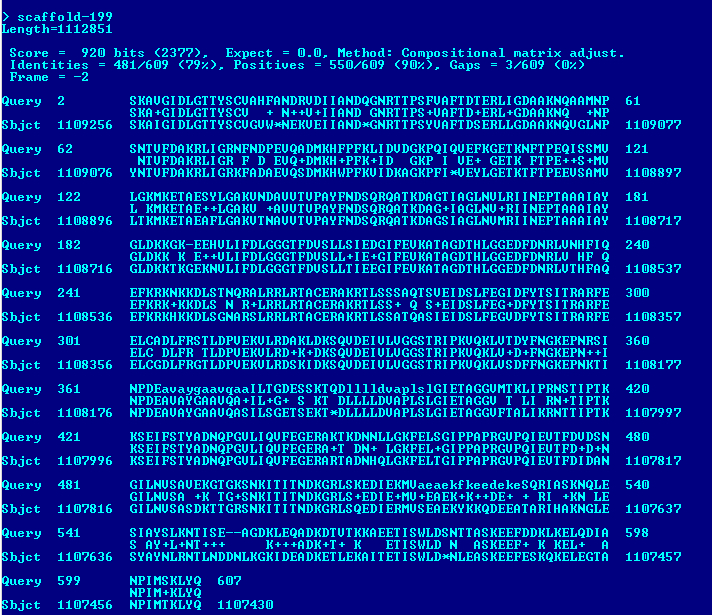
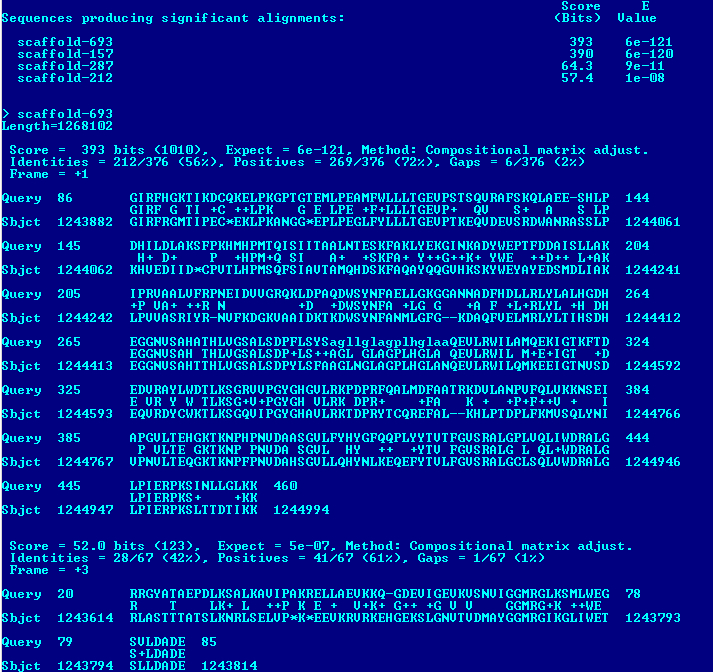
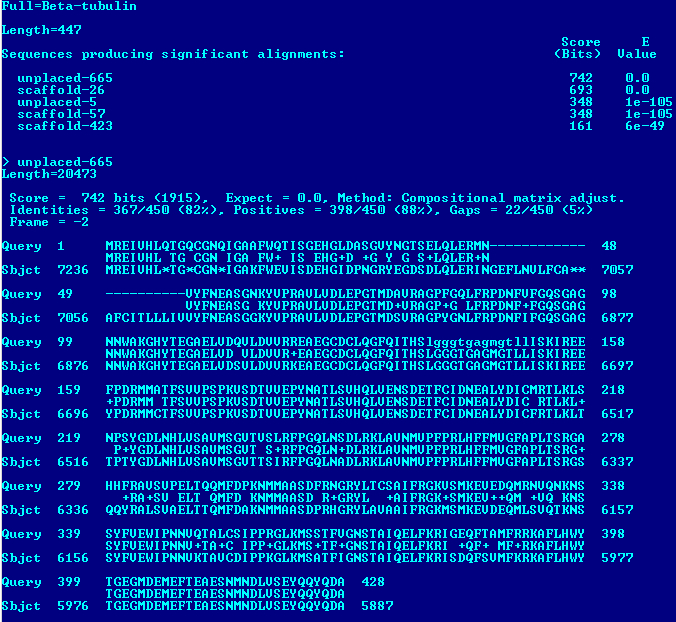
Были выбраны три белка: 1)HSP71_YEAST, шаперон HSP70, белок теплового шока; 2)PRPC_EMENI, митохондриальная цитратсинтаза; 3)TBB_NEUCR, тубулин, белок, участвующий в образовании микротрубочек. Ниже представлены результаты команды tblastn, выявляющая наличие или отсутствие гомологов белков в геноме Amoeboaphelidium protococcarum Несколько слов о выбранных мною белках: 1)шаперон HSP70, белок теплового шока, действуют как восстановители 3,4-чной структур других белков и облегчают их транспорт через мембраны внутри клетки.Белки теплового шока играют важную роль в белок-белковых взаимодействиях, например, при фолдинге и сборке сложных белков, препятствуют нежелательной агрегации белков. Эти белки обнаружены в клетках практически всех живых организмов, от бактерий до человека. 2)митохондриальная цитратсинтаза - это фермент, катализирующий реакцию конденсации ацетата (ацетил-CoA) и оксалоацетата, в результате чего образуется цитрат.Синтетаза цитрата обнаружена практически во всех клетках аэробных организмов, катализируемая реакция является лимитирующей на первом этапе Цикла трикарбоновых кислот. 3)Тубулин — белок, из которого построены микротрубочки.Долгое время полагали, что тубулин характерен только для эукариотических клеток. Однако недавние исследования обнаружили участвующий в делении прокариот гомологичный белок FtsZ, который может быть эволюционным предшественником тубулина.
|
|
|
|
|
|
|
|
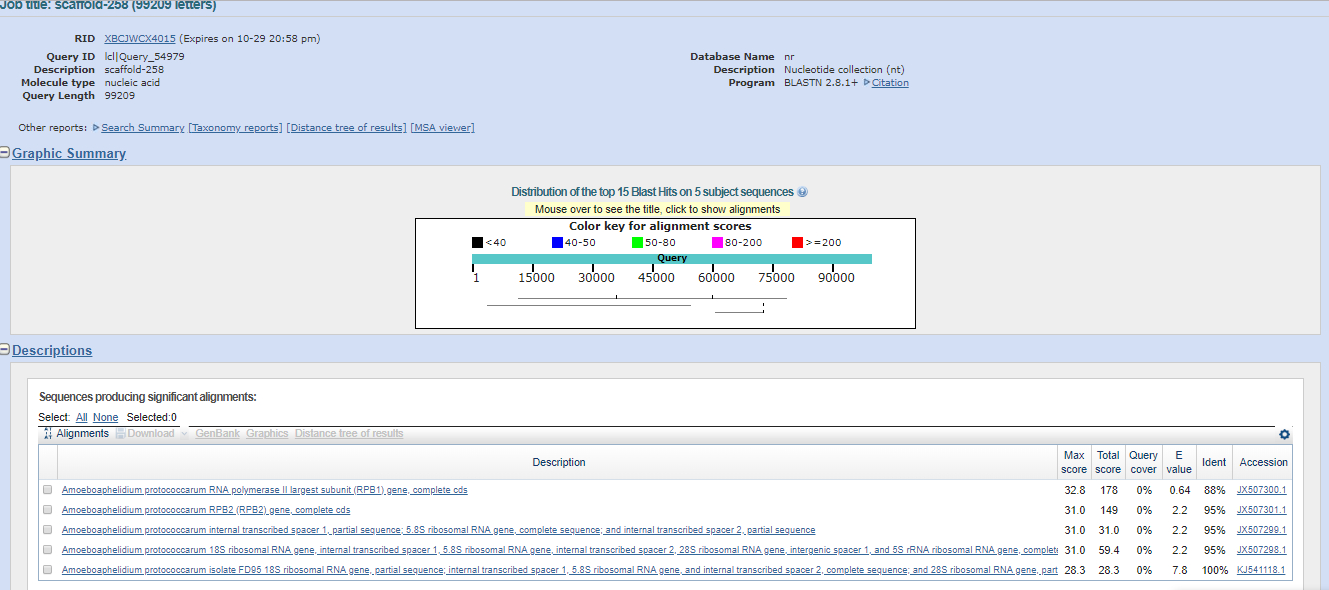
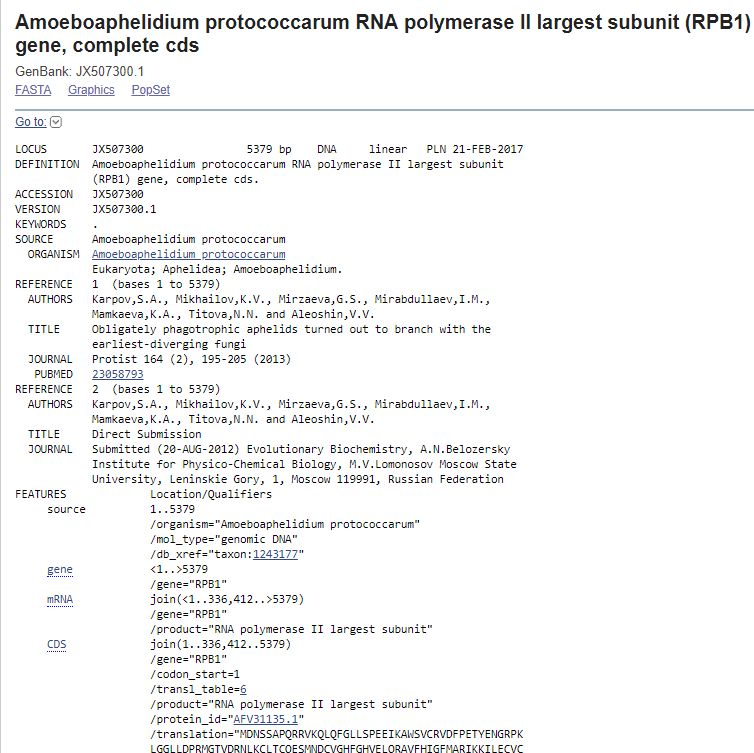
TASK4. Поиск гена белка в одном из контигов
С помощью команды infoseq пакета EMBOSS была получена информация о длинах контигов (infoseq X5.fasta -only -name -length.) Выбранная последовательность: scaffold-258 (длина 99209 нуклеотидов).С помощью команды (seqret X5.fasta:scaffold-258 -out *.fasta) был получен fasta-файл с последовательностью выбранным мною контига.Далее был запущен blastn с параметрами по умолчанию и ограничению по таксону Amoeboaphelidium (taxid:1243176). Результат работы blastn представлен ниже
|
|
|
|
|
|
TASK5. Карта локального сходства геномов двух бактерий
Для этого задания были выбраны бактерии рода Chlamydia: Chlamydia trachomatis(поражает мочеполовую систему) & Chlamydia psittaci(вызывает "попугайную" болезнь). Ниже представлена карта локального сходства геномов данных организмов.
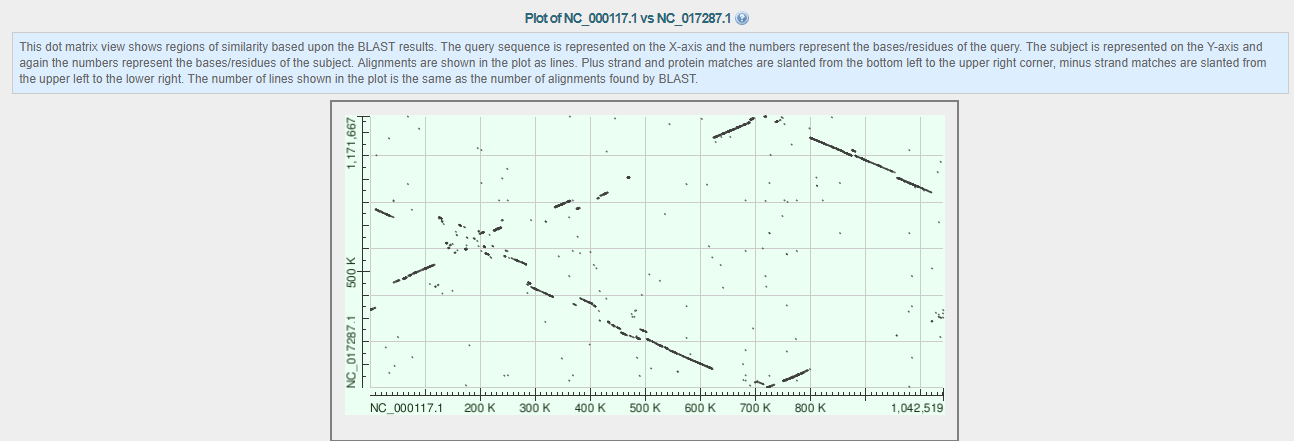
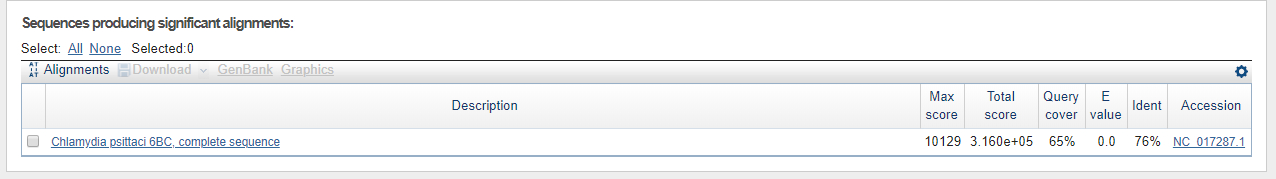
Color key is red & E-value=0. Это значит,что геномы содержат одинаковые участки последовательности, но только расположены они по-разному. Например, перые двести нуклеотидов в геноме Chlamydia psittaci соответствуют 330-350К таковым в геноме Chlamydia trachomatis. А начальный участок(0-240К) генома Chlamydia trachomatis совпадает 500-640К,700-800К участком генома Chlamydia psittaci.
© Цыганов Кирилл, 2017