Для работы взял бактерию Yersinia pestis. Поисковый запрос в Uniprot: keyword:"Purine biosynthesis [KW-0658]" organism:"yersinia pestis" AND reviewed:yes AND organism:"Yersinia pestis [632]"
Из 11 белков отобрал первые 10.
Таблица 1. Отобранные белки
| Идентификатор | Мнемоника | Название белка | Название гена |
|---|---|---|---|
| Q7CHW4 | PURT_YERPE | Formate-dependent phosphoribosylglycinamide formyltransferase | purT |
| Q8ZIV7 | PURA_YERPE | Adenylosuccinate synthetase | purA |
| Q0WD32 | IMDH_YERPE | Inosine-5'-monophosphate dehydrogenase | guaB |
| Q8ZCQ2 | PUR4_YERPE | Phosphoribosylformylglycinamidine synthase | purL |
| Q7CJY7 | FOLD_YERPE | Bifunctional protein FolD | folD |
| Q8ZAR2 | PUR2_YERPE | Phosphoribosylamine--glycine ligase | purD |
| Q8ZCU4 | GUAA_YERPE | GMP synthase | guaA |
| Q8ZCX8 | PUR5_YERPE | Phosphoribosylformylglycinamidine cyclo-ligase | purM |
| Q8ZAR3 | PUR9_YERPE | Bifunctional purine biosynthesis protein PurH | purH |
| Q7CIS2 | PURR_YERPE | HTH-type transcriptional repressor PurR | purR |
Таблица 2. Координаты upstream областей
| Координаты гена | Координаты upstream | Название гена |
|---|---|---|
| complement(2021083..2022264) | complement(2022265..2022364) | purT |
| 392314..392514 | 392214..392313 | purA |
| complement(3205218..3206681) | complement(3206682..3206781) | guaB |
| complement(3263310..3267200) | complement(3267201..3267300) | purL |
| 3151967..3152833 | 3151867..3151966 | folD |
| complement(1875886..1876788) | complement(1876789..1876888) | purD |
| complement(3203461..3205038) | complement(3205039..3205138) | guaA |
| 3154864..3155907 | 3154764..3154863 | purM |
| 4181118..4182707 | 4181018..4181117 | purH |
| 2681035..2682060 | 2680935..2681034 | purR |
Для генов на прямой цепи upstream область лежит до их первой координаты, для генов на обратной - после их последней координаты.
Из полного генома по полученным координатам были вырезаны последовательности. Пример команды: descseq -seq embl::AL590842.txt:392214:392313 -name "purA" -description "" -out purA.fasta
В случае, если ген лежит на обратной цепи: descseq -seq embl::AL590842.txt:2022265:2022364:r -name "purT" -description "" -out purT.fasta
Потом все 10 последовательностей были собраны в единый файл: cat *.fasta >> upstreams.fasta
Поиск мотива производился с помощью программы MEME, установленной на kodomo. Были выбраны следующие параметры:
Команда: ememe -dataset upstreams.fasta -outdir meme -nmotifs 3 -revcomp
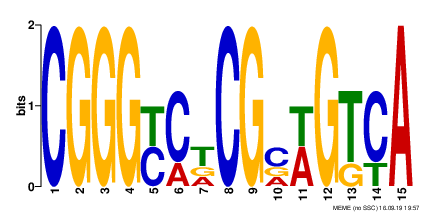

Первый мотив длиной 15 нуклеотидов встречается в 4 из 10 последовательностях. Несмотря на большое E-value = 8.8e+001 информационное содержание 22.6 бита. Думаю стоит поискать похожий мотив у других родственных организмов.
Второй мотив длиной 13 нуклеотидов встречается в 5 из 10 последовательностях. Имеет еще большее E-value = 9.1e+002 и меньшее информационное содержание 18.3 бита. Не считаю этот мотив достоверным.
Третий мотив длиной 19 нуклеотидов встречается в 5 из 10 последовательностях. Имеет E-value = 2.1e+002 и информационное содержание 23.8 бит. Его бы я тоже проверил, поискав в родственных организмах.