Анализ генома бактерии Vescimonas coprocola
Автор: Кирцова Ксения Михайловна1
1Факультет Биоинженерии и биоинформатики Московского государственного университета имени М. В. Ломоносова, Москва, Россия
Контакты: kskirtsova@gmail.com
Аннотация: В данном мини-обзоре приведено качественное и количественное описание генома и протеома бактерии Vescimonas coprocola с использованием функционала электронных таблиц и программирования на языке Python.
1 Введение
Vescimonas coprocola - грамотрицательная облигатно-анаэробная неспорообразующая неподвижная палочка, которая была выделена из фекалий взрослого японца с раком желудка. Точечные дискообразные бело-серые полупрозрачные колонии на анаэробной среде Gifu; оптимальная температура роста колоний 37℃ [1].
1.1 Таксономическая принадлежность [2]
- класс Clostridia;
- семейство Oscillospiraceae;
- род Vescimonas;
- вид Vescimonas coprocola.
1.2 Фотография штаммов Vescimonas coprocola

Рис. 1. Клетки в среде Medium 83 после 4-дневной инкубации при 37℃ в анаэробных условиях: М. Китахара; Научно-технический иновационный кластер RIKEN, Япония; MM52, 2020 [3].
2 Материалы и методы
Информация о геноме и протеоме бактерии была взята из базы данных NCBI Genome [4]. Обработка данных проводилась с помощью электронных таблиц Google sheets и языка программирования Python 3.9. При обработке были использованы только строки таблицы особенностей генома, у которых в колонке “features” стоит “CDS” (повторяющиеся и не подходящие под условия значения были удалены с помощью фильтра).
В пункте 3.1 с помощью функций в Google sheets на основе таблицы с данными о геноме было рассчитано распределение длин белков и была построена гистограмма числа аминокислотных остатков в белках бактерии.
В пункте 3.2 была написана программа на языке Python 3.9, которая с помощью обработки файла с геномом бактерии определила все старт-кодоны в кодирующих последовательностях (CDS) и вычислила, сколько раз встречается каждый старт-кодон.
3 Результаты
3.1 Распределение длин белков
Геном Vescimonas Coprocola кодирует 2375 белков. Из них 2371 синтезируются с хромосом, а 4 - с плазмиды. Белки имеют длину от 37 до 2859 аминокислот. Самый длинный белок - YDG domain-containing protein, самый короткий - 50S ribosomal protein L36. Среднее значение длин белков - 307.47 аминокислоты. Исходя из гистограммы, в геноме преобладают белки длиной 100-300 аминокислот. Эта длина белков является нормальной для бактерий.
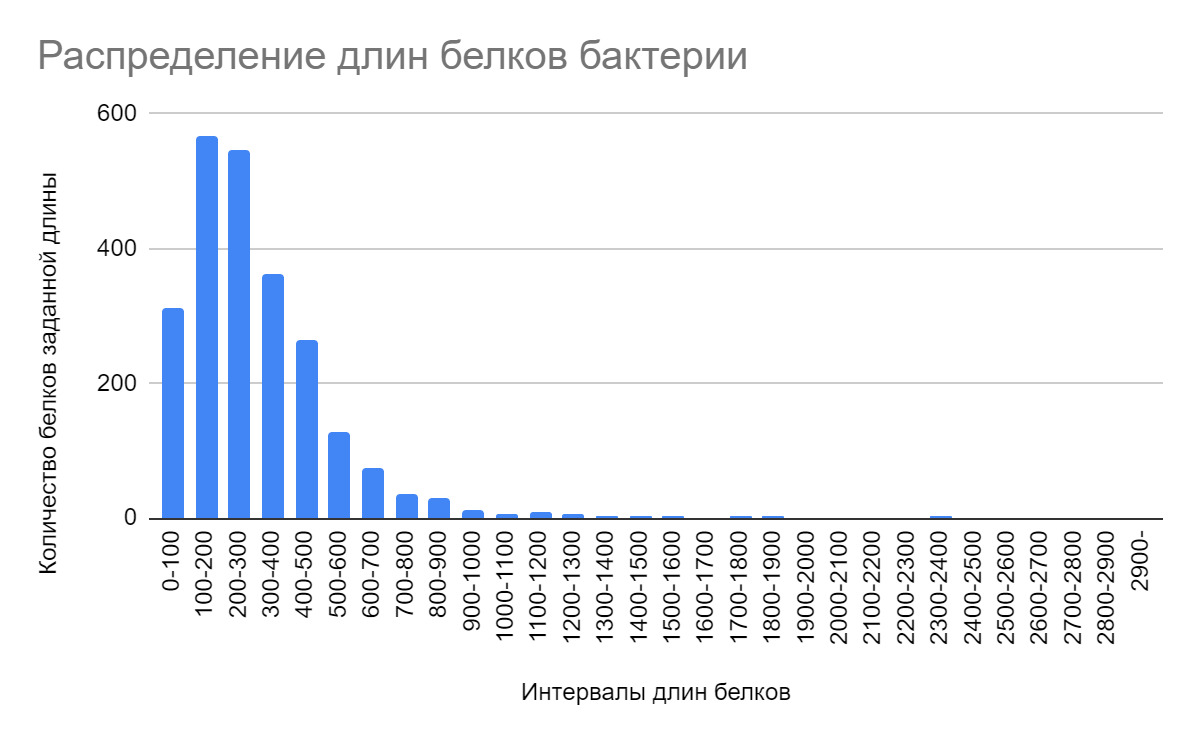
Рис. 2. Гистограмма длин белков
3.2 Анализ стартовых кодонов
Старт-кодон или инициаторный кодон - первый кодон матричной РНК, c которого начинается трансляция белка в рибосоме. У бактерий старт-кодон всегда кодирует модифицированный метионин (N-формилметионин). Старт-кодону предшествует 5′-нетранслируемая область (5'-UTR). В 5'-UTR бактерий локализована последовательность Шайна-Дальгарно (AGGAGG), которая служит для связывания рибосомы и отделена спейсером от старт-кодона [5].
Подавляющее большинство последовательностей Vescimonas coprocola начинается со старт-кодона ATG в смысловой цепи ДНК (AUG в мРНК), что неудивительно, так как этот триплет является наиболее распространенным стартовым кодоном. Также встречается существенное число альтернативных старт-кодонов GTG (GUG) и TTG (UUG). Альтернативные старт-кодоны обычно кодируют метионин, когда они находятся в начале белка (даже если, находясь в остальных местах последовательности, они кодируют другую аминокислоту). Так в случае, если кодоны GTG и TTG находятся внутри кодирующей последовательности, они кодируют аминокислоты валин и лейцин соответственно. Если же какой-либо из этих кодонов расположен в начале последовательности, он кодирует стартовый метионин. Это происходит потому, что для инициации трансляции используется специальная транспортная РНК. Антикодоном инициирующей аминоацил-тРНК всегда является CAU, он полностью комплементарен основному старт-кодону AUG и частично комплементарен более редким кодонам [6].
Кроме частично комплементарных GTG и TTG в исключительных случаях в клетках бактерий инициация может начинаться с других триплетов. Также при секвенировании могла возникнуть погрешность. Поэтому в качестве старт-кодонов у бактерии Vescimonas coprocola встречаются триплеты ДНК ATC, CTG, ATA, ATT, CAG, GAC, AAG, ACC, CAT, CGT, CTT, GCT, GGT, TCT, TTA. Эти так называемые «слабые» кодоны могут выполнять свою функцию в комбинации с сильными последовательностями Шайна-Дальгарно или другими структурными элементами, способствующими инициации [6].
| ATG | 2117 |
| GTG | 161 |
| TTG | 102 |
| ATC | 16 |
| CTG | 9 |
| ATA | 5 |
| ATT | 3 |
| CAG | 2 |
| GAC | 2 |
| AAG | 1 |
| ACC | 1 |
| CAT | 1 |
| CGT | 1 |
| CTT | 1 |
| GCT | 1 |
| GGT | 1 |
| TCT | 1 |
| TTA | 1 |
Табл. 1. Распределение количеств старт-кодонов
4 Заключение
- В геноме бактерии Vescimonas coprocola наиболее распространены белки, состоящие из 100-300 аминокислот. Однако есть и более короткие (0-100 аминокислот), и более длинные (2800-2900 аминокислот).
- В геноме присутствуют разные триплеты, выполняющие роль стартовых кодонов, самый часто встречающийся из них - ATG (AUG). Реже встречаются старт-кодоны GTG (GUG) и TTG (UUG).
5 Сопроводительные материалы
- Таблица особенностей генома Vescimonas coprocola: https://docs.google.com/spreadsheets/d/1v3_DryMp9izwHxYC5LSXTMF1PVr-RaS0XTf2ZIitnP0/edit?usp=sharing
- Программа для анализа старт-кодонов на языке Python 3.9: https://drive.google.com/file/d/1vp6Vt1fh9vutOtJerTkxK8vLtiyu5rWk/view?usp=sharing
6 Литература
- Общее описание бактерии: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9879126/
- Таксономия: https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2714355
- Фотография клеток Vescimonas coprocola: https://www.jcm.riken.jp/cgi-bin/jcm/jcm_number?JCM=34014
- Полный геном Vescimonas coprocola: https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/018/408/575/GCF_018408575.1_ASM1840857v1
- Информация о старт-кодонах: Пибоди Д. С. Инициация трансляции у триплетов без AUG в клетках млекопитающих (англ.) // The Journal of Biological Chemistry: журнал. — 1989. — Том 264, № 9. — С. 5031-5035
- Информация о старт-кодонах: Лобанов А. В.; Туранов А. А.; Хэтфилд Д. Л.; Гладышев В. Н. Двойные функции кодонов в генетическом коде (англ.) // Критические обзоры по биохимии и молекулярной биологии (англ.) русск.: журнал. — 2010. — Том 45, № 4. — С. 257-265