Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Я выбрала белок Major outer membrane protein TtoA (рус. основной белок внешней мембраны TtoA). PDB - 3dzm, Uniprot - Q72JD8_THET2. Он был выделен из грамотрицательной бактерии Thermus thermophilus, локализован во внешней мембране.
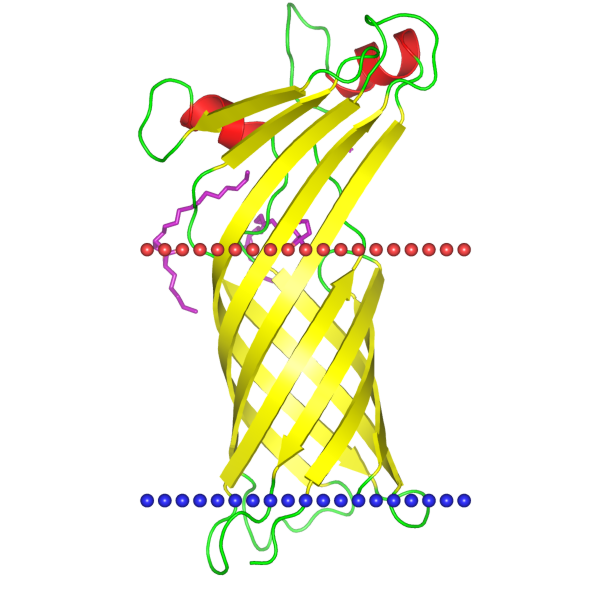
Рис. 1. Структура белка 3dzm из базы данных OPM
Белок состоит из 1 субъединицы. Координаты трансмембранных участков белка: 1(4-12),2(16-25),3(33-42),4(74-83),5(93-104),6(128-139),7(144-155),8(195-206)
Я нашла последовательность белка на сайте UniProt и запустила для нее DeepTMHMM.
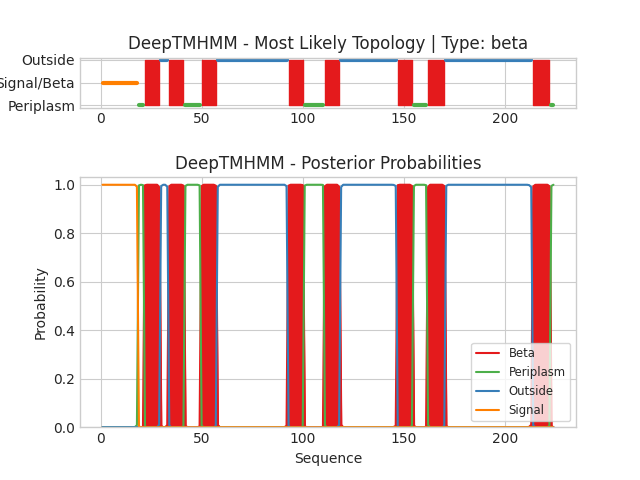
Рис. 2. Графическое изображение результатов DeepTMHMM. На верхнем графике изображена наиболее вероятная топология белка. На нижнем графике показаны вероятности топологий. Здесь можно увидеть вероятности формирования фрагментом белка бета-листа, нахождения его в периплазме или снаружи клетки или его участия в передаче сигнала
Количество бета-листов, предсказанное DeepTMHMM, совпадает с количеством, указанным на сайте OPM. Однако координаты не совпадают.
- Координаты бета-листов:
- 22 29
- 34 41
- 50 57
- 93 100
- 111 118
- 147 154
- 162 170
- 214 222
- Похожие координаты бета-листов:
- OPM: 3(33-42), Deep TMHMM: 2(34-41)
- OPM: 5(93-104), Deep TMHMM: 4(93-100)
- OPM: 7(144-155), Deep TMHMM: 6(147-154)
Разница в координатах β-листов может быть связана с тем, что в UniProt последовательность длиннее (224 аминокислоты), а в PDB (OPM) — короче (208 аминокислот в структуре). Я думаю, что первые 16 аминокислот — это сигнальный пептид, который отрезается при созревании белка. DeepTMHMM анализировал полную последовательность (включая сигнальный пептид), а OPM работал уже с укороченной версией белка. Координаты как раз отличаются на 17-19 АК.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок Proton-gated ion channel, conformation 2 (рус. протон-управляемый ионный канал, конформация 2). PDB - 4lmj, Uniprot - GLIC_GLOVI. Он был выделен из цианобактерии Gloeobacter violaceus, локализован во внутренней мембране.
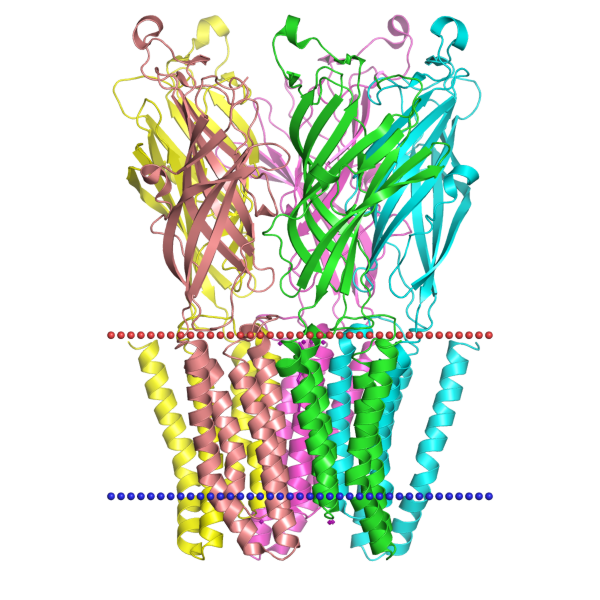
Рис. 3. Структура белка 4lmj из базы данных OPM
Белок состоит из 5 субъединиц. Координаты трансмембранных участков белка (на каждой субъединице): 1( 194- 216), 2( 221- 242), 3( 253- 274), 4( 293- 314).
Я нашла последовательность белка на сайте UniProt и запустила для нее DeepTMHMM.
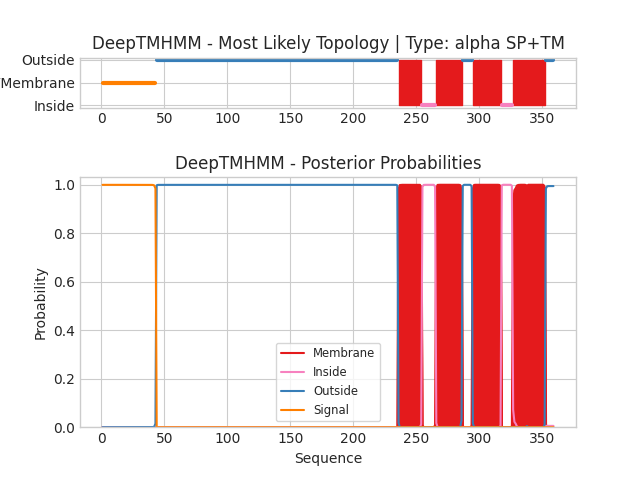
Рис. 4. Графическое изображение результатов DeepTMHMM
Количество альфа-спиралей, предсказанное DeepTMHMM, совпадает с количеством, указанным на сайте OPM. Однако координаты не совпадают.
- Координаты альфа-спиралей:
- 236 254
- 266 286
- 295 317
- 327 352
- Похожие координаты альфа-спиралей:
- OPM: 4(293-314), Deep TMHMM: 3(295-317)
Причина расхождений аналогична той, которая была в 1 пункте: В UniProt последовательность длиннее (350 аминокислот), а в PDB (OPM) — короче (310 аминокислот). Это может означать, что у белка есть дополнительные участки (например, сигнальный пептид или неструктурированные фрагменты), которые отсутствуют в кристаллической структуре. DeepTMHMM анализировал полную последовательность, а OPM работал только со структурной частью. Если из предсказанных DeepTMHMM координат вычесть разницу (40 аминокислот), предсказанные координаты α-спиралей оказываются гораздо ближе к данным OPM.