Мини-обзор генома и протеома бактерии Capnocytophaga gingivalis
Резюме
Данный мини-обзор представляет работу с геномом и протеомом флавобактерии Capnocytophaga gingivalis с использованием программирования на языке Python и функционала электронных таблиц.
ВВЕДЕНИЕ
Capnocytophaga gingivalis представляют собой класс флавобактерий[1], населяющих полость рта и являющихся одним из главных компонентов зубного налета[2]. При микроскопии выявлено, что они представляют собой бактерии палочковидной формы с веретенообразной морфологией, имеющие гранулированную наружную поверхность.
Бактерия встречается в стандартном микробиоме человека. Можно сделать вывод, что для C. gingivalis и полости рта человека характерен такой тип отношений, как комменсализм[3], ведь при попадании бактерии в другую среду она становится патогенной[4]. Исследования показывают, что C. gingivalis может вызывать заболевания всего организма. У пациентов с пневмонией было выявлено высокое содержание бактерии в легких[5]. Также бактерию обнаружили и в крови[6] – бактериемия, что помогает связать ее с лейкемией. Долгое время бактерию считали устойчивой к антибиотикам, однако было выявлено, что типичная клетка C. gingivalis проявляет чувствительность к бета-лактаму. Благодаря этому стало возможно лечение онкологических больных с бактериемией.
Таким образом, изучение данного может позволить найти и другие виды антибиотиков, действующие на C. gingivalis, что поможет в лечение бактериемии у сложных пациентов.
МЕТОДЫ И МАТЕРИАЛЫ
Программа «Анализ нуклеотидного состава генома» определяет нуклеотиды, содержащиеся в геноме бактерии, их общее количество, количество каждого отдельного нуклеотида и их десятичное содержание в геноме.
Программа «Анализ частоты стоп-кодонов в кодирующих последовательностях белков» рассчитывает встречаемость всех стоп-кодонов в CDS геноме бактерии. Помимо расчета количества стоп-кодонов программа создает файл out.txt, в котором находятся все последовательности нестандартных стоп-кодонов. На основе данных этого файла была создана таблица «Последовательности с другими стоп-кодонами».
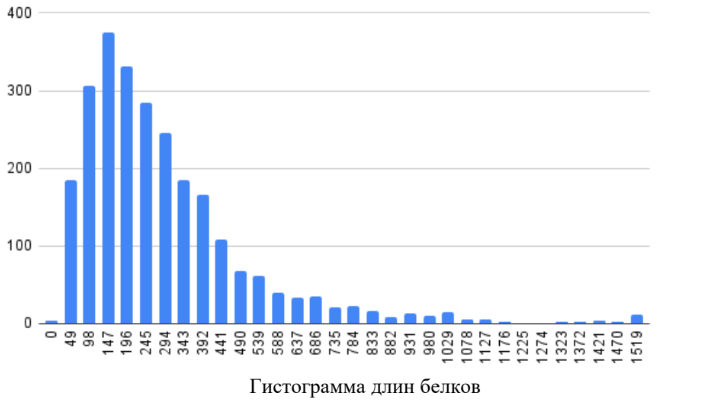
С помощью сервиса Google Sheets и функции СЧЕТЕСЛИМН удалось построить гистограмму длин белка «Гистограмма длин белков» и определить некоторые данные о протеоме бактерии.
С помощью команды «Распределение генов белков на прямой и обратной цепях ДНК» для Linux можно найти распределение белков, псевдогенов и генов РНК на прямой и обратной цепям ДНК. При помощи сервиса Google Sheets находится общее распределение последовательностей по цепям.
РЕЗУЛЬТАТЫ
«Анализ нуклеотидного состава генома». С помощью программы «Анализ нуклеотидного состава генома» удалось выяснить, что геном бактерии Capnocytophaga gingivalis состоит из A, T, G, C нуклеотидов. Общее их содержание равно 2838633 Из полученных данных видно, что содержание парных нуклеотидов является примерно одинаковым, о чем и говорит второе правило Чаргаффа.
| Нуклеотид | Количество | Содержание |
|---|---|---|
| A | 856890 | 0.3018671311155757 |
| T | 843796 | 0.297254347427089 |
| G | 568576 | 0.2002992285371163 |
| C | 569371 | 0.200579292920219 |
«Анализ частоты стоп-кодонов в кодирующих последовательностях белков». С помощью программы «Анализ частоты стоп-кодонов в кодирующих последовательностях белков» удалось выяснить количественное содержание стоп-кодов в геноме Capnocytophaga gingivalis: "TGA": 371, "TAA": 1755, "TAG": 517, количество других стоп-кодонов: 15. В таблице «Последовательности с другими стоп-кодонами» можно найти описания последовательностей, заканчивающихся на нестандартные стоп-кодоны. Все они оказались пвсевдогенами.
| Стоп-кодон | Встречаемость в геноме |
|---|---|
| TAA | 1755 |
| TAG | 517 |
| TGA | 371 |
| Другие стоп-кодоны | 15 |
«Анализ гистограмм длин белков». С помощью сервиса Google Sheets удалось выяснить количественное содержание белков в протеоме Capnocytophaga gingivalis. Наибольшей длиной является 4466. Наибольшее количество белков протеома исследуемой бактерии имеет длину от 147 до 195 аминокислот, далее при увеличении количества аминокислот количество белков постепенно снижается. Минимальная длина белка протеома составляет 38 аминокислот.
«Распределение генов белков на прямой и обратной цепях ДНК». Распределение генов приведено в Таблице 3. Вероятность получить случайно такое распределение генов белков по цепям одной ДНК равна 0,2065138482, то есть вероятность очень мала, что является статистическим значим различием.
| Тип гена | Прямая цепь | Обратная цепь |
|---|---|---|
| Gene | 1392 | 1747 |
| ncRNA | 2 | - |
| tRNA | 6 | 51 |
| tmRNA | 1 | 1 |
| rRNA | 6 | 3 |
| CDS | 1365 | 1747 |
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Таблица особенностей бактерии
Геном бактерии
CDS последовательность
Программа «Анализ нуклеотидного состава генома»
Программа «Анализ частоты стоп-кодонов в кодирующих последовательностях белков»
Таблица «Последовательности с другими стоп-кодонами»
Таблица «Гистограмма длин белков»
Команда «Распределение генов белков на прямой и обратной цепях ДНК»
Таблица «Распределение генов белков по цепям ДНК»
Cсылка на сопроводительные материалы
СПИСОК ИСПОЛЬЗОВАННОЙ ЛИТЕРАТУРЫ
Кагермайер, А., Лондон, Дж. (1986). Идентификация и предварительная характеристика лектиноподобного белка Capnocytophaga gingivalis. Инфекция и иммунитет, 51(2):490-494.
Спратт Д., Гринман Дж., Шаффер А. (1996). Capnocytophaga gingivalis: влияние концентрации глюкозы на рост и продукцию гидролитических ферментов. Микробиология, 142:2161-2164.
Спратт Д., Гринман Дж., Шаффер А. (1999). Рост и продукция гидролитических ферментов Capnocytophaga gingivalis на различных белковых субстратах. Оральная микробиология и иммунология, 14:122-126.
Спратт Д., Гринман Дж., Шаффер А. (1995). Аминопептидаза Capnocytophaga gingivalis: потенциальный фактор вирулентности. Микробиология, 141:3087-3093.
Гейслер В., Мальхотра У., Штамн В. (2001). Пневмония и сепсис из-за резистентной к фторхинолонам Capnocytophaga gingivalis после трансплантации аутологичных стволовых клеток. Трансплантация костного мозга, 28:1171-1173.
Мантадакис Э., Данилатоу В., Кристиду А., Стиакаки Э., Калманти М. (2003). Бактериемия Capnocytophaga gingivalis выявляется только при количественных посевах крови у ребенка, больного лейкемией. Журнал детских инфекционных заболеваний, 22(2):202-204.