Практикумы 1-4
Отобранные бактерии
| Escherichia coli | ECOLI |
| Yersinia pestis | YERPE |
| Pasteurella multocida | PASMU |
| Pseudomonas mendocina | PSEMY |
| Rhizobium meliloti | RHIME |
| Bartonella henselae | BARHE |
| Roseobacter denitrificans | ROSDO |
| Paracoccus denitrificans | PARDP |
Скобочная формула дерева
((((ECOLI,YERPE),PASMU),PSEMY),((RHIME,BARHE),(ROSDO,PARDP)));
Изображение дерева

Ветви дерева
1) (RHIME;BARHE) vs (ROSDO;PARDP;ECOLI;YERPE;PASMU;PSEMY)
2) (ROSDO;PARDP) vs (RHIME;BARHE;ECOLI;YERPE;PASMU;PSEMY)
3) (RHIME;BARHE;ROSDO;PARDP) vs (ECOLI;YERPE;PASMU;PSEMY)
4) (RHIME;BARHE;ROSDO;PARDP;PSEMY) vs (ECOLI;YERPE;PASMU)
5) (RHIME;BARHE;ROSDO;PARDP;PSEMY;PASMU) vs (ECOLI;YERPE)
Нетривиальные ветви:
Enterobacterales(ECOLI; YERPE) vs (RHIME; BARHE; ROCDO; PARDP; PSEMY; PASMY)
Hyphomicrobiales(RHIME; BARHE) vs (ECOLI; YERPE; ROCDO; PARDP; PSEMY; PASMY)
Rhodobacterales(ROCDO; PARDP) vs (ECOLI; YERPE; RHIME; BARHE; PSEMY; PASMY)
Gammaproteobacteria(RHIME; BARHE; ROCDO; PARDP) vs Alphaproteobacteria(ECOLI; YERPE; PSEMY; PASMY)
Практикум 2
Был взят белок RS4. Белок входит в состав 30S (малой субъединицы) РНК прокариот. Этот комплекс участвует в связывании транспортной РНК с информационной РНК (мРНК).
MAFFT → FastME
FastMe - метод минимальной эволюции - матричный метод (дистанционный), создает матрицу длин. В нем выбирается дерево с наименьшей длиной, то есть сумма длин всех ветвей минимальна. Почему минимальной эволюции - потому что чем меньше длина ветвей, тем меньше мутаций произошло и тем ближе организмы друг к другу эволюционно. лучший вариант - организмы прям очень близко, значит 'эволюции' (мутаций) было мало.

Исходное дерево

Укорененное дерево
Скобочная формула
((((PASMU:0.059328,(YERPE:0.038023,ECOLI:0.00378):0.053713):0.041226,PSEMY:0.277731):1.202335,ROSDO:0.102848):0.001704,(BARHE:0.120561,RHIME:0.116222):0.084352,PARDP:0.091153)
MAFFT → TNT
TNT - метод максимальной экономии - символьный метод. Сравниваются отдельные позиции и выбираются деревья с наименьшим количеством различий. Последовательность эволюционных шагов для получения этих деревьев минимальна. Для уменьшения времени работы, алгоритм рассматривает не все сайты, а только те, которые встретились в двух и больше последовательностях, поэтому может выдавать плохие варианты.

Исходное дерево
Скобочная формула дерева
(ECOLI,(YERPE,(PASMU,(PSEMY,(RHIME,BARHE,(ROSDO,PARDP))))))
MAFFT → PhyML
PhyML - наибольшего правдоподобия - тоже символьный. Строит все возможные деревья и выбирает "наиболее правдоподобные". Почему он так хорош - для каждого сайта рассматриваются все возможные мутации и в итоге перебираются вот прям вообще все возможные деревья. Из этих деревьев выбирается вариант с наименьшим числом произошедших мутаций.

Исходное дерево
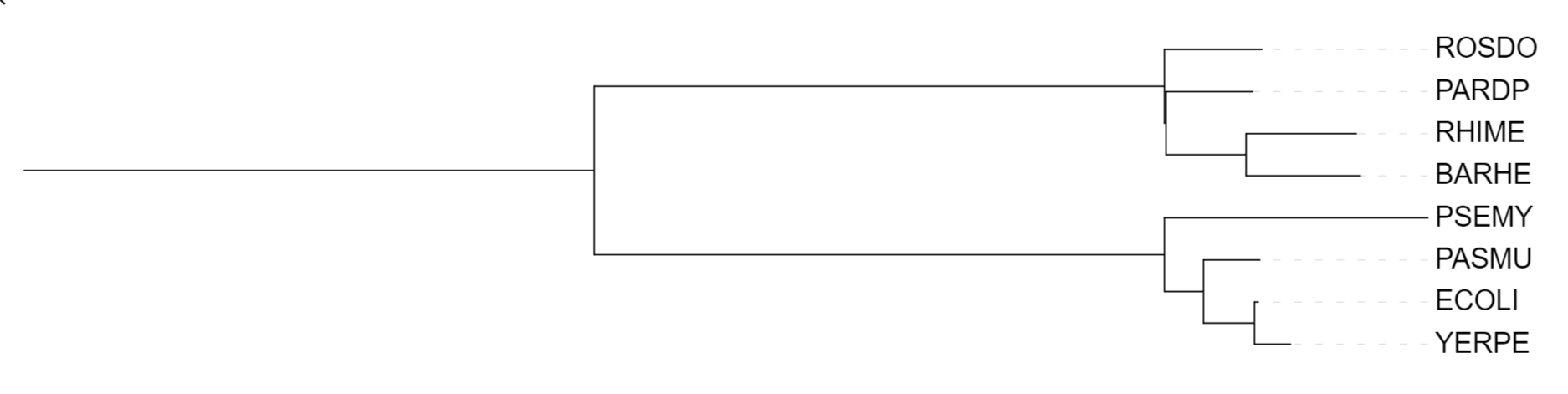
Укорененное дерево
Скобочная формула
(YERPE:0.02920801, ECOLI:0.01726098, (PASMU:0.05427734, (PSEMY:0.2232599, (ROSDO:0.02848571, (PARDP:0.09242506, (RHIME:0.10106647, BARHE:0.14828855) :0.09764209):0.0738853):1.15851319):0.09060238):0.06156671
Практикум 3
Укоренение с использованием BACSU
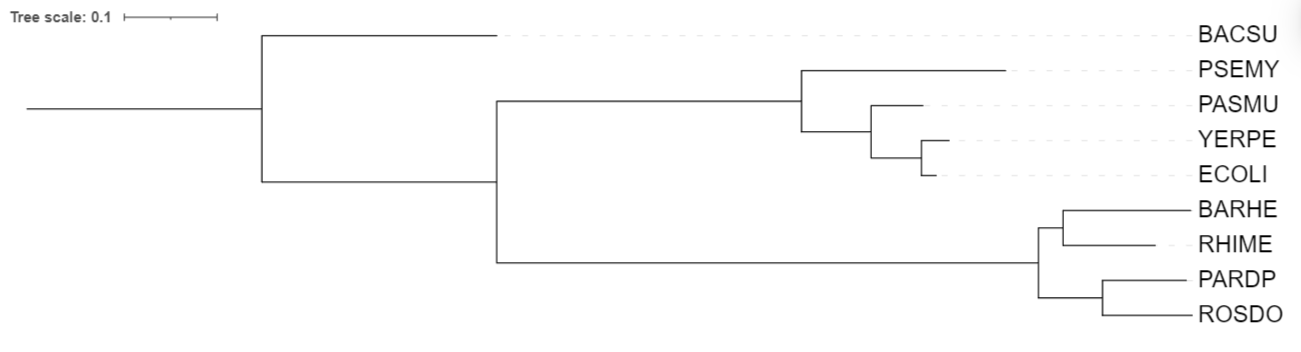
Дерево, полученное при укоренении с BACSU

Исходное дерево
Бутстреп
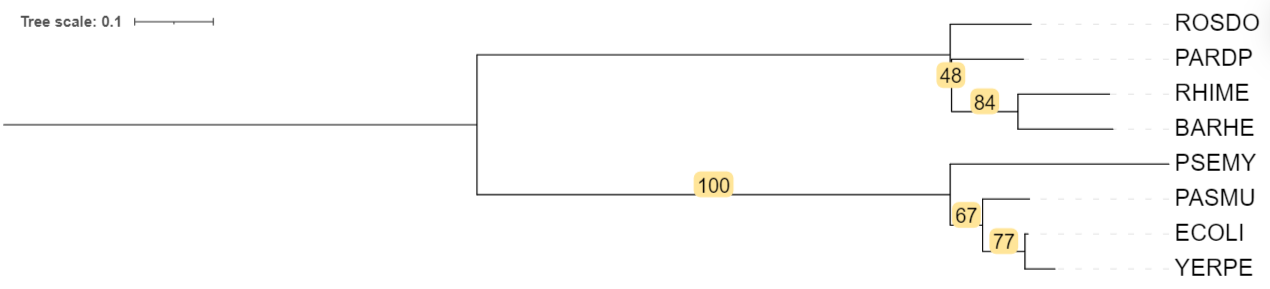
Совпадающие с эталоном ветви поддержаны хорошо.
Построение дерева по нуклеотидным последовательностям

Реконструкция филогении была выполнена с помощью FastME, так как метод PhyML работал бы очень долго, да и такая высокая точность нам не нужна.
Причина, по которой деревья могут не совпадать, кроется в дупликации генов, потери генов и горизонтальный перенос. Если в предковом виде происходит дупликация гена, изменения в каждой из копий происходят независимо от другой, и обе копии наследуются потомками.
Практикум 4
Составление списка гомологичных белков, включающих паралоги

Реконструкция и визуализация

Паралоги:
CLPX_ROSDO HLSU_ROSDO
A1BBJ2_PARDP A1AZV8_PARDP
RUVB_BARHE A0A0H3LXZ4_BARHE
Ортологи:
CLPX_RHIME CLPX_BARHE
RUVB_PARDP RUVB_ROSDO
HSLU_PSEMY HSLU_PARDP
Скобочная формула дерева
По ссылке можно найти скобочную формулу данного дерева.
Ортологи
Ортологи - это гомологичные белки из разных организмов, разошедшиеся в процессе видообразования и выполняющие одну и ту же функцию (чаще всего).
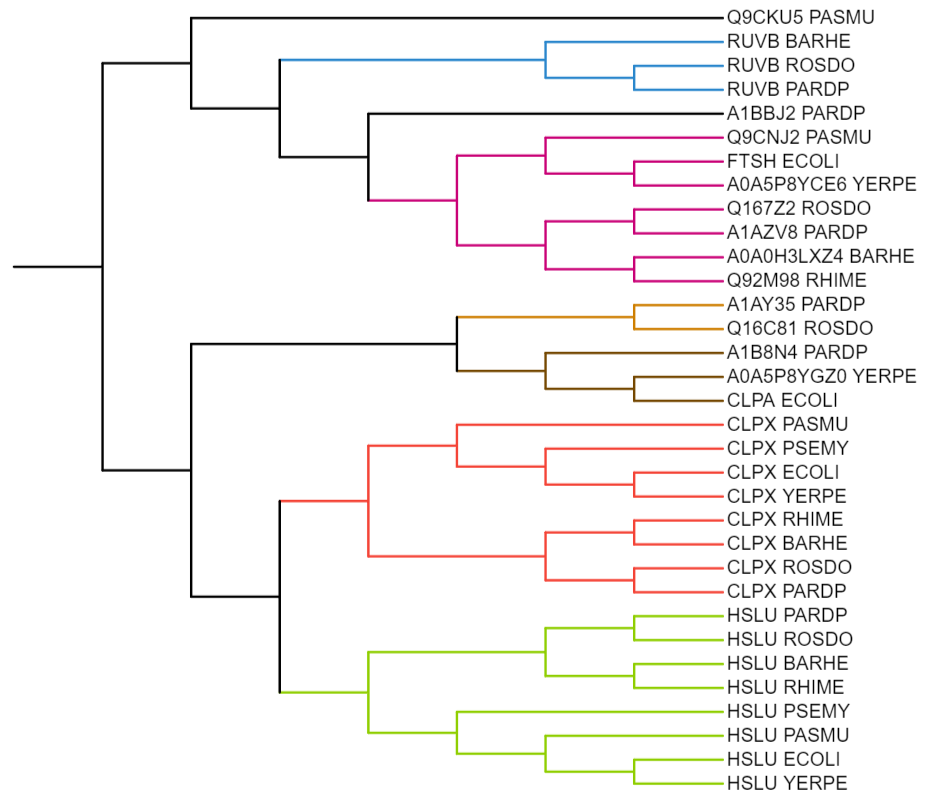
Разным цветом выделены разные группы ортологов.

Зеленым выделена ветка RUVB: RUVB_PARDP, RUVB_ROSDO, RUVB_BARHE
Бордовым выделена ветка ORT_1: Q9CNJ2_PASMU, A0A5P8YCE6_YERPE, FTSH_ECOLI, Q92M98_RHIME, A0A0H3LXZ4_BARHE, A1AZV8_PARDP, Q167Z2_ROSD
Желтым выделена ветка ORT_2: Q16C81_ROSDO, A1AY35_PARDP
Оранжевым выделена ветка ORT_3: CLPA_ECOLI, A0A5P8YGZ0_YERPE, A1B8N4_PARDP
Бирюзовым выделена ветка CLPX: CLPX_PARDP, CLPX_ROSDO, CLPX_BARHE, CLPX_RHIME, CLPX_PASMU, CLPX_PSEMY, CLPX_YERPE, CLPX_ECOLI
Фиолетовым выделена ветка HSLU: HSLU_YERPE, HSLU_ECOLI, HSLU_PASMU, HSLU_PSEMY, HSLU_RHIME, HSLU_BARHE, HSLU_ROSDO, HSLU_PARDP
Паралоги
Паралоги -это гомологичные белки, принадлежащие одному организму. Они возникают в результате дупликации гена и могут разойтись в процессе эволюции настолько, что начнут выполнять разные функции.
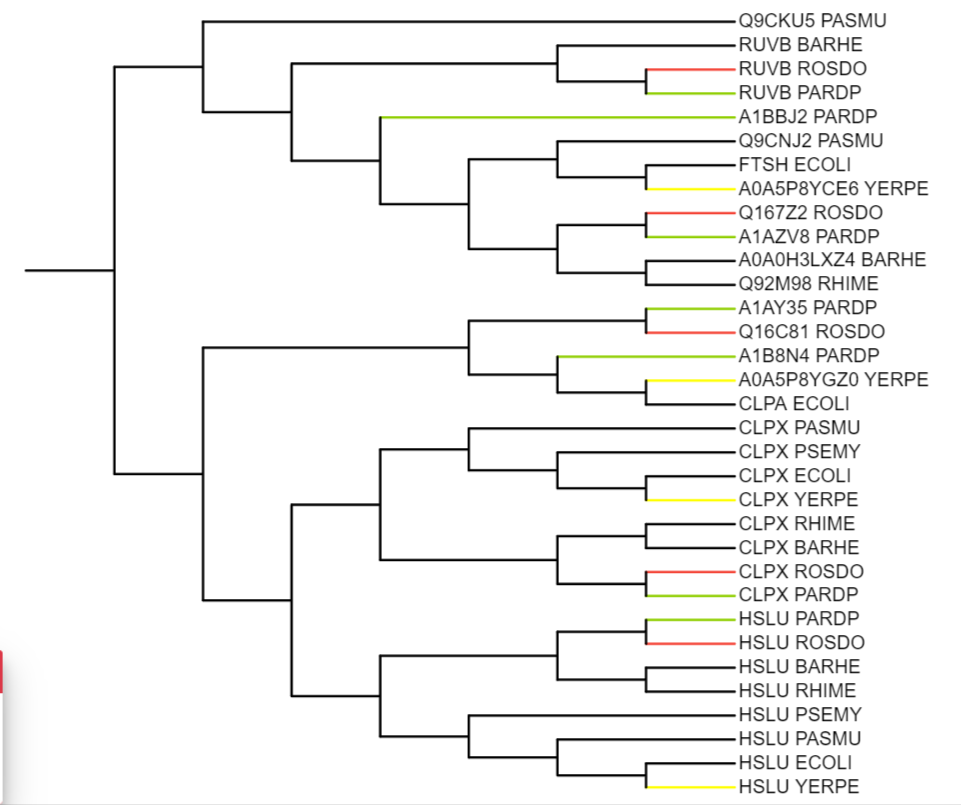
Разным цветом выделены паралоги.
Зеленым выделены белки-паралоги организма PARDP
Красным выделены белки-паралоги организма ROSDO
Желтым выделены белки-паралоги организма YERPE
Выводы:
Найденные белки присутсвуют во всех организмах.
Все реконструкции соответствуют эталонным, хотя в некоторых деревьях есть неразрешенные ветви.