Предсказание генов эукариот.
1. Описание скэффолда

Рис. 1. E. guineensis1 |
В первом задании я буду описывать скэффолд NW_011550920. Он принадлежит Пальме масличной. Систематика:
Домен: Eukaryota
Царство: Viridiplantae
Тип: Embryophyta
Отдел: Magnoliophyta
Класс: Liliopsida
Семейство: Arecaceae
Род: Elaeis
Вид: E. guineensis
E. guineensis — растение семейства Пальмовые (Arecaceae), вид рода Масличная пальма (Elaeis).
Родиной этого растения считаются прибрежные районы экваториальной Западной Африки от 16° с. ш. до 15° ю.ш.
Культивируется, кроме Африки, и в других странах с тропическим климатом (Малайзия, Индонезия и др.)
для получения ценного пищевого и технического масла.В дикорастущем виде масличная пальма представляет собой дерево высотой до 20—30 м,
в культуре она редко бывает выше 10—15 метров. Ствол появляется лишь на четвёртый—шестой год жизни,
а под пологом леса иногда только через 15—20 лет[3]. Диаметр ствола у взрослого дерева достигает 25 см.
Корневая система мощная, но залегает обычно неглубоко. У взрослых растений от основания ствола отходят
многочисленные придаточные корни, у некоторых экземпляров они густо покрывают ствол до высоты 1 м.
Листья крупные, перистые, длиной до 6—7 м. В кроне взрослого растения их 20—40 штук, причём до 20—25 листьев ежегодно отмирает,
заменяясь новыми. На черешках листьев имеются крупные буроватые шипы.
Соцветия крупные (длиной до 70 см), расположены в пазухах листьев. Обычно мужские и женские соцветия образуются на одном растении,
хотя изредка встречаются и двудомные растения. Иногда в одном соцветии можно обнаружить цветки обоих полов, чаще это бывает у молодых пальм[3].
Мужское соцветие содержит до 150 тысяч цветков и производит огромное количество пыльцы: до 1 млрд пыльцевых зёрен с одного соцветия.
Ось женского соцветия значительно толще, чем у мужского. На нём развивается до 600—800 плодов, общая масса плодовой кисти 10—30 кг,
иногда и больше. В год на одном растении образуется 3—6, иногда до 10 таких кистей.
Плод масличной пальмы — простая костянка величиной со сливу (3—5 см длиной), массой в среднем 6-8 граммов.
Окраска скорлупы обычно оранжевая, хотя встречаются растения с фиолетовыми и чёрными плодами.
Всего было два проекта по секвенированию генома этого организма,
результаты были предоставлены EMBL (сборка GCA_000442705.1)1,2.
Немного информации о скэффолде
NW_011550920:
- длина: 2683772 п.н.;
- число генов и CDS: 40;
- число некодирующих РНК (ncRNA): 1;
- файл с последовательностью: 1.fasta
Для этого соответствующая запись была найдена в БД Nucleotide и
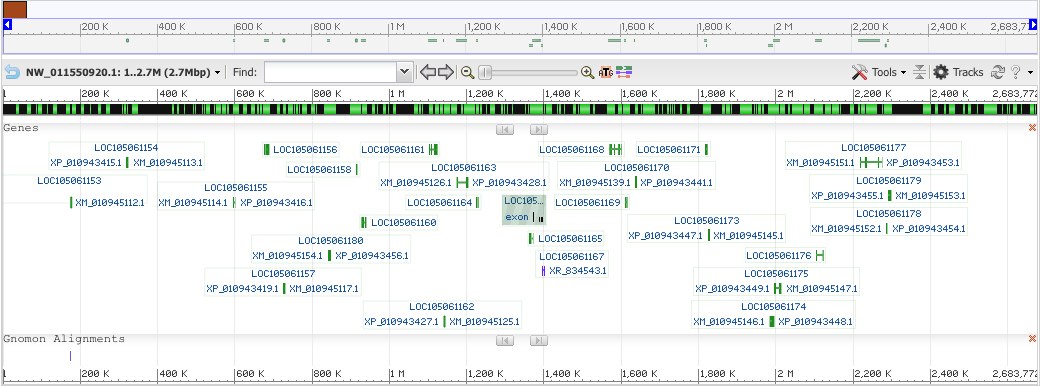
открыта в геномном браузере. Далее вручную был найден ген, для которого по данным базы данных
предсказывается альтернативный сплайсинг (рис. 2). В качестве продуктов трансляции мРНК
предсказаны 2 изоформы фумарилацетоацетат-гидролазы (ФАА гидролазы).
Ссылки на записи для вариантов мРНК и соответствующих изоформ на сайте NCBI:
- мРНК:
XP_010943415.1, изоформа X1: XM_010945113.1
- мРНК:
XM_010945154.1, изоформа X2: XP_010943456.1
На рис. 3 показан домен упомянутой изоформы (1).
 Рис. 3. Изоформа X1 Рис. 3. Изоформа X1 |
Таким образом, в состав изоформы Х1 помимо ФАА-гидролазы и N-концевой ФАА-гидролазы входит
еще и BAG-домен.
ФАА гидролаза— фермент, гидролизующий фумарилацетоацетат с образованием фумарата
и ацетоацетата (в ходе катаболизма тирозина). Мутации, вызывающие дефект в ферменте, приводят
к накоплению катаболитов тирозина. У людей, например, мутации в ФАА-гидролазе
вызывают тирозинемию, приводящую к повреждению почек, печени, периферических нервов и т.д.
N-концевая ФАА-гидролаза — домен, имеющий неохарактеризованную функцию. Имеет
конформацию бочонка, как у SH3-домена.
BAG-домен — домен-регулятор активности шаперона Hsp70, предотвращающего
мисфолдинг белков и и их дальнейшую агрегацию, стимулируя рефолдинг денатурированных
белков. Bag1, к примеру, образует с Hsp70 комплекс и ингибирует рефолдинг
белков.
2. Предсказание генов и белок-кодирующих областей в выданном скэффолде
Для выполнения задания использовался web-сервер AUGUSTUS (режим Prediction). Входные параметры:
AUGUSTUS parameter project identifier: arabidopsis
Genome file: 1.fasta
User set UTR prediction: true
Report genes on: both strands
Alternative transcripts: few
Allowed gene structure: predict any number of (possibly partial) genes
Ignore conflictes with other strand: false
Для параметров модели был выбран организм Arabidopsis thaliana (Резуховидка Таля),
т.к. согласно современной системе классификации цветковых растений APG III порядок
Brassicales, которому принадлежит этот вид, ближе всех из предложенных
в AUGUSTUS к порядку Elaeidinae, к которому принадлежит наша пальма.
Также в параметрах я установила предсказание UTR, т.к. судя по GenBank они есть.
Еще было отмечено наличие нескольких альтернативных транскриптов, которые тоже есть в
записи (мы рассматривали один из примеров в п.1.). Все остальные параметры совпадают с
параметрами по умолчанию. В таблице 1 приведено описание полученных файлов.
Табл. 1. Описание полученных файлов
| Файл |
Описание содержимого |
| predictions.tar.gz |
Архив со всеми файлами, выданными программой |
| augustus.gff |
Предсказания генов в формате .gff |
| augustus.gtf |
Предсказания генов в формате .gtf |
| augustus.aa |
Последовательности трансляций предсказанных генов в формате .fasta |
| augustus.cdsexons |
Предсказанные экзоны в формате .fasta |
| augustus.codingseq |
Предсказанные CDS в формате .fasta |
| augustus.gbrowse |
Координаты найденных генов, мРНК и т.д. для геномного браузера |
| augustus.mrna |
Предсказанные мРНК (с UTR) в формате .fasta |
Так как сравнивать целиком гены в двух файлах проблематично, я сравнивала
экзоны (вернее, все найденные CDS), полученные из обоих файлов .gff.
Для того, чтобы сверить найденные CDS, был написан скрипт
pr12.py.
Затем с помощью команды (входные файлы: 1.gff3,
augustus.gff):
python pr12.py 1.gff3 augustus.gff results.out
был получен файл results.out, содержащий координаты CDS,
одинаковых в обоих файлах, а также общую статистику.
Прим. Некоторые координаты в выходном файле повторяются дважды. Это связано с
предсказанием двух транскриптов, а, следовательно, CDS для них повторяются дважды. В
записи GenBank такое тоже встречается. В качестве примера может служить обсуждаемый
в п.1 ген фумарилацетоацетазы.
Таким образом, из 263 CDS, найденных AUGUSTUS, только 149 (84,65% от CDS в записи)
совпадают с аннотированными в GenBank. Не найдено было 27 (15,34%) CDS, так что в
принципе я бы сказала, что процент предсказанных так же, как в записи GenBank
кодирующих последовательностей довольно высок. Однако помимо 149 верно найденных CDS,
web-сервер нашел 114 (!), не аннотированных в GenBank. Так что несмотря на
хороший процент верно найденных CDS, число неверно найденных слишком велико, чтобы
говорить о высокой точности алгоритма. Возможно, выбор более близкого организма для
параметров модели улучшил бы ситуацию.
Что касается обсуждаемого в п. 1 гена, AUGUSTUS тоже, как ни странно, предсказал 2
транскрипта (см. g.45). Однако С-конец был предсказан по-другому: последний экзон имеет
координаты 268231..268106, в то время как в аннотации есть еще экзон 267452..267488 для
транскрипта Х1 и + 266146..266855 для транскрипта Х2 (здесь говоря об экзонах имеются в виду
и соответствующие участки CDS). Таким образом, частично веб-сервер смог предсказать
обсуждаемый ген и даже сделал ту же ошибку, предсказав два трансрипта.
© Mishchenko Polina 2016