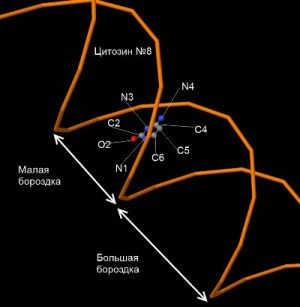
Рис. 1. Показана ориентация атомов в сторону малой или большой бороздки в цитозине №8. Малая и большая бороздки подписаны. Сахарафосфатный остов ДНК представлен в виде линий.
С помощью программы fiber из пакета 3DNA получены pdb файлы, содержащие A-, B- и Z-формы дуплекса ДНК, сосоящие из повторяющейся 5 раз последовательности нуклеотидов "gatc":
А-форма - gatc-a.pdb, B-форма - gatc-a.pdb, Z-форма - gatc-z.pdb
В спиралях ДНК можно легко отличить внешнюю бороздку от внутренней по внешнему виду. Атомы нуклеотидов могут быть направлены как в сторону малой, так и в сторону большой бороздки. Рассмотрим это на примере нуклеотида цитозина (№8 в структуре gatc-b.pdb) (рис. 1)

Рис. 1. Показана ориентация атомов в сторону малой или большой бороздки в цитозине №8. Малая и большая бороздки подписаны. Сахарафосфатный остов ДНК представлен в виде линий.
В сторону большой бороздки обращены атомы C4, N4, C5, C6.
В сторону малой бороздки обращены атомы N3, C2, O2, N1 (атом N1 соединяется с остовом) (рис. 2).
Для а- формы спирали наблюдается аналогичная картина. А для z- формы спирали атомы направлены по другому (рис. 3)
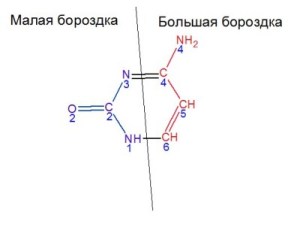
Рис. 2. На рисунке представлена схема нуклеотида цитозина в спиралях типа a и b. Синим выделены атомы, ориенторованные в сторону малой бороздки. Красным - в сторону большой.Исходный файл, полученный с помощью программы Chemsketch.
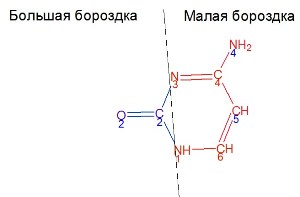
Рис. 3. На рисунке представлена схема нуклеотида цитозина в z-спирали. Синим выделены атомы, ориенторованные в сторону большой бороздки. Красным - в сторону малой.Исходный файл, полученный с помощью программы Chemsketch.
Анализ сгенерированный структур приведен в таблице 1.
| Таблица 1 | |||
| Параметр | A-форма | B-форма | Z-форма |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (A) | 28,0 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 (6 на половину витка) |
| Ширина большой бороздки (A) | 16,8 (13 - 30) | 17,2 (12 - 26) | 18,3 (4 - 14) |
| Ширина малой бороздки (A) | 8,0 (5 - 30) | ~11,7 (12 - 33) | ~11, 6(4 - 19) |
Торсионные углы для цитозина из структуры с a-спиралью показаны на рисунке 4, а для структуры с b-спиралью - на рисунке 5. Сравнение торсионных углов приведено в таблице 2.
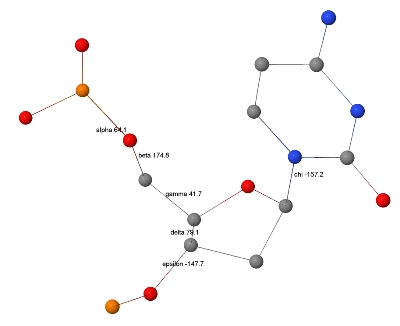
Рис. 4.Скрипт в программе Jmol, с помощью которого можно получить данное изображение.
| Таблица №2 | |||||||
| угол | α | β | γ | δ | ε | ζ | χ |
| A-форма из презентации | 62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| A-форма gatc-a.pdb | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма из презентации | 63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| B-форма gatc-b.pdb | 85.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Торсионные углы в нуклеотидах днк структуры с pdb кодом 1TRO были определены с помощью программ find_pair и analyze. Результат представлен в таблице №3. Также данные были проанализированы, найдены средние углы для каждого нуклеотида, а также среднеквадритичное отклонение углов от следних по структуре для каждого нуклеотида. Как видно из таблицы, наибольшее среднеквадратичное отклонение у Т №17. Его торсионные углы больше всех отличаются от среднего значения в этой структуре.
| Таблица №3 | |||||||||
| № | Base | alpha | beta | gamma | delta | epsilon | zeta | chi | |
| Среднее | C | 3,6 | 85,1 | 24,6 | 79,8 | -156,1 | -85,6 | -158,3 | |
| G | -71,9 | -2,8 | 64,4 | 123,6 | -28,8 | -94,0 | -126,3 | ||
| T | -64,3 | 15,0 | 53,6 | 130,6 | -131,5 | -95,2 | -116,6 | ||
| A | -65,9 | 95,1 | 41,7 | 128,0 | -29,1 | -110,9 | -110,7 | ||
Определение структуры водородных связей
Для определения структуры водородных связей нуклеотидов рнк структуры с pdb кодом 1f7u были использованы программы find_pair и analyze.
| Таблица №4 | ||||||
| № пары | Тип стебля | Цепь 1 | Цепь 2 | Особые места в спирали | ||
| № остатка | Название нуклеотида | № остатка | Название нуклеотида | |||
| 1 | Акцепторный стебель | 901 | PSU | A | 972 | |
| 2 | 902 | U | A | 971 | ||
| 3 | 903 | C | G | 970 | ||
| 4 | 904 | C | G | 969 | ||
| 5 | 905 | U | G | 968 | ||
| 6 | 906 | C | G | 967 | ||
| 7 | 907 | G | C | 966 | ||
| 8 | Т-стебель | 949 | 5MC | G | 965 | |
| 9 | 950 | C | G | 964 | ||
| 10 | 951 | A | U | 963 | ||
| 11 | 952 | G | C | 962 | ||
| 12 | 953 | G | C | 961 | ||
| 13 | 954 | 5MU | 1MA | 958 | ||
| 14 | 955 | PSU | G | 917 | меняется направление спирали | |
| 15 | Антикодоновый стебель | 939 | C | G | 931 | |
| 16 | 940 | C | G | 930 | ||
| 17 | 941 | A | U | 929 | ||
| 18 | 942 | G | C | 928 | ||
| 19 | 943 | A | PSU | 927 | ||
| 20 | 944 | A | M2G | 926 | ||
| 21 | D-стебель | 910 | 2MG | C | 925 | |
| 22 | 911 | C | G | 924 | ||
| 23 | 912 | C | G | 923 | ||
| 24 | 913 | C | C | 922 | ||
| 25 | 914 | A | U | 908 | ||
| 26 | 915 | A | U | 948 | меняется направление спирали | |
| 27 | 918 | G | C | 956 | изолированная пара | |
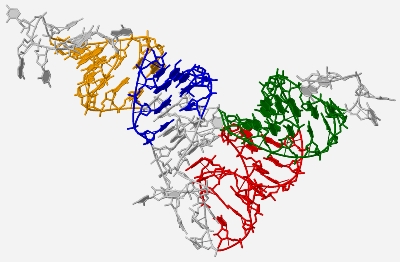
Рис. 6. Изображение структуры рнк с покрашенными стреблями. Скрипт в программе Jmol, с помощью которого можно получить данное изображение.
Поиск стэкинг-взаимодействий
В файле со стекинг взаимодействиями найдкны взаимодействия с наибольшим и наименьшим перекрытием.
13 uP/Ga 13.77
14 PC/GG 0.00
Полученные изображения с помощью программы analyse можно посмотреть на рисунках 7 и 8. Изображения, полученые в программе Jmol можно посмотреть на рисунках 9 и 10.
| © Mishchenko Polina 2016 |