В этом практикуме было необходимо при помощи программы Chromas прочитать последовательность ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру. Капиллярный секвенатор по Сэнгеру выдает файлы с хроматограммой и автоматически прочтенной последовательностью в формате .ab1. Мне были даны дла работы следующие файлы: Прямая цепь, Обратная цепь
Оба файла были открыты в программе Chromas. Для обратной цепи была открыта комплементарная последовательность (Edit>Reverse+Complement). Затем две хроматограммы были выровнены с помощью поиска подслов (find). Сначала были определены нечитаемые участки для обеих цепей, они оказались следующими: прямая цепь: 5' 1-15, 3' 711-714,716 (очень мал); обратная цепь: 5' 2-3(незначит.), нечитаемый 3' 700-722. Кроме того, 1 нуклеотид прямой цепи, (до которого идет нечитаемый участок) соответствует 39 нуклеотиду обратной цепи (здесь и далее речь будет идти о комплементарной последовательности). В целом обе хроматограммы достаточно качественные если не считать нечитаемых концов и содержат неболшое количество ошиок. Совмещая две хроматограммы, можно добиться минимального возможного количества ошибок в определенной нуклеотидной последовательности.
После удаления нечитаемых участков и замены проблемных нуклеотидов, в файл была записана данная итоговая последовательность, а в Jalview было получено выравнивание, представленное на последующем рисунке. Раскраска по нуклеотидам.

На примере следующих картинок попробуем понять почему некоторые нуклеотиды были заменены на другие.
 |
 |
||||||
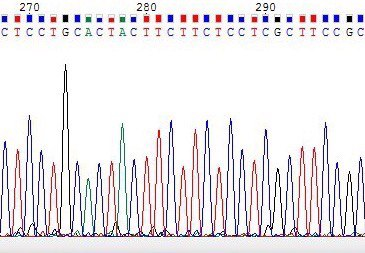
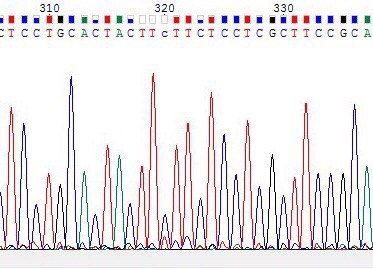
| На хроматограмме обратной цепи(справа) был неопределен 320 нуклеотид. Это связано с тем что шум достигал половины высоты пика, который сам по себе не высок. Соответствующий данному нуклеотиду 282 нуклеотид прямой последовательности однозначно определен как Цитозин. В связи с этим в обратной последовательности был поставлен С-нуклеотид. | |||||||
 |
 |
||||||
| Ситуация сходная с предыдущей, на обратной цепи(слева) был неопределен 371 нуклеотид, в связи с оносительно низким пиком и шумом. В обратной прямой посл-ти(слева) соответствующий данному 333 нуклеотид был определен как Цитозин, при этом пик довольно высокий. По этим причинам в прямой последовательности был поставлен С-нуклеотид. | |||||||
 |
 |
||||||
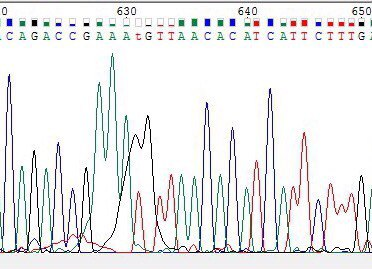
| В данном случае на хроматограмме обратной последовательности(слева) не был определен 631 нуклеотид, так как в данном отрезке хроматограммы наблюдается пятно в окраске Гуанина. На хроматограмме прямой последовательности(справа) соответствующий данному 593 нуклеотид был однозначно определен Тимин. В связи с этим в обратной последовательности был поставлен Т-нуклеотид. | |||||||
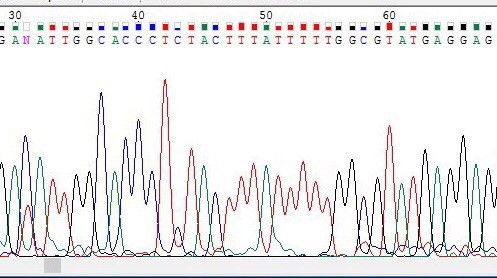 |
 |
||||||
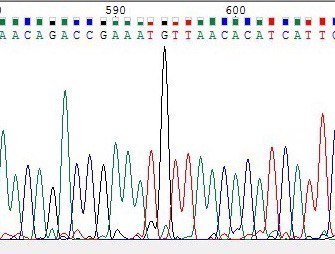
| На хроматограмме прямой последовательности(справа) были неопределены 3-8 нуклеотиды, так как на данном отрезке хроматограммы имеютсятся пятна в окраске Гуанина (приемущественно), а также эти нулеотиды являются одними из первых после нечитаемого участка. На хроматограмме обратной последовательности (слева) соответствующие данным 41-46 нуклеотиды был однозначно определены как Цитозин, Тимин, Цитозин, Тимин, Аденин и Цитозин. В связи с этим в обратной последовательности были поставлены C,Т,С,Т,А,С-нуклеотиды. | |||||||
В некоторых случаях уровень шума даже сравним с сигналами от нуклеотидов, что затрудняет чтение хроматограммы. Никаких резких ухудшений качества найдено не было, за исключение нескольких пятен. Передние и задние концы нечитаемы (из-за наличия коротких фрагментов, на которых отжигается праймер - презентация), но сопоставление двух хроматограмм позволяет делать вывод о верности определения нуклеотидов на тех или иных позициях. В целом моя хроматограмма была достаточно качественной, было небольшое количество неопределенных нуклеотидов как в прямой, так и в обратной последовательностях, случаев ошибок и полиморфизма не было обнаружено. Пятна наблюдались преимущественно на хроматограмме обратной цепи.
На рисунке расположенном ниже приведен пример нечитаемой хроматограммы. Причины могут различаться, в препарате возможно присутствует несколько ДНК, или он чем-то загрязнен и так далее. Скачать нечитаемую хроматограмму.
