Задание 1
С помощью команды seqret можно несколько файлов в формате fasta собрать в единый файл. Для этого был создан список mylist.txt, содержащий адреса исходных последовательностей. Далее с помощью команды:
С помощью команды seqretsplit можно один файл в формате fasta с несколькими последовательностями разделить на отдельные fasta файлы. В качестве исходного файла использовался файл sequences.fasta, полученный в прошлом пункте. С помощью команды:
С помощью команды seqret можно из файла с хромосомой в формате .gb три кодирующих последовательности по указанным координатам "от", "до", "ориентация" и сохранить в одном fasta файле. Использовался файл sequences.gb. Далее был создан список list2.txt, в котором содержит координаты кодирующих последовательностей. С помощью команды:
С помощью команды transeq можно транслировать кодирующие последовательности, лежащие в одном fasta-файле, в аминокислотные, используя указанную таблицу генетического кода, при этом результат — в одном fasta-файле. На вход команде подавался файл sequences2.fasta, полученный в предыдущем пункте:
С помощью команды transeq можно транслировать данную нуклеотидную последовательность в шести рамках.
Задание 2
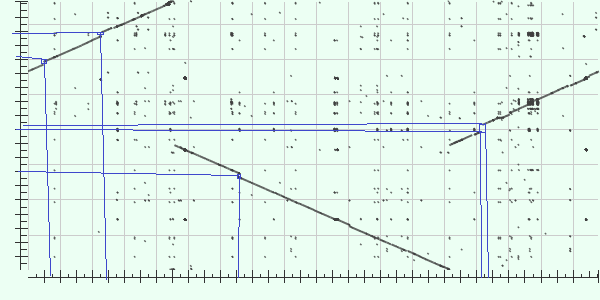
В этом задании надо было построить карту локального сходства для двух геномов и описать крупные эволюционные события на пути от общего предка.Для карты локального сходства была выбрана бактерия Ruminiclostridium (Clostridium) thermocellum DSM 1313 из первого семестра и бактерия того же вида Clostridium thermocellum ATCC 27405. С помощью алгоритма blastn и blast2seq на сайте NCBI была построена карта локального сходства для выбранных бактерий.
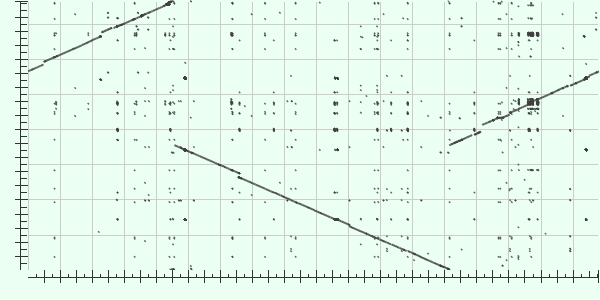
По оси Х в качестве query отложена бактерия DSM 1313, по оси Y в качестве subject бактерия ATCC 27405.
| Max score | Total score | Query cover | E-value | Ident | Identities |
| 2.975e+05 | 1.069e+07 | 96% | 0.0 | 99% | 155397/155702(99%) |
Предположим последовательности комплиментарны. Согласно карте локального сходства есть 1 крупное эволюционное событие - инверсия среднего куска ДНК. Она отмечена красным на рисунке.
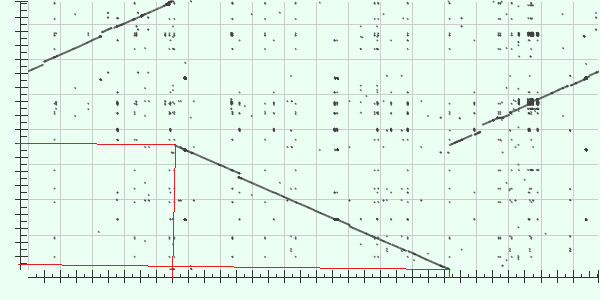
Также на карте видны небольшие делеции/вставки в последовательностях. Они отмечены синим на рисунке.