
Для выполнения задания мне был дан файл с ридами chipseq_chunk27.fastq. В начале я посмотрела нужна ли очистка файла. Для этого я пользовалась
программой FastQC.
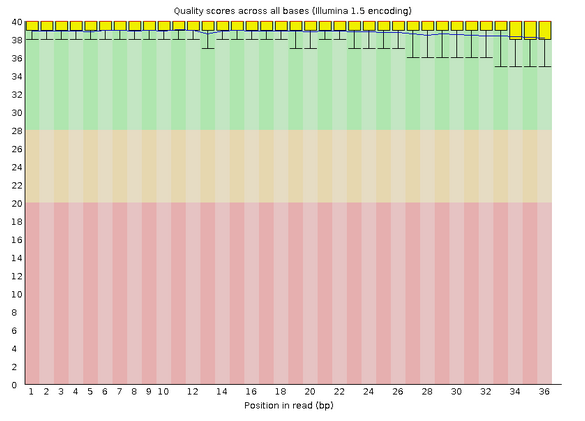
Так бал получен файл .html, который содержал информацию о ридах. Низкокачественных ридов найдено не было ("Sequences flagged as poor quality: 0"). И усы изначально находятся
в зеленой зоне, поэтому я не вижу смысла чистить файл. $ fastqc chipseq_chunk27.fastq

Картирование выполнялось с помощью команды BWA для проиндексированного референса:
$ /srv/databases/ngs/klukva$ bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk27.fastq > chipseq_chunk27.sam
Далее для анализа картирования было выполнено несколько команд. Сначала с помощью пакета samtools файл с выравниванием был переведен в бинарный формат .bam:
$ samtools view chipseq_chunk27.sam -bSo chipseq_chunk27.bam
Получившийся файл chipseq_chunk41.bam был подан на вход другой команде, чтобы отсортировать выравнивание ридов с референсом по координате в референсе начала рида (параметр -T позволяет записать временные файлы в файл chip_temp, т.к. иначе они выводятся в stdout):
$ samtools sort chipseq_chunk27.bam -T chip_temp -o chipseq_chunk27_sorted.bam
Далее полученный файл был проиндексирован:
$ samtools index chipseq_chunk27_sorted.bam
А затем я выяснила, сколько чтений откартировалось на геном:
$ samtools idxstats chipseq_chunk27_sorted.bam > chip.out
получив результат, записанный в файл chip.out. Таким образом, из 11018 ридов 10566 (95.90%) были откартированы на 21 хромосому, так что предполагаю, что риды с именно этой хромосомы были предложены мне для анализа.
Peak calling - компьютерный метод поиска областей генома, на которые откартировалось большое число ридов из данных эксперимента ChiP-Seq. Для поиска пиков использовалась программа MACS.
$ macs2 callpeak -n chipseq_chunk27 -t chipseq_chunk27_sorted.bam --nomodel
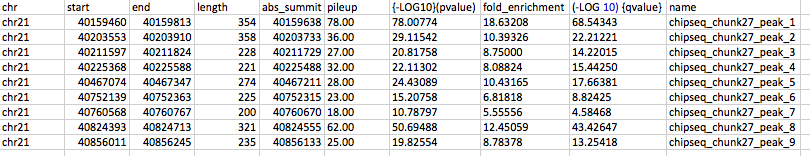
При этом было получено три файла (.xls, .narrowPeak, .bed ) с информацией о пиках. Всего было найдено 9 пиков на 21 хромосоме (таблица на рисунке 3).
Визуализация пиков в UCSC Genome Browser представлена на рисунке 4. Для визуализации в браузер подавался файл chipseq_chunk27_peaks.narrowPeak, в начало которого было дописано: track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 27" browser position chr21:40159460-40856250.
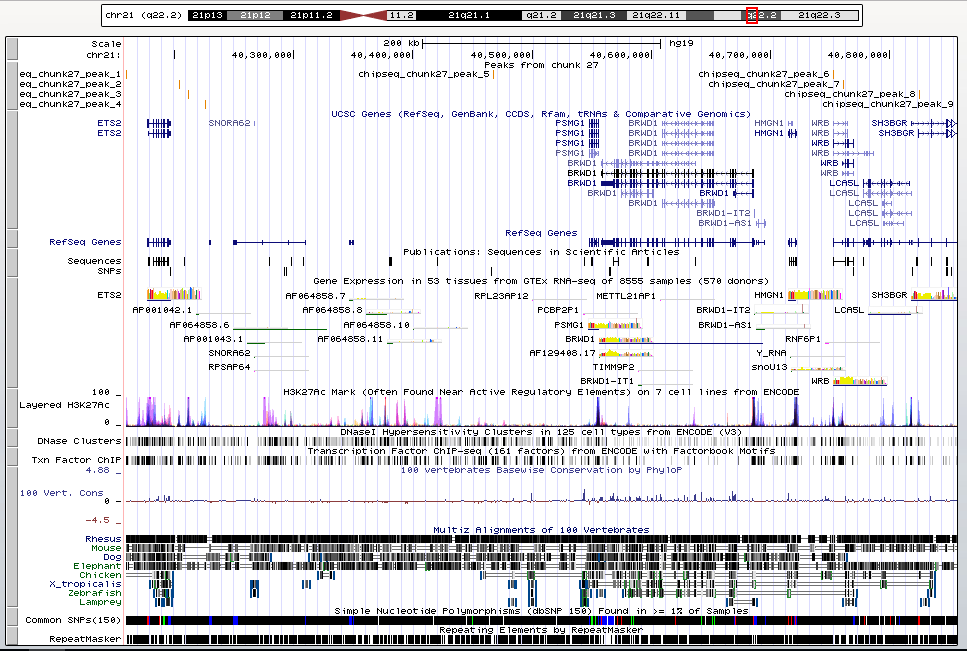
Каждому найденному пику соответствует число (-log10(p-value)), позволяющее оценить достоверность находки. При этом чем больше это число, тем меньше p-value, тем выше достоверность находки. Самая высокая достоверность у первого пика и восьмого пика. На рисунках 5 и 6 представлены изображения двух этих пиков в UCSC Genome Browser.

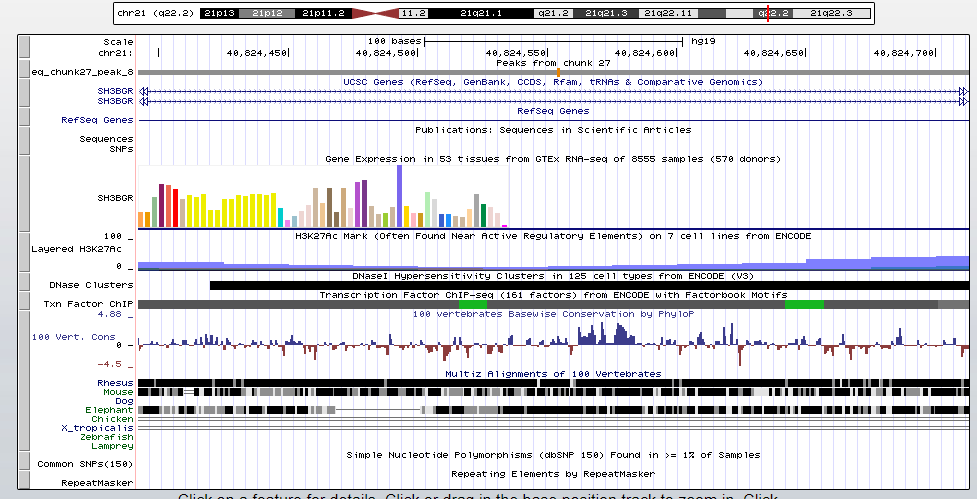
Восьмой пик пересекся с геном SH3BGR. Ширина первого пика-354, ширина восьмого-321. Что касается достоверности, -log10(p-value) у пиков соответственно: 78.00774 и 50.69488, что на порядок выше чем у остальных пиков.