Построение деревьев по нуклеотидным и аминокислотным последовательностям
Я получила последовательности 16S рибосомальной РНК каждой из бактерий из предыдущего практикума из файлов .frn из базы полных геномов NCBI.
Они были записаны в общий файл. Полученный файл с последовательностями я открыла в Jalview и построила выравнивание. Ниже представлено полученное
выравнивание:
Полученное выравнивание было импортированно в MEGA и методом Neighbour-joining было построено дерево, представленное ниже:

Полученное мною дерево отличается от дерева, которое было
получено в предыдущих практикумах. В реконструированном по 16S рРНК дереве НЕТ ветви отделяющей { CLOTE, CLOBA, BACAN } от { LACAC, LACLM, STRP1, STRPN }.
В реконструированном по 16S рРНК дереве ЕСТЬ ветвь отделяющая { LACAC, BACAN } от { CLOTE, CLOBA, LACLM, STRP1, STRPN }.
Не могу сказать достоверно чем было вызвано такое различие.
В качестве вывода могу сказать, что дерево, построенное по
последовательностям одной рРНК, может быть не совсем точным.
Построение и анализ дерева, содержащего паралоги
Паралоги - гомологичные белки из одного организма. Ортологи - гомологичные
белки из разных организмов или гомологичные белки, для
которых разделение их общего предка на линии, ведущей к ним, произошло в
результате видообразования. В данном задании надо было найти в своих бактериях достоверные
гомологи белка
CLPX_BACSU и построить дерево этих гомологов. Затем, считая, что дерево построено
верно, определить ортологов и паралогов, а также указать некоторые
эволюционные события.
Сначала я создала файл с протеомами моих бактерий и подала его на вход blastp.
Таким образом были найдены предполагаемые гомологи белка CLPX_BACSU.
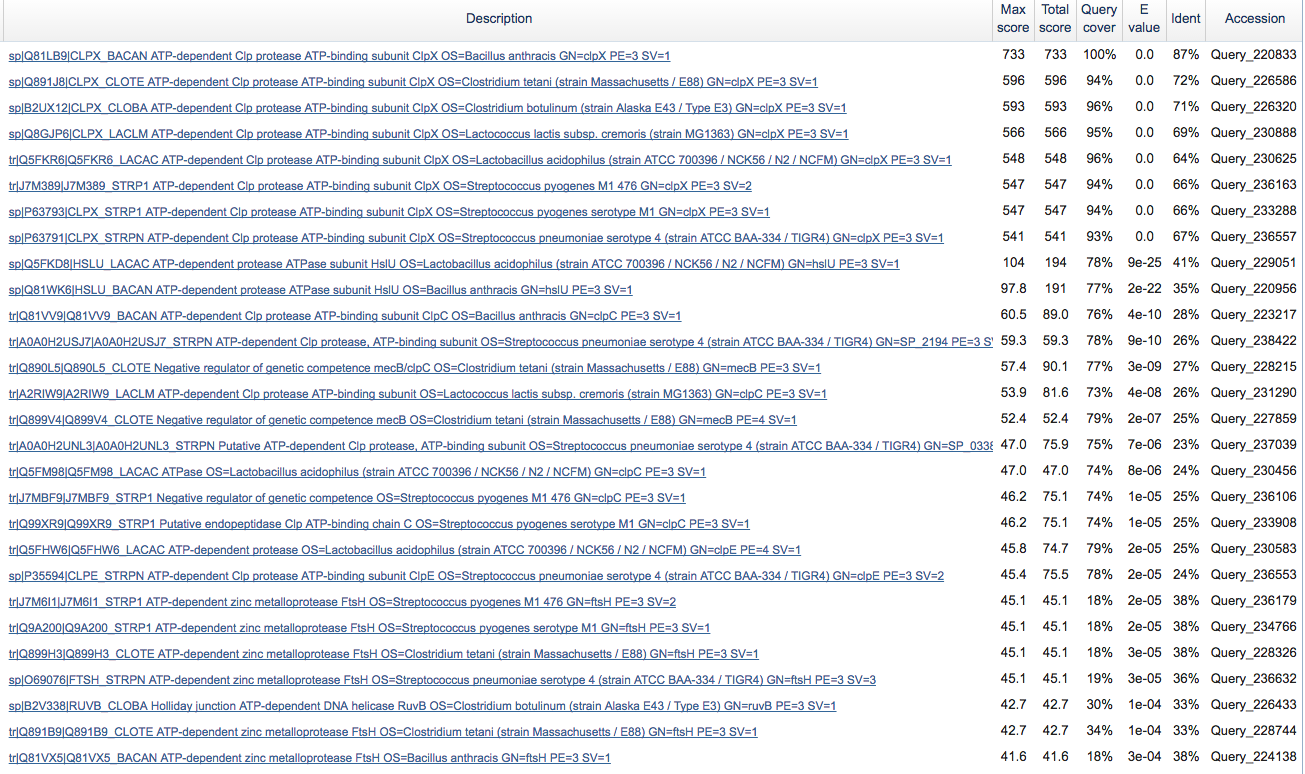
Последовательности найденных белков были загружены в Jalview и было построено выравнивание. файл с выравниванием Выравнивание приведено ниже:
По выравниванию в MEGA было построено дерево ( метод Neighbour-joining ). Оно изображено ниже:
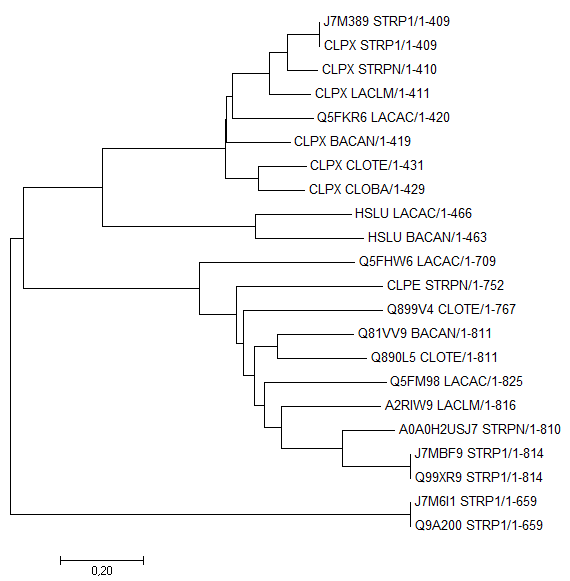
Clp — эндопептидаза, катализирующая гидролиз белков в
присутствии АТФ. Состоит из нескольких субъединиц: ClpP/ClpQ (обладают протеазной
активностью) и ClpX/ClpC/ClpE/и др. (связывают АТФ). Этот белковый комплекс найден
в бактериях, а также в митохондриях и хлоропластах эукариот.
HlsVU — АТФ-зависимая протеаза, присутствующая у многих бактерий.
Состоит из субъединиц: HslU (АТФ-зависимый шаперон и активатор второй субъединицы) и HslV
(обладает протеазной активностью).
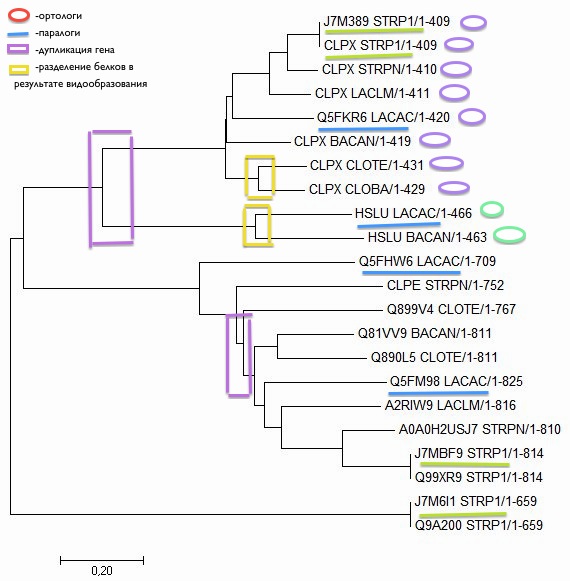
Таким образом, некоторые группы ортологов, представленные на дереве:
- J7M389_STRP1, CLPX_BACAN, CLPX_STRP1, CLPX_LACLM, CLPX_CLOTE, CLPX_CLOBA, CLPX_LACAC:
одна и та же субъединица ClpX;
- HSLU_LACAC, HSLU_BACAN: одна и та же субъединица HslU;
- CLPE_STRPN, Q81VV9_BACAN: одна и та же субъединица ClpE;
Все они кодируют определенную субъединицу, выполняющую одну и ту же роль в разных
бактериях.
Принимая во внимание тот факт, что HslU и СlpX имеют схожее строение и функцию,
некоторые паралоги, представленные на дереве:
- CLPX_BACAN и HSLU_BACAN;
- HSLU_LACAC и Q5FKR6_LACAC;
- CLPX_STRPN и CLPE_STRPN.
© Mishchenko Polina 2018




