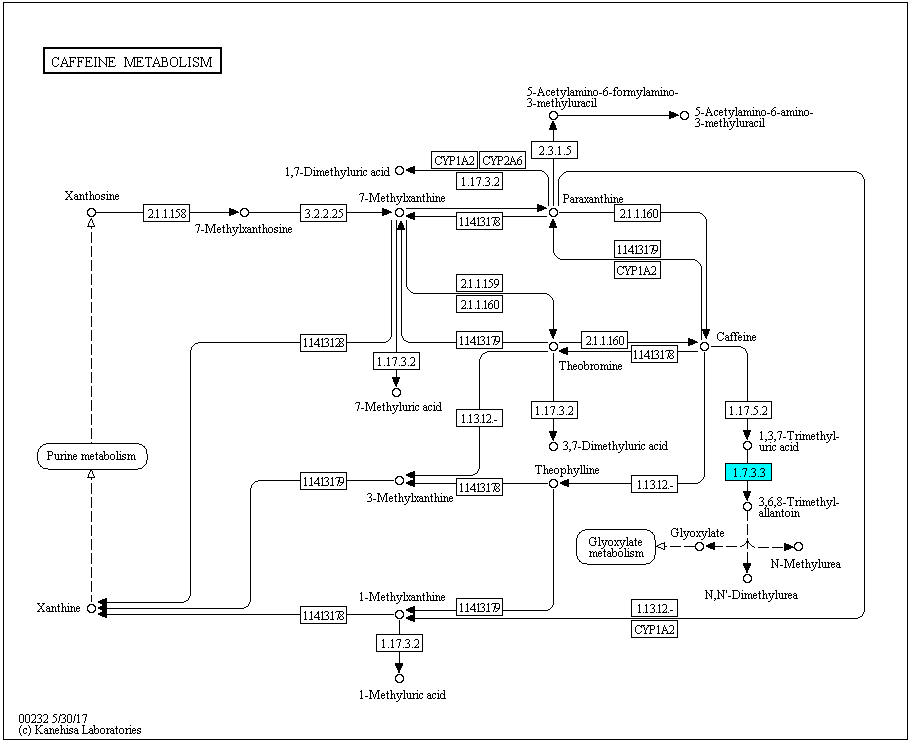
В базе данных KEGG был выбран метаболический путь Caffeine metabolis - метаболизм кофеина (Рис.1).
В нем была выбрана реакция EC:1.7.3.3 (помечена голубым цветом на карте),катализируемая двумя ортологическими рядами белков:
1. K00365 - urate oxidase (число генов:834 ;число белковых последовательностей (из них в UniProt):364(346))
2. K16838 - urate oxidase / 2-oxo-4-hydroxy-4-carboxy-5-ureidoimidazoline decarboxylase
(число генов:47 ;число белковых последовательностей (из них в UniProt):15(14))

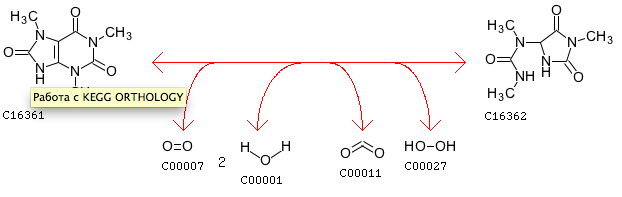
Схема указанной реакции (идентификатор: R00228) представлена на Рис.2.
1,3,7-Trimethyluric acid + Oxygen + 2 H2O <=> 3,6,8-Trimethylallantoin + CO2 + Hydrogen peroxide
На странице ортологических рядов я нашла ссылку на перечень идентификаторов белков в базе данных UniProt, относящихся к указанному ортологическому ряду. Далее я ввела полученные идентификаторы в окно поиска "Retrieve/ID mapping" сайта UniProt и получила на выходе таблицу со всеми нужными мне белками. Полученные последовательности были сохранены в файлы:k00365.fasta, k16838.fasta. Далее эти файлы были объеденены в один, после чего сервис Muscle построил множественное выравнивание. Полученное выравнивание было открыто в программе JalView и раскрашено по Clustalx, учитывающей химическую природу аминокислотных остатков. Полученный проект представлен в файле. Далее была проведена чистка в процессе которой я удаляла короткие, а также очень плохо выровненные белки. В результате была удалена 31 последовательность. Полученное выравнивание представлено в файле. Проект находиться в .jvp файле. Несмотря на чистку в итоге мы получили не самое лучшее выравнивание, белки одного ортологического ряда выровнялись между собой, при этом белки другого ряда выровнены намного хуже. Также если выравнивать отдельно белки разных ортологических белков, выравнивания будут намного лучше. Это может свидетельствовать о том, что белки одного ортологического ряда гомологичны, в то время как белки разных рядов не гомологичны. К сожалению мое выравнивание слишком большое и Jalview не экспортирует его изображение. Я считаю, что филогенетическое дерево построенное по данному выравниванию не отобразит достоверно филогению выбранных рядов, поэтому его построение не имеет смысла.