Для большей информативности, ниже приведена последовательность моего белка NP_603408.1: >gi|19703846|ref|NP_603408.1| D-lactate dehydrogenase
MQKTKIIFFDIKDYDKEFFKKYGADYNFEMTFLKVRLTEETANLTKGYDVVCGFANDNIN
KETIDIMAENGIKLLAMRCAGFNNVSLKDVNERFKVVRVPAYSPHAIAEYTVGLILAVNR
KINKAYVRTREGNFSINGLMGIDLYEKTAGIIGTGKIGQILIKILRGFDMKVIAYDLFPN
QKVADELGFEYVSLDELYANSDIISLNCPLTKDTKYMINRRSMLKMKDGVILVNTGRGML
IDSADLVEALKDKKIGAVALDVYEEEENYFFEDKSTQVIEDDILGRLLSFYNVLITSHQA
YFTKEAVGAITVTTLNNIKDFVEGRPLVNEVPQNQ
Результаты выполнения первого задания содержатся в Таблице 1.
| Таблица 1. Характеристики КОГов из хитов поиска CDD | ||||
| ID | E-value | Интервал | Название | Функциональная категория |
| COG1052 | 4.50e-120 | 4-334 | Lactate dehydrogenase or related 2-hydroxyacid dehydrogenase (Лактат-дегидрогеназа или родственная 2-гидроксикислотная дегидрогеназа) |
[Energy production and conversion] Выработка и преобразование энергии |
| COG0111 | 3.49e-77 | 1-333 | Phosphoglycerate dehydrogenase or related dehydrogenase (Фосфоглицерин дегидрогеназа или родственная дегидрогеназа) |
[Coenzyme transport and metabolism] Коэнзимный транспорт и метаболизм |
| COG0373 | 2.56e-04 | 143-204 | Glutamyl-tRNA reductase (Глутамил-тРНК редуктаза) |
[Coenzyme transport and metabolism] Коэнзимный транспорт и метаболизм |
| COG0499 | 2.01e-03 | 147-254 | S-adenosylhomocysteine hydrolase (S-Аденозилгомоцистеин гидролаза) |
[Coenzyme transport and metabolism] Коэнзимный транспорт и метаболизм |
При помощи базы данных STRING я получила визуализацию геномного окружения для КОГа с лучшим e-value (COG1052). Для поиска были установлены следующие параметры: минимальная требуемая оценка взаимодействия - 4.000; максимальное число отображаемых взаимодействий со входной последовательностью - не более чем 10 взаимодействующих белков. На рисунке 1 представлен граф взаимодействий, на рисунке 2 расшифровка взаимодействий, а на рисунке 3 найденные КОГи и их взаимодействия.
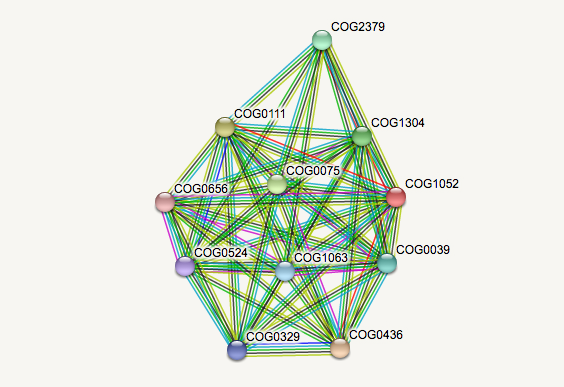
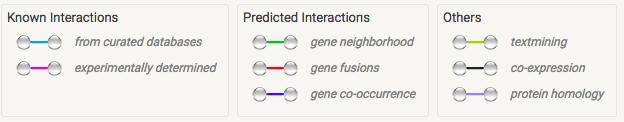
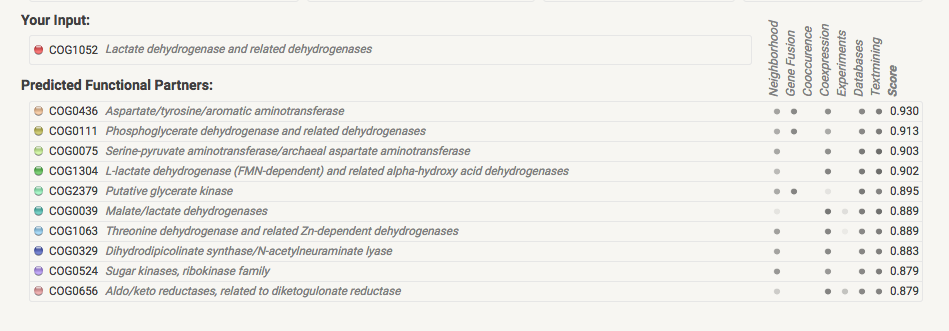
На рисунке один представлен граф, вершинами которого являются КОГи а ребра графа отражают наличие свидетельств о существовании связи между их белками. На рисунке 2 показано что обозначают разные цвета ребер графа. Голубой (Из курируемых баз данных) и розовый (Доказанные экспериментально) цвета обозначают известные взаимодействия. Зеленый (Соседство генов), красный (Слияние генов) и синий (Совместное нахождение генов) цвета для предсказанных взаимодействий. Оставшиеся цвета : желтый (Совместные упоминания), черный (Совместная экспрессия) и фиолетовый (Гомология белков) для остальных типов. Из таблицы на рисунке 3 видно, что экспериментально доказанных взаимодействий 3 из 10, но все 10 взяты из различных баз данных. Все находки подтверждены совместным упоминанием в литературе.
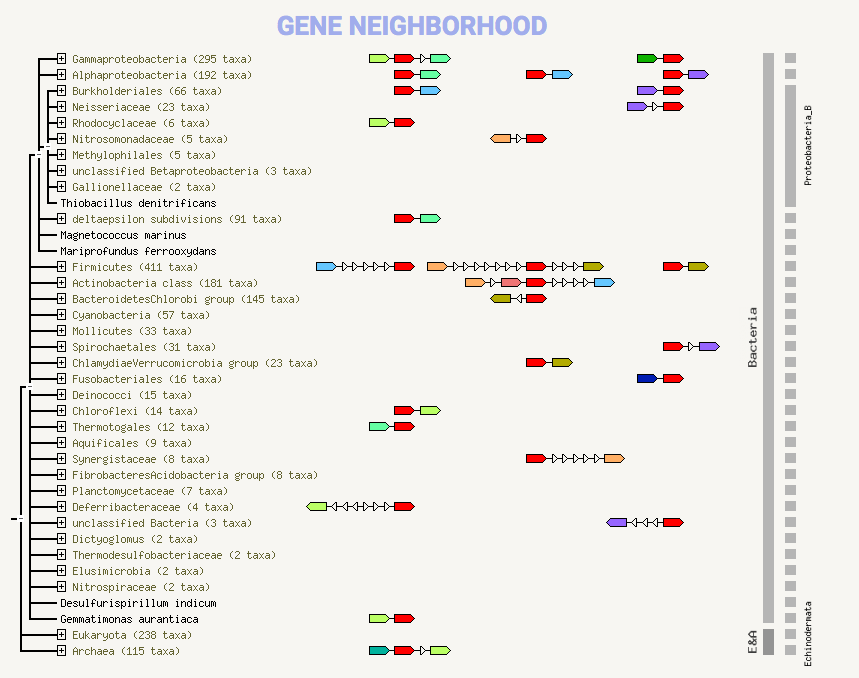
На рисунке 3 мы видим, что столбец Neighborhood тускло выражен, это первый сигнал что нет консервативного окружения. На 4 рисунке (ниже) мы не видим повторяющихся паттернов. Так что ответом на вопросы будет то, что консервативного окружения нет.

Я использовала поиск по fasta-последовательности белка сервиса AmiGO. Лучшая находка - D-lactate dehydrogenase из Shewanella oneidensis MR-1 с e-value = 8.1e-76, identities = 150/328 (45%), positives = 217/328 (66%). Могу предположить что это очень близкий гомолог к нашему белку. Его идентификатор в Uniprot - Q8EI78. Он имеет две ассоциации. В таблице 2 указаны термины GO, отнесенные к нашему белку.
| Таблица 2. | ||||
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Biological process | GO:0006113 | fermentation | Ферментация | ISS |
| Molecular function | GO:0008720 | D-lactate dehydrogenase activity | Д-Лактат-дегидрогеназная активность | ISS |
Код типа достоверности ISS расшифровывается как Inferred from Sequence Similarity. Данный код присваевается если анализ был основан на последовательностях и обзор доказательств и аннотаций делается вручную. В итоге мы нашли ближайший гомолог нашего белка с терминами GO.