Определение биологической роли TF в бактерии
Работа проводилась с бактерией Klebsiella pneumoniae subsp. pneumoniae MGH 78578. Использовался файл LsrR.fasta с участками связывания транскрипционного фактора LsrR в мембране.
Поиск мотива связывания производился при помощи сервиса MEME. Данные представлены в следующей таблице:
| Паттерн | Лого | PWM | e-value |
| ффф | .png) |
файл | 9.7e-056 |
Далее с использованием сервиса tomtom проводился поиск похожего мотива по БД RegTransBase. Параметры лучшей находки приведены в следующей таблице:
| ТФ | Выравнивание | PWM | e-value |
| Predicted lactose transport regulator in Rhizobiales(предсказанный регулятор транспорта лактозы) | 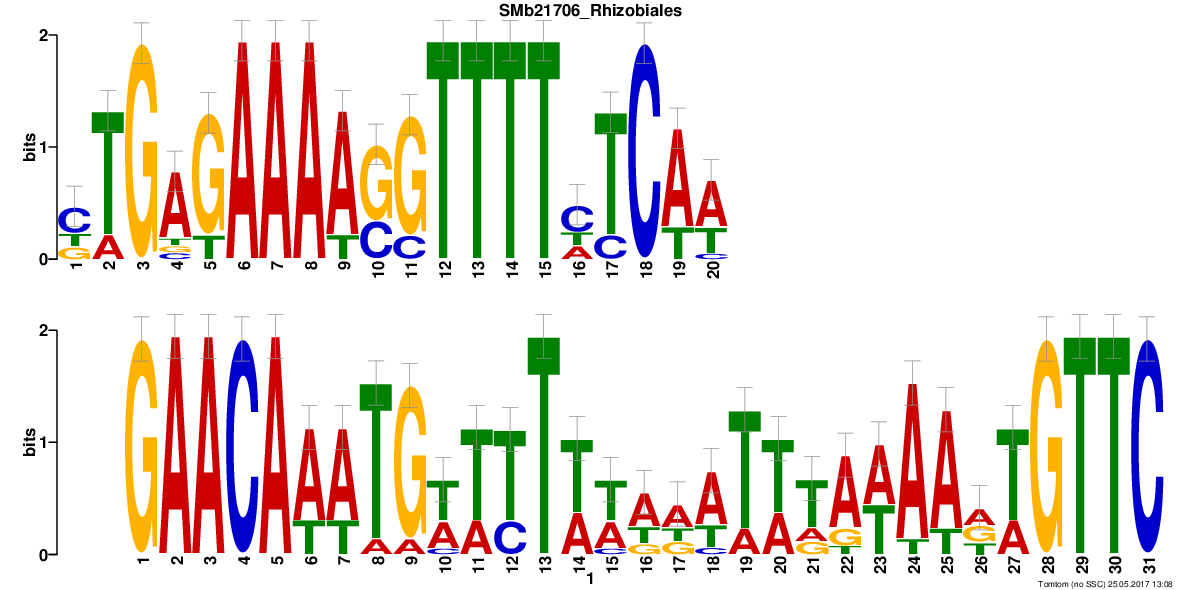 |
файл | 2.17e-02 |
Из выранивания выданного сервисом Tomtom видно, что найденный мотиф короче исходного на 11 нуклеотидов и совпадающих мотивов очень мало. Также найденный ТФ является предсказанным, что также заставляет усомниться в достоверности.
Исходный мотив был передан сервису FIMO. В качестве БД, по которой проводилось сканирование на поиск схожих мотивов, была выбрана Upstream Prokaryotic для исследуемой бактерии Klebsiella pneumoniae subsp. pneumoniae MGH 78578
Все находки FIMO в текстовом формате лежат в файле fimo.txt. Список координат находок и их p-value представлен в файле fimo.xlsx. Первые несколько находок также представлены на следующем рисунке:
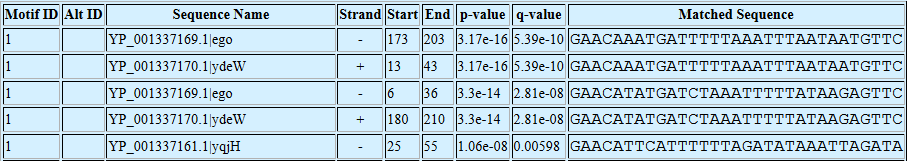
Поиск проводился только по upstream regions, потому что связывание факторов транскрипции происходит именно в участке, предшествующему гену(upstream).
Мы видим что у двух первых найденных гена имеется по два сайта вязывания. Координаты гена ego: complement(3868021..3868051), гена ydeW: 3869567..3870597. Был проведен поиск по БД STRING, в результате было показано, что эти гены входят в один метаболический путь:
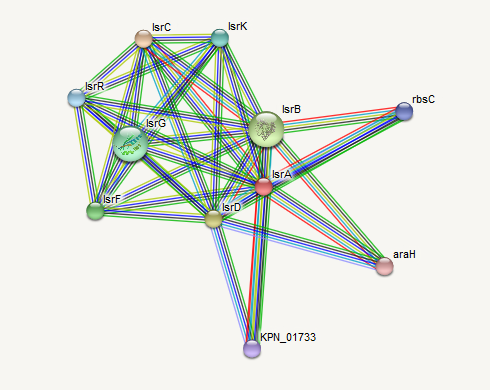

IsrA(ген ego) - АТФаза; Часть комплекса ABC-транспортеров LsABCD, участвующая в импорте автоиндуктора 2.
IsrR(ген ydeW) - регулятор транскрипции семейства SorC, регулирует транскрипцию многих различных генов.
Влияние метилирования на связывание ТФ со своим сайтом
Программа fuzznuc использовалась для поиска сайтов метилирования, пересекающихся с двумя найденными FIMO мотивами (были взяты сами мотивы и участки ± 12 нуклеотидов по бокам от них)(ego.fa)(ydeW.fa). В участках искались сайты из файла MT-sites.pat, содержащего сайты метилирования. В результате были получены файлы: ego.out, ydeW.out. Для гена ego было найдено 86 пересечений, а для гена ydeW - 90. При этом ногие из них состоят из малого числа нуклеотидов, так что не могут считаться достоверными.
В БД REBASE был найден геном нашей бактерии и закодированные в нем метилтрансферазы. Для некоторых из них была указано специфичность, эти последовательности совпадали с pattern173, pattern291 и pattern513. При этом ни один из этих сайтов не был найден в наших участках, что говорит о том, что связывание ТФ со своими сайтами никак не регулируется их метилированием.
© Mishchenko Polina 2017