Секвенирование по Сэнгеру
1.Сборка последовательности по прочтениям
Ссылка на исходные файлы:
forward-read reverse-readРезультат:
консенсусная последовательностьХарактеристика хроматограммы:
| Участок в начале | Участок в конце | |
| Прямая цепочка | проблем нет | 568-669 нуклеотид |
| Обратная цепочка | 1-177 нуклеотид | проблем нет |
*При автоматическом пороге в 30 нуклеотидов
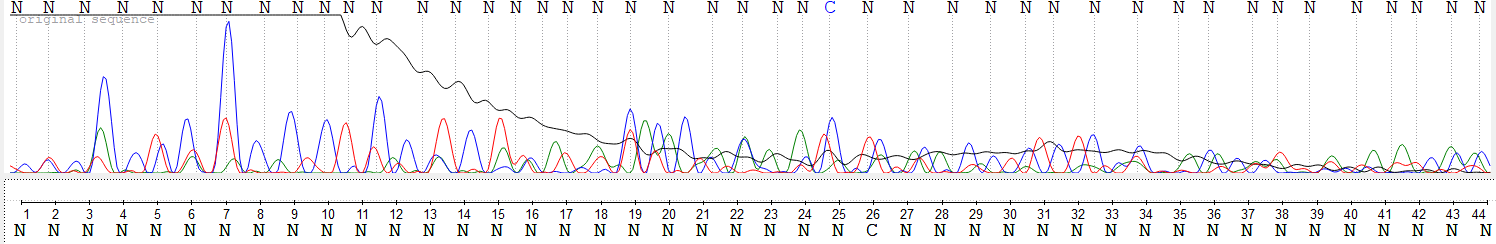
Визуальная оценка неравномерности сигнала: для прямого и обратного прочтений характерны четкие, соотносимые по высоте пики в середине и неравномерные, сильно разбросанные по высоте пики в начале и конце обоих последовательностей.
Визуальная оценка неравномерности шума: в начале и конце прямого и обратного прочтений пики имеют разнообразную, "неправильную", форму, часто пересекаются или сливаются, в середине последовательности пики а хроматограмме хорошо различимы, сигналы небольшого фонового загрязнения находятся на очень низком уровне.
В целом можно сказать, что оба прочтения достаточно хорошего качества, а вырезанные проблемные участки каждого прочтения можно восстановить по другой цепи.
Выравнивания:
Выравнивание последовательностей прямой и обратной цепей с консенсусной последовательностью:
fasta-файлВыравнивание отредактированных последовательностей прямой и обратной цепей с референсной последовательнстью:
fasta-файлПосле выравнивания прямого и обратного прочтения на прямое прочтение и редактирования всех нечитаемых участков вручную, с помощью blastn NCBI и полученной консенсусной последовательности была найдена последовательность генов идентичная консенсусной — Scoloplos acutissimus voucher W.44175.001 18S ribosomal RNA gene.
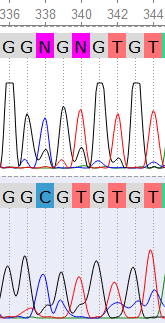
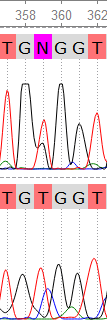
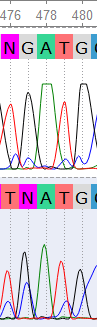
Исправление полиморфизмов:
2.Пример нечитаемой хроматограммы
В качестве примера был взят файл L46_COI_F_A07_WSBS-Seq-07-10-16.ab1 из директории /bad. Хроматограмма этого файла очень короткая, поэтому была рассмотрена целиком. На хроматограмме отчетливо видно, особенно в начале, сильное загрязнение сигналом G нуклеотида (загрязнение красителем), также ближе к концу видно практически отсутствие сигналов, это может быть связано с загрязнениями образца ингибиторами, либо с нехваткой продуктов ПЦР.