Данный мини-обзор представляет из себя работу с геномом и протеомом бактерии Dickeya solani с использованием программирования на языке Python и функционала электронных таблиц.
Dickeya solani – грамотрицательная бактерия вызывающая черную ножку и мягкую гниль картофеля [1]. Этот вид впервые обнаружили при выращивании клубней картофеля ( Solanum tuberosum ) в Европе в начале 2000-х годов [2]. Бактерии из рода Dickeya могут находиться внутри растения, не вызывая симптомов заболевания. Когда условия окружающей среды становятся благоприятными, бактерии переходят из латентной фазы в патогенную. В патогенной фазе бактерии начинают быстро размножаться и вырабатывать ферменты, разрушающие клеточную стенку растений [3]. Dickeya solani из-за своей высокой вирулентности вызывает значительные потери в сельском хозяйстве во всем мире. Поэтому изучение генома этой бактерии является необходимым для защиты картофеля и поддержания его высокой урожайности.
В данной работе приведены и проанализированы результаты некоторых вычислительных исследований генома и протеома бактерии.
Данные о последовательности генома были взяты с сайта национального центра биотехнологической информации (NCBI) (см. [1] в сопроводительных материалах).
Для поиска процента встречаемости нуклеотидов использовался сценарий, написанный на Python 3.10.12 (см. [2] в сопроводительных материалах). Для анализа распределения белков по их длинам и для анализа распределения генов использовались электронные таблицы Google Tables (см. [3] в сопроводительных материалах).
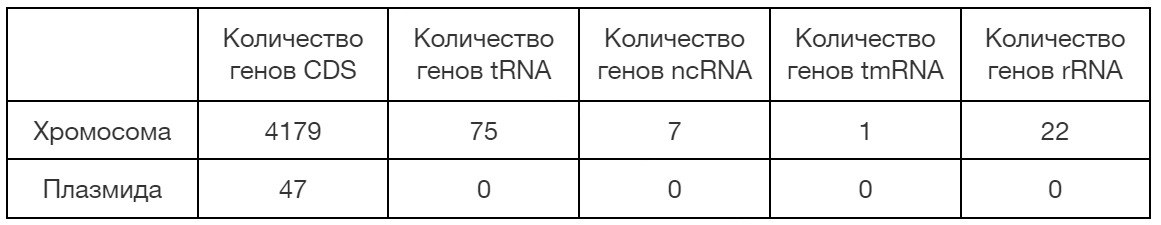
Геном Dickeya solani состоит из одной кольцевой хромосомы и одной плазмиды (см. таблицу 1). В результате анализа типов кодирующих последовательностей выявлено, что на хромосоме большинство генов относятся к CDS (coding sequence — последовательность нуклеотидов, кодирующая белок). Также на хромосоме обнаружены 4 типа RNA (tRNA, ncRNA, tmRNA, rRNA). Общее количество генов разных типов RNA: 105. Это составляет примерно 2.5% от всех найденных генов на хромосоме (см. [3] в сопроводительных материалах, лист RNA_stat). Это заметно меньше, чем количество белок-кодирующих генов (CDS).
На плазмиде было найдено 47 белок-кодирующих генов (CDS). Количество CDS на плазмиде составляет 0,01 долю количества CDS на хромосоме (см. [3] в сопроводительных материалах, лист Plasmid_stat). Это существенная разница.
Таблица 1. Информация о количестве генов на хромосоме и плазмиде Dickeya solani
Анализ процента встречаемости нуклеотидов проведен для проверки выполнения правила Чаргаффа, была написана программа подсчитывающая число и процент встреч A, T, C, G на хромосоме (см. таблицу 2).
Таблица 2. Количество и процент встречаемости нуклеотидов A, T, C, G на хромосоме
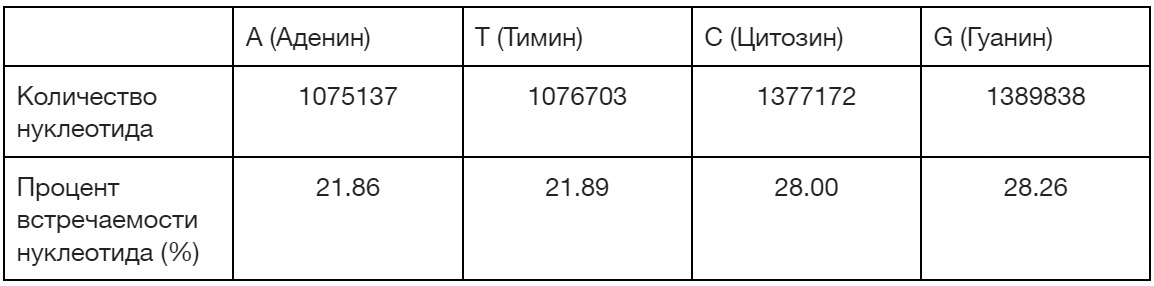
Процент встречаемости A примерно равен проценту встречаемости T (21.86% ≈ 21.89%), процент встречаемости C примерно равен проценту встречаемости G (28.00% ≈ 28.26%). Исходя из полученных результатов делаем вывод, что правило Чаргаффа выполняется. Также можно увидеть, что количество пар GC превышают количество пар AT, следовательно, в ДНК больше тройных связей между комплементарными нуклеотидами.
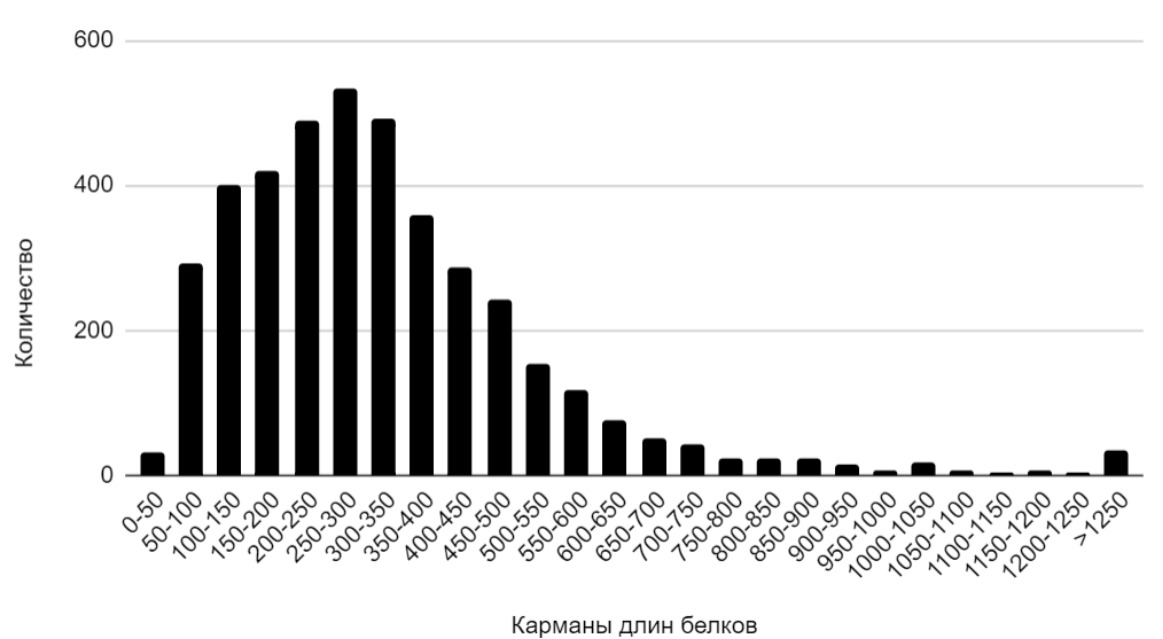
Была составлена гистограмма длин белков на основе данных таблицы особенностей генома данной бактерии (см. рис 1).

На гистограмме видно, что для протеома Dickeya solani классического “двугорбого” распределения длин белков не наблюдается. Наибольшее количество белков имеют длину в диапазоне от 250 до 300 аминокислот. С ростом длины белка их количество уменьшается. Длину меньше 50 аминокислот имеет малое количество белков.
1. Данные NCBI по геному и протеому бактерии Dickeya solani
2. Сценарий на Python 3.10.12 для поиска процента встречаемости нуклеотидов
3. Таблица анализа белков и распределения генов
1. Toth, I. K.; Van Der Wolf, J. M.; Saddler, G.; Lojkowska, E.; Hélias, V.; Pirhonen, M.; Tsror Lahkim, L.; Elphinstone, J. G. (2011). "Dickeya species: An emerging problem for potato production in Europe". Plant Pathology. 60 (3): 385. doi:10.1111/j.1365-3059.2011.02427.x.
2. van der Wolf J. M., Nijhuis E. H., Kowalewska M. J., Saddler G. S., Parkinson N., Elphinstone J. G., et al.. (2014). Dickeya solani Sp. nov., a pectinolytic plant-pathogenic bacterium isolated from potato (Solanum tuberosum). Int. J. Syst. Evol. Microbiol. 64, 768–774. doi: 10.1099/ijs.0.052944-0
3. Hugouvieux‐Cotte‐Pattat, N. (2016) Metabolism and virulence strategies in Dickeya–host interactions. Prog. Mol. Biol. Transl. Sci. 142, 93–129.