Из базы данных Pfam исходя из критериев выбрала домен PF00372 (Hemocyanin_M) – Hemocyanin, copper containing domain (медьсодержащий домен гемоцианина).
Скачала выравнивание seed, состоящее из 73 последовательностей. Далее удалила 2 последовательности идентичные на 90% (осталась 71 последовательность). При окрашивании Clustal и уровне IC=90% выделяются рядом стоящие консервативные колонки, по ним составила паттерн Jalview: [RK][D][PSA].{2}[YFW]. Мотив нашелся ровно в 71 последовательности (друг под другом).
Перевела паттерн в формат Prosite: [RK]-D-[PSA]-x(2)-[YFW]. В результате работы ScanProsite мотив нашелся 1032 раза в 1000 последовательностей (то есть он встречается не только друг под другом в выравнивании). После поиска мотива в выравнивании этих последовательностей видно, что он (мотив) не находится в выравнивании друг под другом и также распознается в большем количестве мест, чем всего последовательностей.
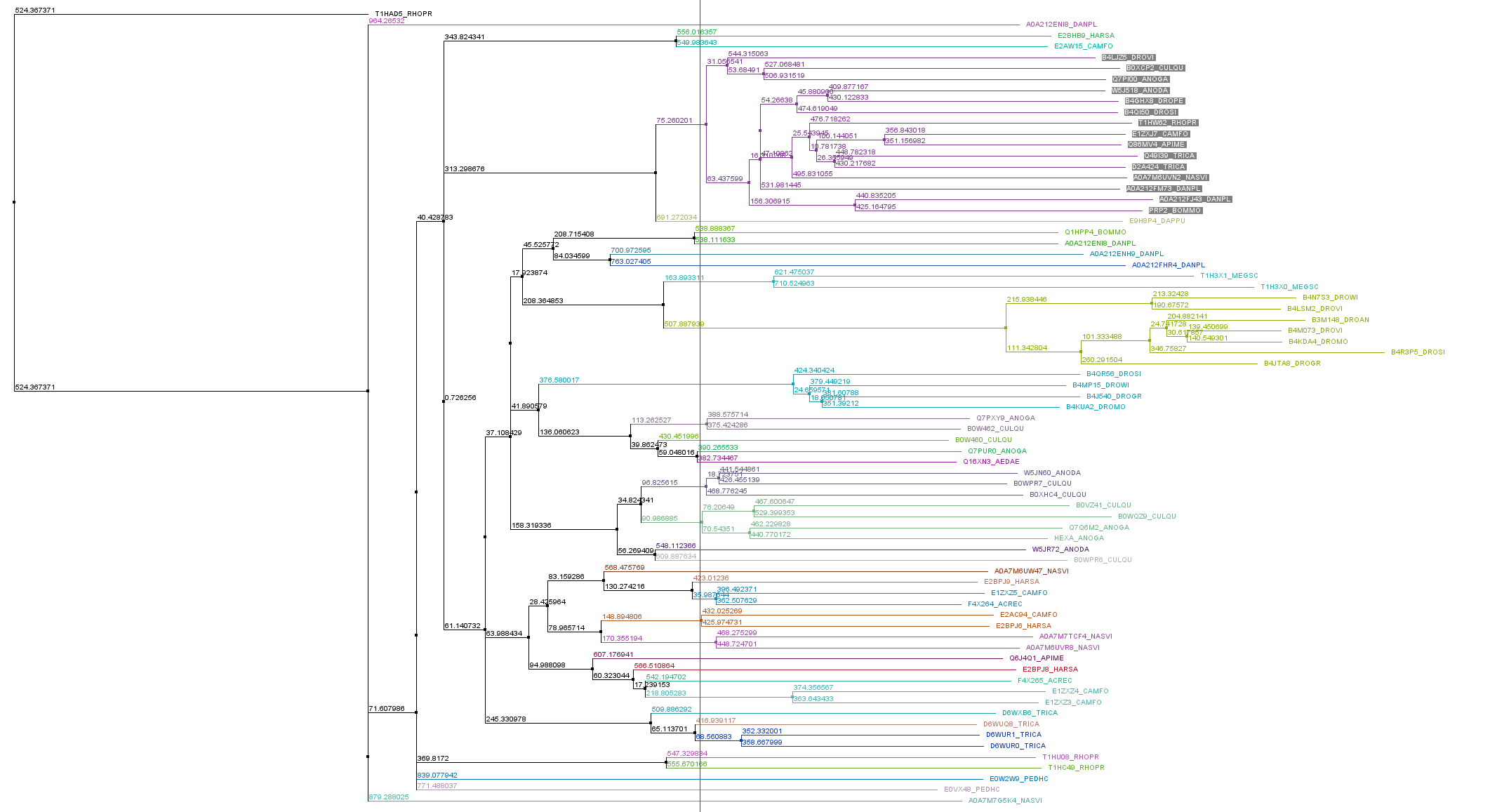
Далее построила филогенетическое дерево методом NJ и выбрала кладу (выделенные на рис.1 последовательности).

В отделенных 15 последовательностях нашла консервативный мотив: [H][H].[H][W][H][L][V][Y][P]. Этот мотив нашелся только в последовательностях, входящих в кладу. Делаю вывод, что этот мотив консервативен для этой клады.

Выбрала из списка АС: Q7VDL2 – белок ингибирующий клеточные деления (ингибирует полимеризацию белка, образующего Z-кольцо) из Цианобактерии (Prochlorococcus marinus (strain SARG / CCMP1375 / SS120). Выбрала Swiss-Prot в качестве базы данных и запустила PSI-BLAST.
На третьей итерации не нашлось новых белков, все имеют одинаковые названия. Таким образом, PSI-BLAST хорошо выделил семейство за 3 итерации.
Таблица 1. Результаты поиска PSI-BLAST по идентификатору Q7VDL2
| Номер итерации | Число находок выше порога (0.005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 146 | Q9AG20.1 | 0.005 | A8GFG7.1 | 0.005 |
| 2 | 188 | B6JKX0.1 | 7e-08 | - | - |
| 3 | 188 | Q9ZM51.1 | 2e-12 | A7H8E6.1 | 0.014 |
Отобрала 156 последовательностей; для 51 последовательности из них примененила программу MEME. Нашлось 4 мотива, часть одного из них я выделила во 2 задании у отдельной клады.
Выдача программы MEME: html и txt
Далее запустила программу FIMO, нашла 814 достоверных находок мотивов в 156 последовательностях. То есть некоторые мотивы повторялись, это видно из выдачи MEME (в последовательностях два раза встречается сочетание двух мотивов рядом).
Выдача программы FIMO: html
Моя бактерия: Dickeya solani. Хочу узнать представленность сайта GATC в геноме бактерии, запустила программу:
cbcalc -s sites.txt -M -o result.tsv GCF_002846995.1_ASM284699v1_genomic.fna
-s sites.txt – указывает файл с сайтами для анализа, -M – включает марковскую модель для расчёта ожидаемых частот, -o result.tsv – задаёт имя выходного файла.
Далее по таблице из результатов работы программы составила гистограмму контрастов obs/exp по методу Карлина с соав.
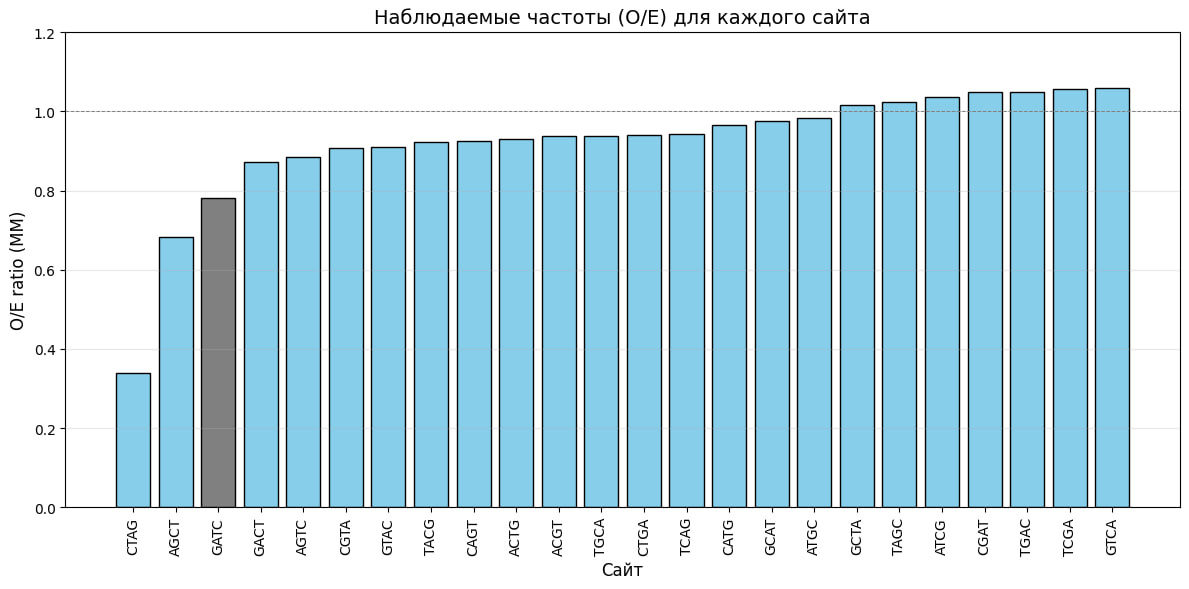
Видно, что сайт GATC "в дефиците". Возможно это связано с защитой от собственных рестрикционных ферментов.