
Создала выравнивание последовательностей цитохромов B из выбранных при выполнении предыдущего практикума животных:
muscle -align cyb.fasta -output cyb-alignment.fastaДалее с помощью библиотеки BioPython изменила формат файла с fasta на phylip-relaxed для дальнейшей работы программы FastME (получила файл cyb.phy).
Реконструировала дерево с помощью программы FastME:
Использовав модель p-distance: fastme -pp -i cyb.phy -o cyb-p.treИспользовав модель MtREV: fastme -pm -i cyb.phy -o cyb-m.tre
Реконструировала дерево программой IQ-Tree: iqtree -s cyb.phy
С помощью программы iTOL получила изображение деревьев, далее нужно их сравнить с деревом, построенным на основе систематики NCBI.

|

|
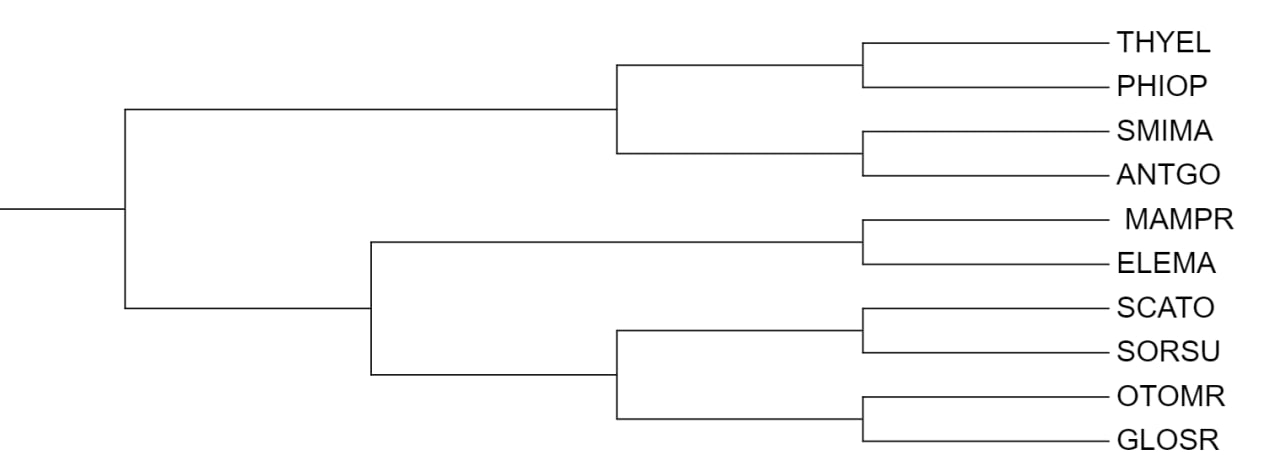
| Рис. 1. Дерево, построенное на основе систематики NCBI | Рис. 2. Дерево, реконструированное программой FastME, модель p-distance |
Сравнение Рис.1 и Рис. 2: деревья имеют одинаковую топологию

|
|
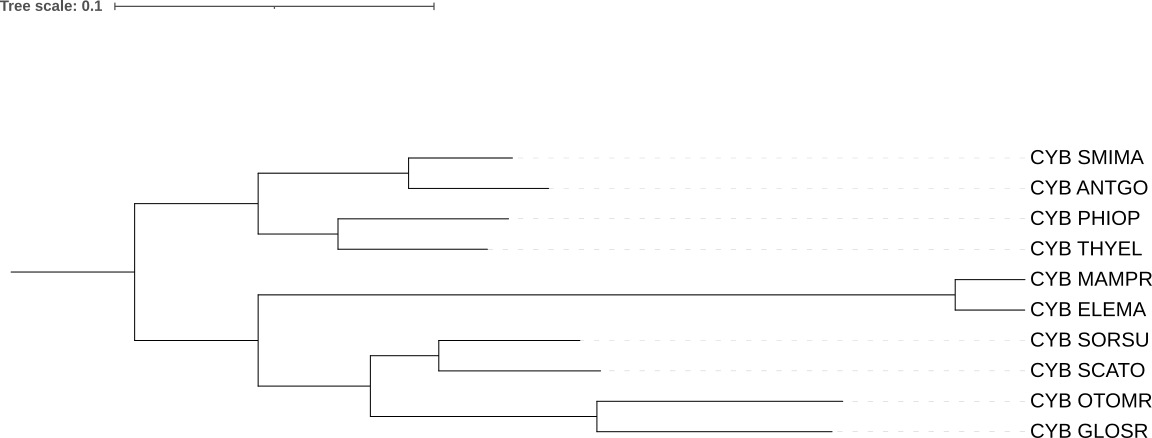
| Рис. 1. Дерево, построенное на основе систематики NCBI | Рис. 3. Дерево, реконструированное программой FastME, модель MtREV |
Сравнение Рис.1 и Рис. 3: деревья имеют одинаковую топологию

|
|
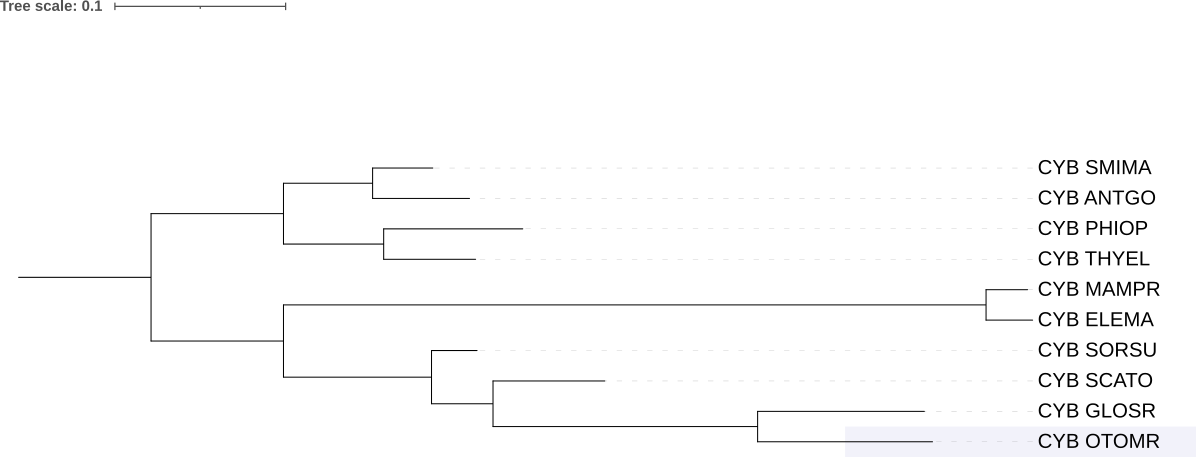
| Рис. 1. Дерево, построенное на основе систематики NCBI | Рис. 4. Дерево, реконструированное программой IQ-Tree |
Сравнение Рис.1 и Рис. 4: На Рис. 1 (дерево, построенное на основе систематики NCBI) SCATO и SORSU образуют отдельную кладу, а на Рис. 2 (дерево, реконструированное программой IQ-Tree) ветвь SORSU отходит раньше, чем ветвь SCATO.