Выбрала 7 бактерий отдела Pseudomonadota: ACICJ, AGRFC, AROAE, BARHE, BORPE, BRUSU, BURMA.
Создала локальную базу данных: makeblastdb -in prot.fasta -dbtype prot
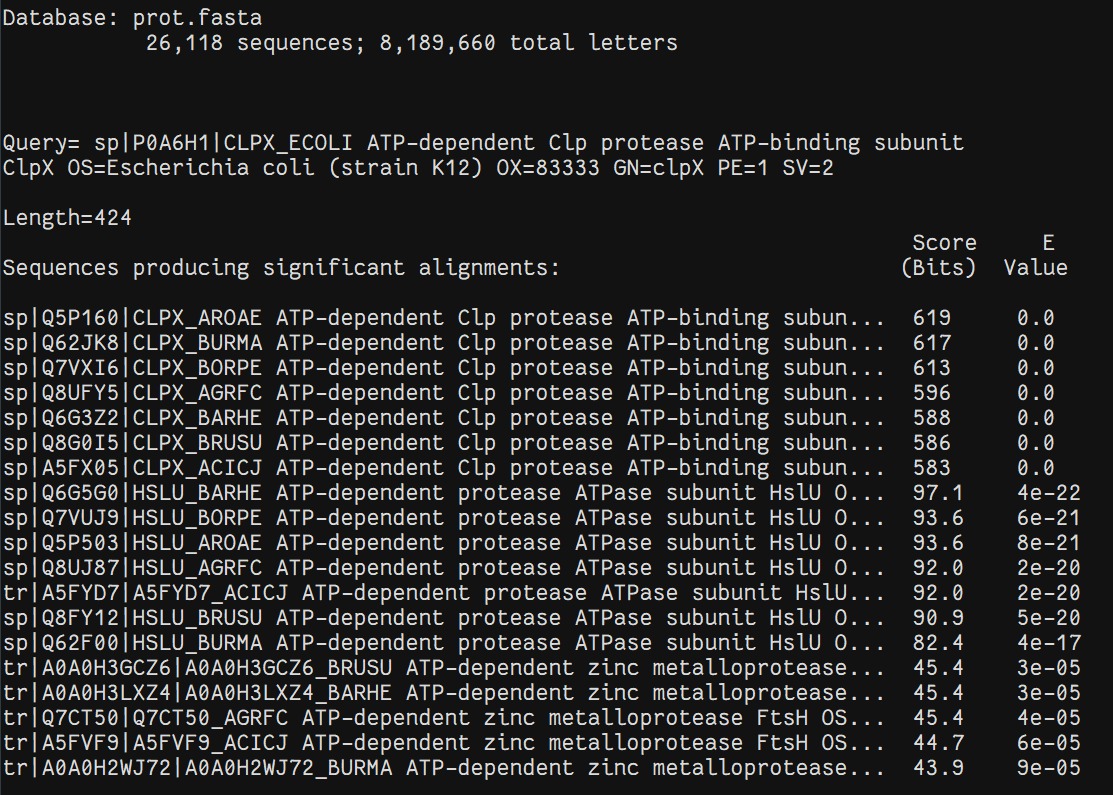
Нашла в выбранных протеомах достоверные гомологи белка CLPX_ECOLI: blastp -task blastp -query CLPX_ECOLI.fasta -db prot.fasta -out blastp.out -evalue 0.0001

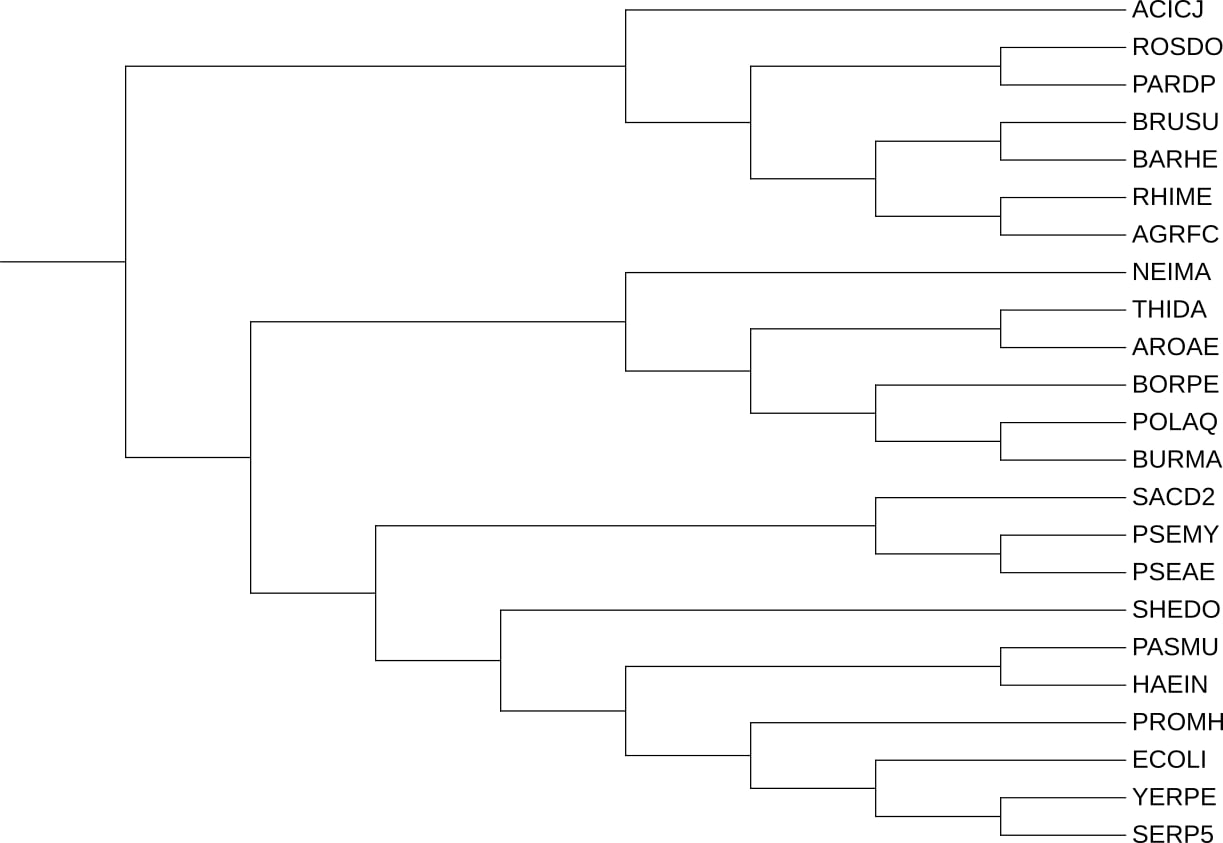
Реконструировала дерево программой FastME, модель p-distance: дерево в формате Newick
Ортологи: HSLU_BRUSU и HSLU_BARHE, HSLU_BORPE и HSLU_BURMA, CLPX_AGRFC и CLPX_BRUSU
Паралоги: CLPX_BURMA и HSLU_BURMA, CLPX_BORPE и HSLU_BORPE, CLPX_BRUSU и HSLU_BRUSU
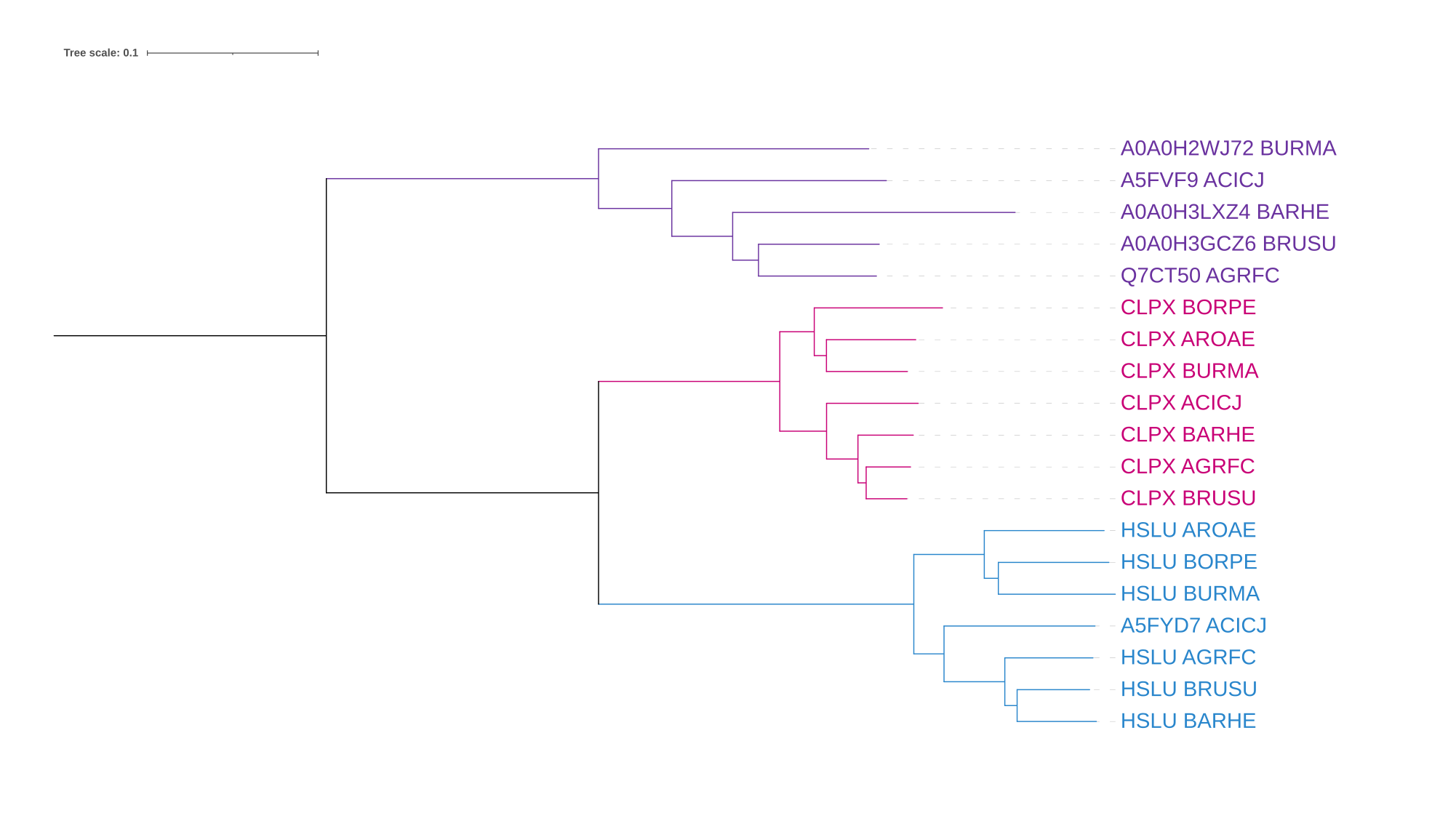

В фиолетовой кладе (ATP-dependent zinc metalloprotease) представлены белки из пяти бактерий, в розовой (ATP-dependent Clp protease (CLPX)) из всех семи бактерий, в голубой (ATP-dependent protease ATPase subunit HslU (HSLU)) также из всех семи бактерий.
Филогения фиолетовой клады: BRUSU сближена с AGRFC, а на общем дереве BRUSU сближена с BARHE.
Филогения розовой клады: BRUSU сближена с AGRFC, а на общем дереве BRUSU сближена с BARHE; BURMA сближена с AROAE, а на общем дереве BURMA сближена с BORPE.
Филогения голубой клады: филогения совпадает с общим деревом.

|
|
| Рис. 1. Выделение цветом разных ортологических групп | Рис. 3. Филогения бактерий |