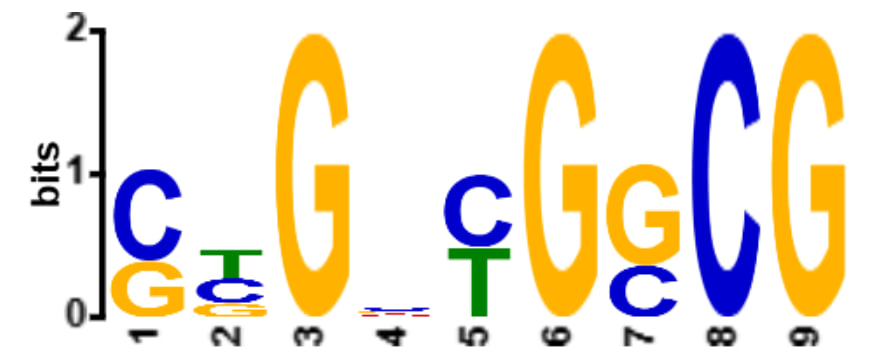
Выбранная в первом семестре бактерия: Dickeya solani. Для поиска последовательности Shine-Dalgarno (сайт посадки рибосомы на мРНК) скачала файл с геномом и файл с его аннотацией. С помощью bedtools вырезала последовательности длиной 100 нуклеотидов, расположенные перед старт-кодонами генов.
Индексирую геном:
samtools faidx GCF_002846995.1_ASM284699v1_genomic.fna
Создаю BED-файл с координатами областей длиной 100 п.н.:
bedtools flank -i GCF_002846995.1_ASM284699v1_genomic.gff -g GCF_002846995.1_ASM284699v1genomic.fna.fai -l 100 -r 0 -s > promoters.bed
-i – входной файл аннотации генов, -g – индексация генома, -l – длина области перед геном, -r – длина области после старт-кодона, -s &ndash учет ориентации гена (+/-)
Извлекаю нуклеотидные последовательности промоторов из генома на основе координат из BED-файла:
bedtools getfasta -fi GCF_002846995.1_ASM284699v1_genomic.fna -bed promoters.bed -fo promoters.fasta
-fi – геном, -bed – координаты промоторных областей, -fo – выходной файл
Также вырезаю последовательности длиной 100 п.н. после старт-кодона – отрицательный контроль (команды аналогичны, меняются только опции:
-l 0 -r 100
Далее провожу поиск мотива с помощью MEME:
meme promoters.fa -o meme_sd_results -dna -mod zoops -minw 5 -maxw 9 -revcomp -evt 0.01
-o – выходная директория, -dna – тип последовательности ДНК, -mod zoops – программа будет искать не более одного вхождения мотива в каждой последовательности, -minw 5 -maxw 9 – длина мотивов, -revcomp – учет ориентации генов, ev 0.01 – порог значимости
Нашлось два мотива (рис. 1, рис. 2). Выдача MEME
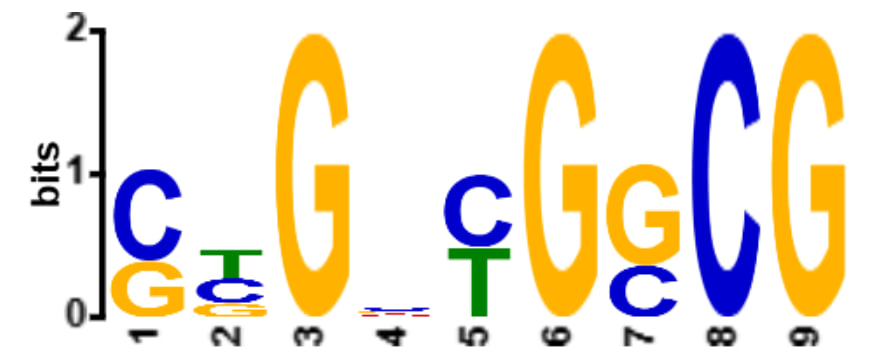

Теперь нужно поискать эти мотивы в промоторных областях и контроле, пример программы для промоторных областей:
fimo meme_sd_results/meme.txt promoters.fa
Выдача FIMO для промоторной области, Выдача FIMO для контроля
В промоторной области 1130 находок мотивов, а в контроле 1545. На странице html вывода FIMO не видно, что второй мотив тоже находится в контроле довольно часто (это посмотрела в файле fimo.tsv).
Вывод: у меня получился тот самый "отрицательный результат", специфичность найденных мотивов в промоторной области не подтвердилась, последовательность SD не найдена.