Краткий обзор вида бактерии Aeromonas allosaccharophila
Подготовил студент ФББ МГУ им. Ломоносова, 2023, Кучевский Максим Владимирович
Аннотация
Данная работа предоставляет информацию по длинам интервалов между последовательностями, кодирующими белки, обеих цепей единственной хромосомы бактерии Aeromonas allosaccharophila. Кроме того, рассматриваются длины интервалов, участвующих в кодировке сразу двух разных белков, для последовательностей обеих цепей хромосомы. Также приведена статистика длин всех белков, кодируемых геномом, который представлен хромосомой и плазмидой.
Введение
Подвижные представители рода Aeromonas являются вездесущими обитателями пресноводных и устьевых сред; эти бактерии являются возбудителями системных и локализованных заболеваний у различных рептилий, рыб, млекопитающих (в том числе и человека); анаэробы[1][2].
Aeromonas allosaccharophila — грамотрицательная, каталаза-положительная бактерия, которая была выделена из больных представителей Anguilla anguilla (европейский угорь) в Валенсии, Испания. Вид Aeromonas allosaccharophila принадлежит домену Bacteria, типу Pseudomonadota, классу Gammaproteobacteria, отряду Aeromonadales, семейству Aeromonadaceae, роду Aeromonas[1].
Материалы и методы
Работа была проведена в “Google Таблицах” со списком кодирующих последовательностей генома Aeromonas allosaccharophila (RefSeq NCBI)(сортировка и отбор последовательностей по тому или иному признаку, построение гистограмм).
Диаграмма длин белков основывается на данных столбца “product_length” (длина продукта)(см. страницы S5, S6, S13).
Диаграммы длин интервалов (которые не кодируют белки) между кодирующими последовательностями и длин попарного пересечения (такая последовательность участвует в кодировании сразу двух белков) кодирующих последовательностей для обеих цепей хромосомы NZ_CP065745.1 (RefSeq NCBI) были получены следующим образом:
- Из исходной таблицы генома (см. таблицу S0) были выбраны только кодирующие белки участки (CDS)(см. таблицу S6).
- Полученная таблица была разделена на две: первая содержит CDS только прямой цепи (+ в столбце “strand”)(см. таблицу S7) хромосомы, а вторая — CDS только обратной цепи (- в столбце “strand”)(см. таблицу S8) хромосомы.
- CDS обеих таблиц были отсортированы по их концам (для удобства). Затем в каждой таблице был добавлен столбец (“intervals”), в котором считается разница между концом гена строкой выше (для первой строки брался конец последнего гена в таблице) и началом гена на текущей строке (см. таблицы S7, S8). Для получения корректного результата в формуле отнимается единица.
- В каждой таблице в столбце “intervals” получались положительные и отрицательные значения. Положительные значения соответствуют длинам промежутков между каждыми двумя ближайшими CDS, а отрицательные — длинам попарных пересечений CDS. Положительные (включая 0) и отрицательные значения были разделены между собой на листы “intervals_+(S9)”/“intervals_-(S10)” и “double_+(S11)”/“double_-(S12)” соответственно (см. страницы S9-S12).
- Используя последние четыре листа со списками длин были построены диаграммы (см. диаграммы S1-S4, S13) длин интервалов между ближайшими CDS и длин попарных пересечений CDS для обеих цепей хромосомы.
Аналогичные диаграммы для плазмиды NZ_CP065746.1 (RefSeq NCBI) не были построены в виду количества CDS на ней — всего 7. Тем не менее, таблицу со значениями длин интервалов и пересечений можно посмотреть в сопроводительных материалах (см. таблицу S14).
Результаты
I. Длины белков бактерии Aeromonas allosaccharophila
На диаграмме (рис. 1) представлена зависимость количества видов белков, синтезируемых данной бактерией, от их длины. Видно, что большинство белков имеет в основном длину от 50 до 500 аминокислотных остатков. Малое количество (21 из 4055 — около 0,5%) белков имеют длину до 50 аминокислотных остатков, значительная часть белков имеет длину больше 500 аминокислотных остатков.
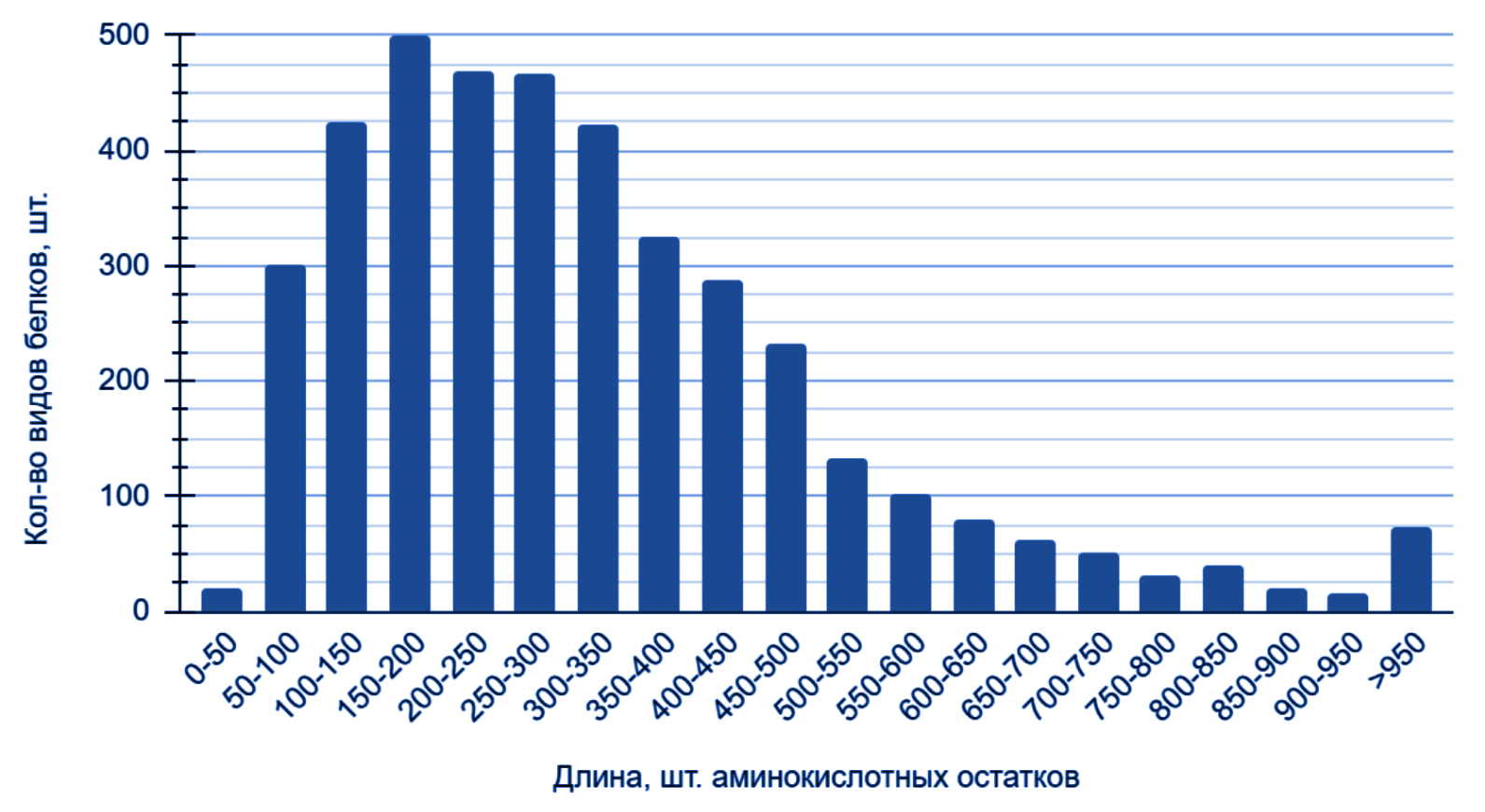
Выводы: интересных особенностей в отношении длин белков выявить не удаётся.
II. Длины интервалов между CDS бактерии Aeromonas allosaccharophila
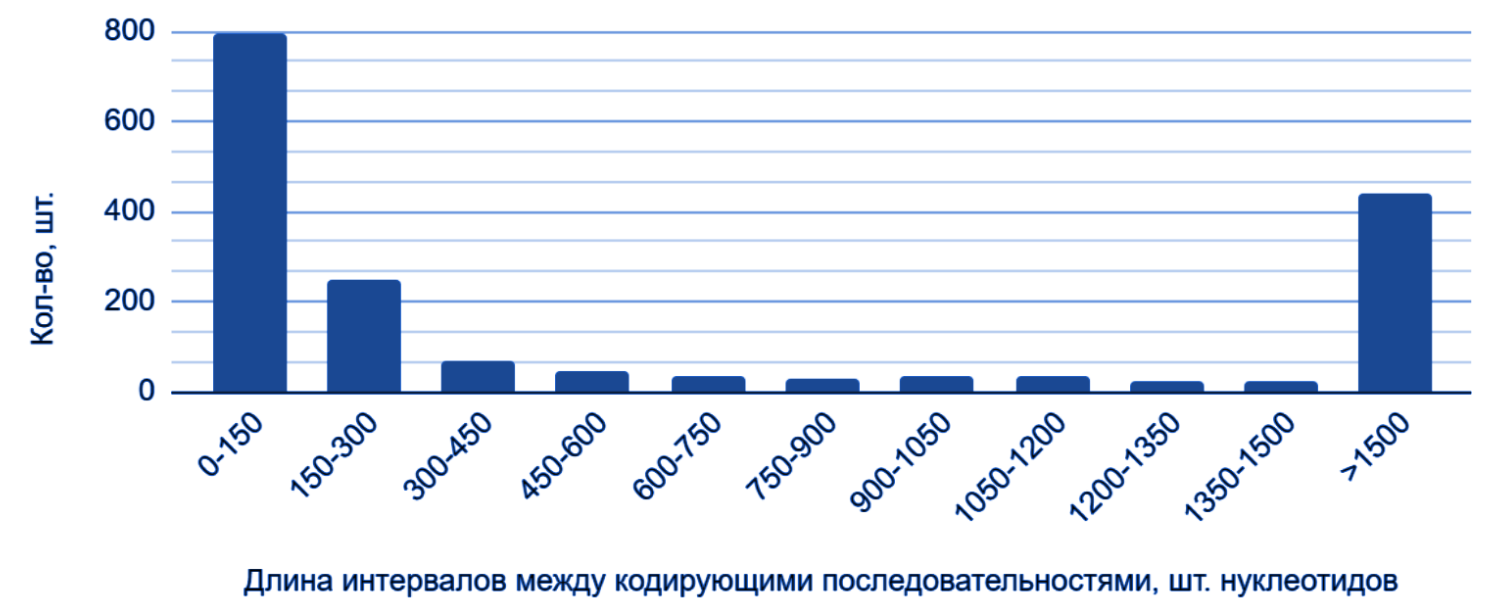
На диаграмме (рис. 2) представлена зависимость количества (не кодирующих белки) интервалов между ближайшими CDS от их длины на прямой (+) цепи хромосомы. Видно, что эти интервалы в основном имеют длину от 0 до 150 нуклеотидов, однако велика доля и интервалов длинной 150-300 нуклеотидов. Интервалы с большей длиной встречаются примерно в одинаково малом количестве.
Аналогичная диаграмма (рис. 3) для обратной (-) цепи хромосомы. Диаграммы (рис. 2 и рис. 3) почти одинаковы.
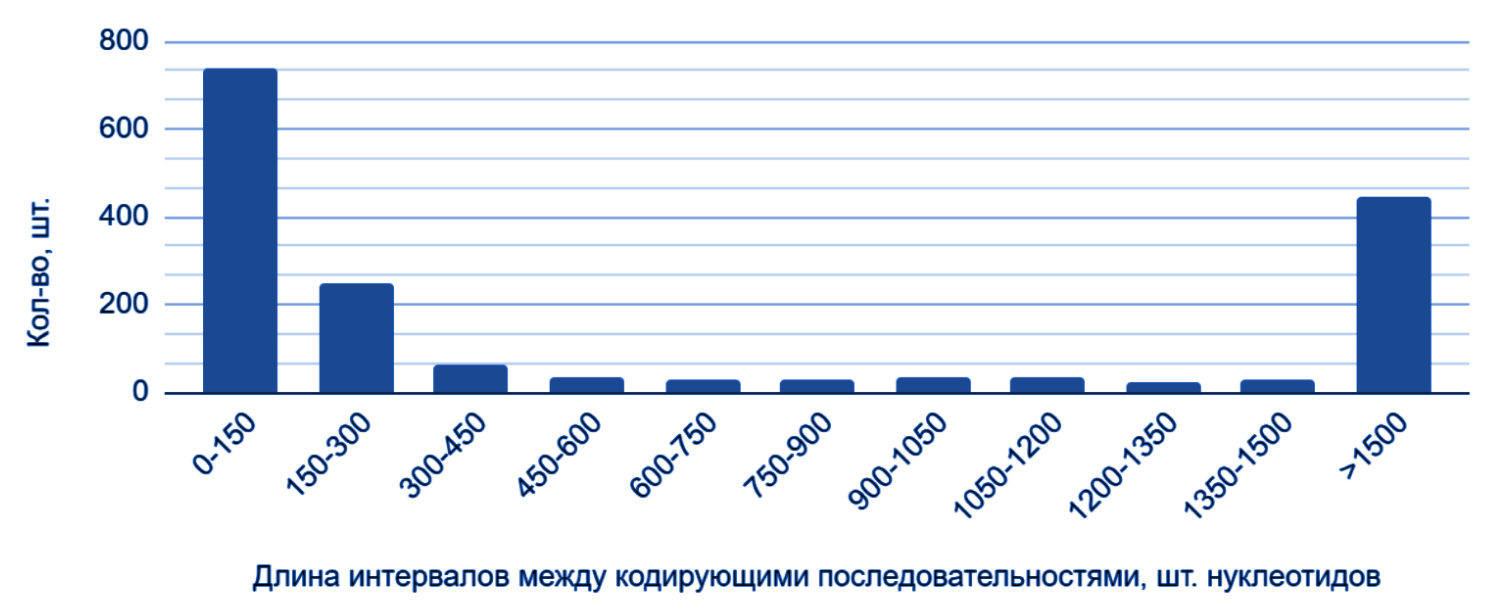
Такое возможно, если для каждого гена одной цепи найдется ген с приблизительно такой же длиной и они будут расположены в цепи аналогичным друг другу образом (или каким-либо похожим способом). Но также нельзя забывать о том, что цепи комплементарны, антипараллельны, а транскрипция идет с 3’ к 5’ концу. Тогда возникает новый вопрос: насколько похожи белки обеих цепей по свойствам и функциям?
Выводы: цепи хромосомы имеют одинаковые зависимости кол-ва интервалов между CDS от их длины [интервалов]. Неплохо бы узнать, как сильно отличаются белки цепей по функциям и свойствам для дальнейших выводов.
III. Длины пересечений CDS бактерии Aeromonas allosaccharophila
Рис. 4 и рис. 5, на которых представлены длины пересечений CDS и их встречаемость, снова показывают сходства двух цепей хромосомы.
В обеих цепях наблюдается отсутствие значений длин, кратных 3, т. е. как если бы пересечения приходились на целые кодоны — рамка считывания первой CDS сохранялась бы и распространялась на вторую CDS. Но в таком случае получался бы лишь 1 белок, а не 2, и из 2 последовательностей — одна кодирующая (которая кодирует белок, как минимум, с двумя остатками метионина) с двумя старт-кодонами и одним стоп-кодоном, а вторая — некодирующая с одним лишь стоп-кодоном на конце.
Невозможность пересечения в 2 нуклеотида легко доказывается. Это пересечение одновременно должно быть ТТ, ТЦ или ЦТ для стоп-кодона (АТТ, АТЦ, АЦТ для ДНК, 3’-5’) первого и ТА для старт-кодона (ТАЦ, для ДНК, 3’-5’) второго.
Аналогично для пересечений длиной 5. Третий нуклеотид одновременно должен быть аденином для стоп-кодона первого и тимином для старт-кодона второго.
По какой причине пересечения длиной 8 встречаются чаще, чем длиной 7, 11 чаще 10 и 14 чаще 13 не могу ответить.
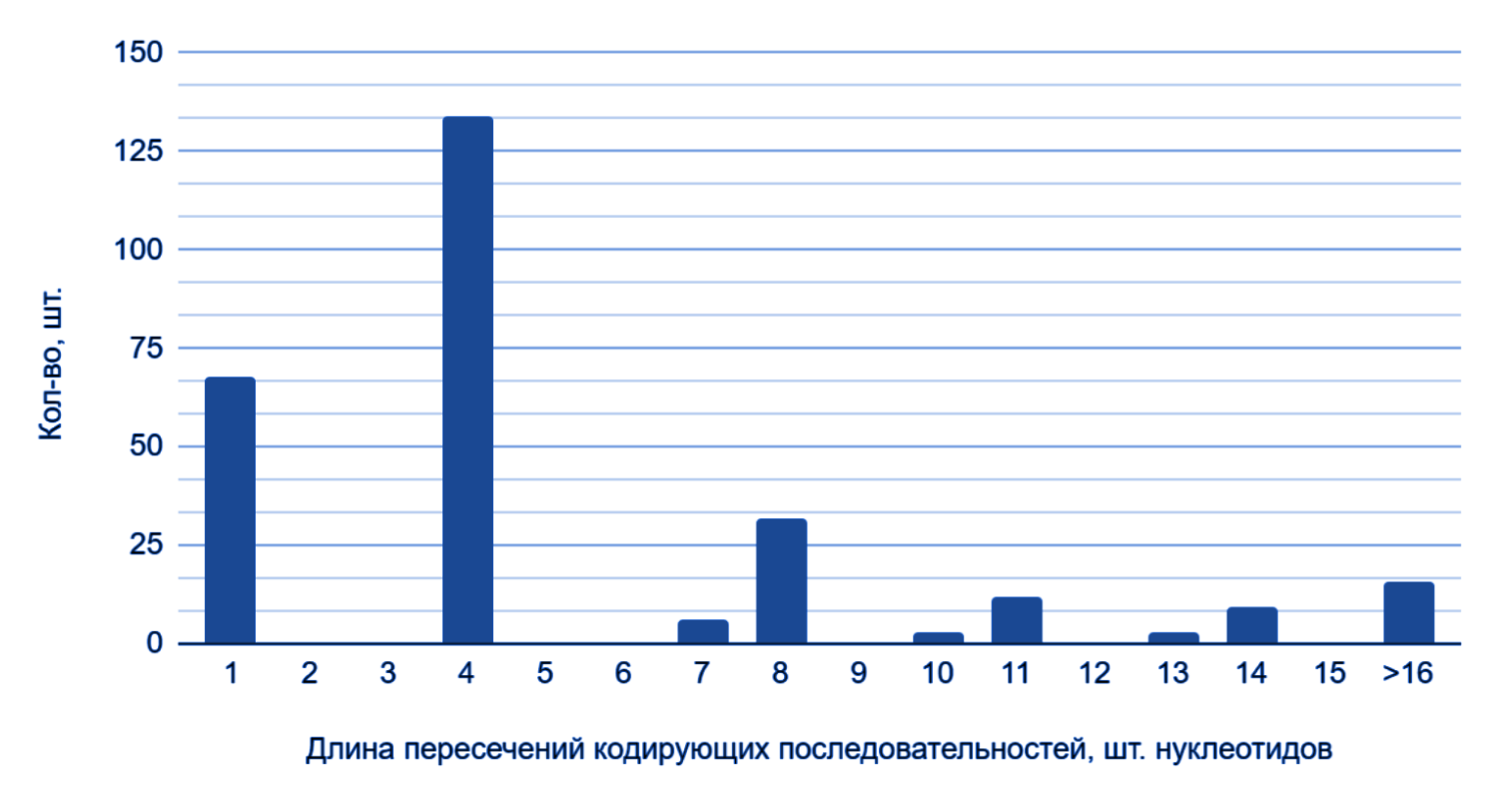
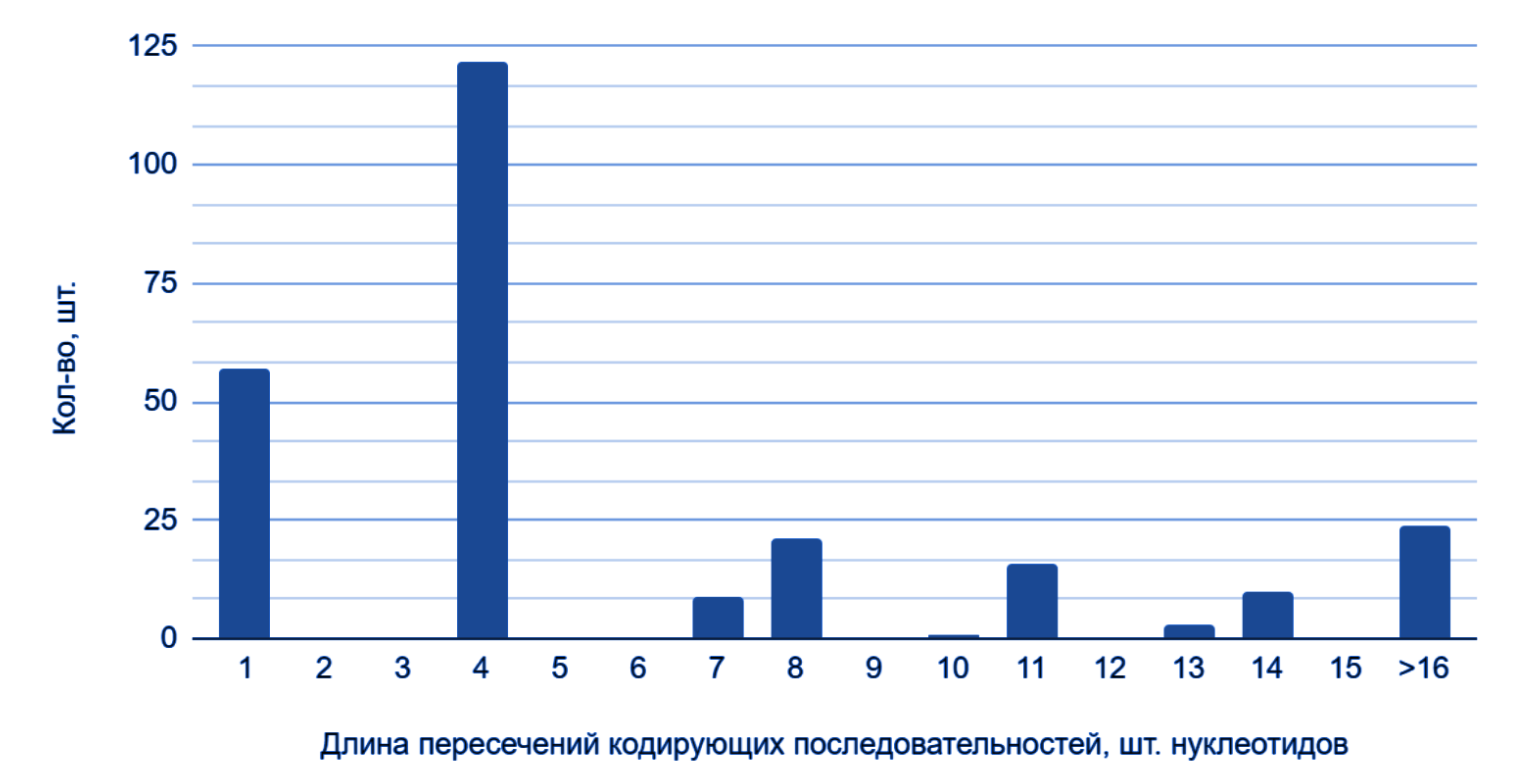
Вывод: обе цепи показывают одинаковую зависимость частоты от длины пересечений. Полученные результаты почти полностью объяснимы. Есть ли закономерность, по которой длина 8 встречается чаще 7, 11 — 10, а 14 — 13, неизвестно.
Сопроводительные материалы
“Google Таблица” с геномом бактерии, промежуточными таблицами и диаграммами:
S0 — feature_tables_unchanged(S0) — таблица с полным геномом бактерии
Обобщённые данные и диаграммы:
S1 — count_intervals_+(S1) — интервалов в ДНК (strand +)
S2 — count_intervals_-(S2) — интервалов в ДНК (strand -)
S3 — count_double_+(S3) — пересечений в ДНК (strand +)
S4 — count_double_-(S4) — пересечений в ДНК (strand -)
S5 — count_len_CDS(S5) — длин белков
Таблицы с CDS:
S6 — proteins_(CDS)(S6) — обеих цепей хромосомы
S7 — CDS_withprotein_chromosome_+(S7) — strand +
S8 — CDS_withprotein_chromosome_-(S8) — strand -
Интервалы:
S9 — intervals_+(S9) — strand +
S10 — intervals_-(S10) — strand -
Пересечения:
S11 — double_+(S11) — strand +
S12 — double_-(S12) — strand -
Прочее:
S13 — all_diagrams(S13) — все диаграммы
S14 — CDS_withproteins_plasmid(S14) — таблица CDS плазмиды
Литература
- A.J. Martinez-Murcia, C. Esteve, E. Garay, M.D. Collins. Aeromonas allosaccharophila sp. nov., a new mesophilic member of the genus Aeromonas (стр. 199). FEMS Microbiology Letters 91 (1992) 199-206 © 1992 Federation of European Microbiological Societies 0378-1097/92
- Brian Austin, Dawn A. Austin (2012). Bacterial Fish Pathogens: Disease of Farmed and Wild Fish (5th ed.). Springer. ISBN 978-9400748835.