Семейство доменов из Pfam
Cytidylyltransferase family
Общее
- Pfam: PF01148 | InterPro: IPR000374 | CTP_transf_1
- seed: 34
- Всего последовательностей: 57k, из них 60 - Swiss-Prot.
- Доменных структур: 143
Таксономическая распространённость
Бактерии составляют 88,81% из всех видов-обладателей белков с данным семейством доменов, эукариоты - 11,05%, археи - 0,14%.
Среди эукариот белки распространены в основном среди представителей Ascomycota, Basidiomycota, Chordata и Streptophyta.
Структура
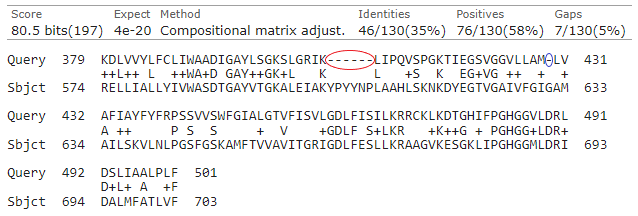
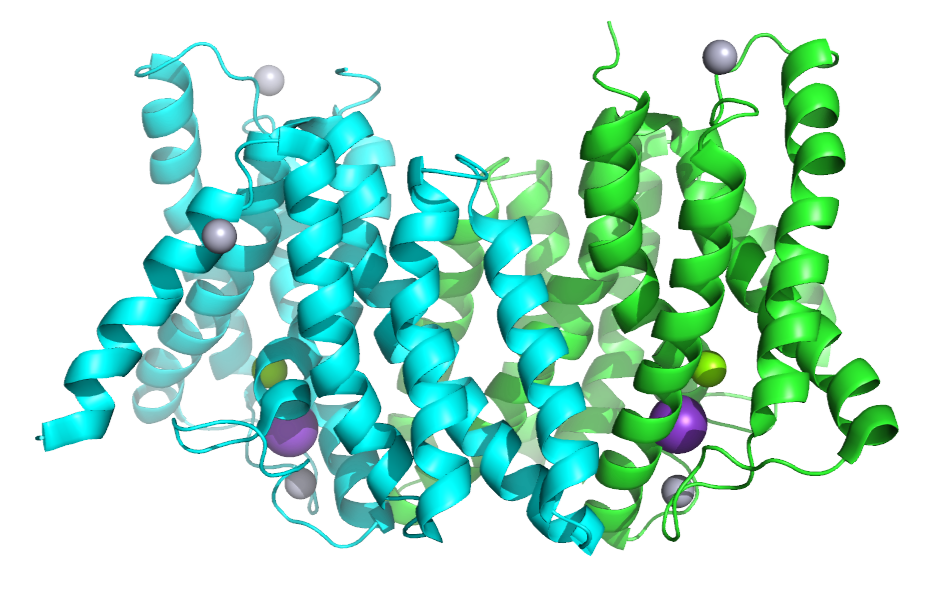
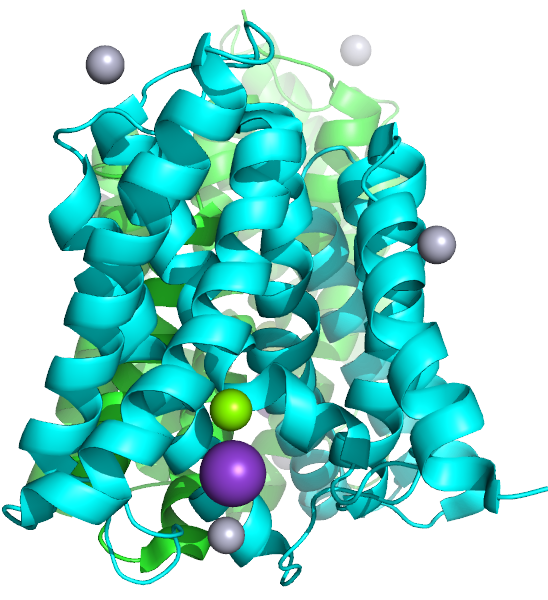
Домены (Рис. 1-4) являются внутримембранными структурами. Два домена разных цепей могут связываться друг с другом.
Домены состоят из α-спиралей. Свзяываются с двумя ионами двухвалентной ртути (обозначены серым цветом) и ионом магния (зеленый). В активной форме домен дополнительно связан с ещё одним ионом ртути и ионом калия (фиолетовый). В неактивной форме домен связан с нонил бета-D-глюкопиранозидом (простой эфир нонола и глюкозы) (Рис. 1 - зеленый; рис. 3 - желтый).
Функции
Членами этого семейства являются цитидилилтрансферазы интегральных мембранных белков. Семейство включает фосфатидатцитидилилтрансферазу EC: 2.7.7.41, а также Sec59 из дрожжей (долихолкиназа - EC: 2.7.1.108).

О выравнивании seed
- Проект Jalview. Два окна - со всеми последовательностями и мною избранными из всех.
- Максимальные достоверные блоки для всех последовательностей - 44 (единственная абсолютно консервативная аминокислота в округе), 198-205 и 221-228 (между ними индели во всех последовательностях, кроме одной), 322-353.
- Для рассмотрения достоверных блоков, включающих не все последовательности, выбрал следующие последовательности, сильно выделяющиеся среди остальных - CDS2_HUMAN/92-91, CDSA_CAEEL/118-117 (нематода), CDS_DROME/106-105 (дрозофила), CDS1_SOLTU/72-71 (картофель), CDS3_ARATH/119-118 (арабидопсис), CDS1_YEAST/85-84, CDSH_SCHPO/75-74 (дрожжи), Q8ILZ6_PLAF7/284-283 (малярийный плазмодий).

- Данные последовательности принадлежат эукариотическим организмам; остальные последовательности принадлежат бактериям. Максимальные достоверные блоки - 3-21, 44-59, 71 (единственная абсолютно консервативная аминокислота в округе), 139-209, 217-256, 307-369 (визуально самый консервативный блок)
- Блок, не имеющих достоверных подблоков - 49-57.
- Вывод - выравнивание отражает гомологию среди всех последовательностей главным образом в середине и конце представленных последовательностей. Наблюдается наибольшее сходство последовательностей среди рассмотренных эукариот: животных, дрожжей, высших растений и малярийного плазмодия.
Архитектуры
Выбраны архитектуры PF01255-PF01148 и PF00102-PF01148 (рис.5) с 13 и 9 белками соответственно. Использованы все белки данных архитектур. Проект Jalview c этими 22 белками.
Общие максимальные достоверные блоки - 249, 864-871, 890.
Максимальные достоверные блоки для PF01255-PF01148 архитектуры - 210-213, 248-261, 315, 349-354, 408-418, 446-469, 479-485, 864-875, 890-891.
Для PF00102-PF01148 архитектуры - 186-190, 205-207, 249-251, 259, 299-301, 307-323, 337-361, 424, 431, 537-562, 583-591, 633-642, 657-674, 691-737, 750-755, 759-778, 784-785, 792-799, 816-819, 832-898, 912-921, 929-931, 957-960, 1006-1019, 1048-1054, 1098-1105, 1167-1172, 1211-1217, 1301-1303, 1353-1361.

Сравнение доменов PF01148 выбранных архитектур
Ссылка на проект Jalview. Все выравнивания в ходе работы проводились с помощью алгоритма MSAprobs. Достоверные блоки выделил в группы в проекте (блоки обведены в рамки).
Для первой архитектуры достоверных блоков получилось мало. Домен PF01148 для архитектуры PF01255-PF01148 достаточно вариабельный, чего не скажешь в случае второй архитектуры.
Для второй архитектуры нашлось очень много достоверных блоков, они покрывают большую часть последовательностей архитектуры PF00102-PF01148.
Уровень сходства кажется не очень большим. Белки первой архитектуры принадлежат различным группам бактерий (9/13 белков), пауку-ткачу (2/13), мухе цеце и остракоде (пресноводное ракообразное). Белки второй архитектуры принадлежат буквально одному роду фитофторы (7/9 белков) и одному виду пероноспоровых оомицет (2/9).
Таким образом, сравнивая один и тот же домен двух архитектур, было получено выравнивание, в котором заметно отделяются друг от друга несколько видов животных, паразитических псевдогрибов и бактерий.
Dot plot
Диаграмма dot plot в BLASTp при вводе белков W0BDF7 (query) и H3GIC0 (subject) архитектур PF01255-PF01148 и PF00102-PF01148 соответственно - рис. 6.
На диаграмме видны два разрыва, которые соответствуют инделям внутри W0BDF7 (рис. 7).