Алгоритмы и программы множественного выравнивания
Сравнение алгоритмов множественного выравнивания
Выбор последовательностей и алгоритмов
Для выполнения задания выбрал 7 последовательностей деоксирибонуклеаз-2-α:
- Человека - O00115 (Swiss-Prot)
- Домашнего быка - P56541 (S-P)
- Домашней свиньи - O62855 (S-P)
- Серой крысы - Q9QZK8 (S-P)
- Домовой мыши - P56542 (S-P)
- Американского лобстера - A0A8J5MWC5 (TrEMBL)
- Pseudolycoriella hygida (двукрылое насекомое, сциарида) - A0A9Q0S3Z0 (TrEMBL)
и 3 алгоритма выравнивания:
- MSAprobs - в качестве референсного, т.к. выравнивания данного алгоритма гораздо ближе к эволюционному.
- MAFFT - как компромисс качества и скорости.
- MUSCLE - самый быстрый алгоритм с самыми далёкими от эволюционного выравниваниями из выбранных алгоритмов (предварительные оценки произведены согласно обзору из презентации, слайд №12).
Одинаково выравненные участки
Проект Jalview. 4 окна - без выравнивания и выравненные алгоритмами MSAprobs, MAFFT, MUSCLE.
В начале и конце 1-3 последовательности в участках без крупных инделей выравниваются с длинными инделями и малым количеством кусочков других последовательностей. Поэтому случайно совпавшие одиночные колонки в начале и конце не учитываются
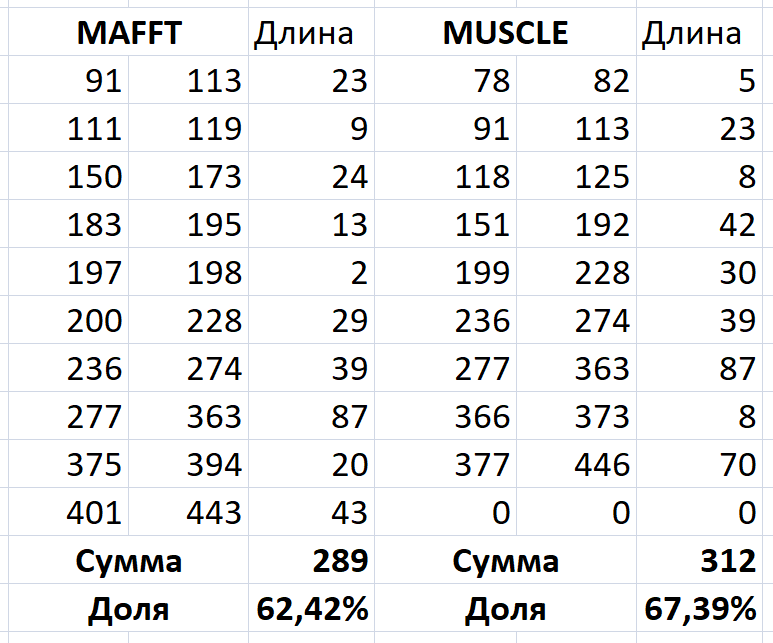
MSAprobs и MAFFT: 91-113=90-112; 111-119=115-123; 150-173=149-172; 183-195=184-196; 197-198=198-199; 200-228=201-229; 236-274=237-275; 277-363=278-364; 375-394=376-395; 401-443=402-444
MSAprobs и Muscle: 78-82=79-83; 91-113=92-114; 118-125=119-126; 151-192=155-196; 199-228=203-232; 236-274=240-278; 277-363=281-367; 366-373=370-377; 377-446=381-450
Комментарии к табл. 1 и выводы
Общая длина значимых совпадающих участков для алгоритмов MSAProbs и MAFFT - 289 п.н., для MSAProbs и MUSCLE - 312. Длина вырванивания MSAProbs - 463 п. н.
Отношение общей длины значимых совпадающих участков к длине выравнивания MSAProbs составили примерно 62% и 67% для алгоритмов MAFFT и MUSCLE соответственно.
Вопреки ожиданиям, в данном опыте выравнивание алгоритма MUSCLE оказалось ближе к референсному от MSAProbs, чем от MAFFT.
Вероятно, что такой результат был получен из-за странного набора организмов, выбранных для опыта. Кроме того, посл-ти членистоногих принадлежат базе данных TrEMBL и выводятся из гомологии, в то время как у млекопитающих посл-ти имеют неоспоримые доказательства существования (белок или транскрипт гена) и принадлежат базе Swiss-Prot.
Сравнение алгоритмов выравнивания PDB и MSAProbs
Пояснения к проекту Jalview
"Original" - попарные выравнивания (объединённые в одно окно для удобства дальнейшей работы) с сайта PDB референсного A0A011NU96 (далее для краткости - NU96) с A0A011MFQ1 (MFQ1) и с A0A009S065 (S065).
"MV_final" (moved version, final) - выравнивание, выполненное вручную добавлением инделей. Алгоритм: передвигаем NU96 нижнего выравнивания так, чтобы начало совпадало с началом NU96 первого выравнивания, передвигаем на то же число гэпов и MFQ1 нижнего выравнивания; добавляем индель сначала для NU96 во втором выравнивании с MFQ1 в том месте, где он стоят в NU96 первого выравнивания с S065, добавляем этот же индель в аналогичном месте для MFQ1. Последнюю операцию с добавлением инделей повторяем, пока не дойдём до конца. Когда все индели были внесены в последовательности второго выравнивания, для удобства можно убрать второй NU96. Полученный результат можно наблюдать в окошке "MV_final". Полученный результат достаточно близок к выдаче MAFFT или MSAprobs.
"MAFFT" - множественное локальное выравнивание NU96, S065 и MFQ1 с помощью алгоритма MAFFT.
"MSAprobs" - то же самое, но с алгоритмом MSAprobs.
Комментарии к проекту:
Легко заметить, что все три выравнивания (с сайта PDB, MAFFT и MSAprobs) очень похожи: различия наблюдаются только в начале, где приходится выравнивать последовательность почти без гэпов с обрывками второй последовательности и рядом гэпов для самой короткой из них, и в последних нескольких позициях. Маловероятно, что выравнивания в началах имеют биологический смысл, зато все три выравнивания показывают явную гомологию для большого блока от примерно середины до конца выравнивания.
О белках и организмах
(к сожалению, о функциях непосредственно белков информации недостаточно):- A0A011NU96 (взят за референсный) - белок споры бактерии Accumulibacter regalis (организм, принадлежащий группе полифосфатаккумулирующих организмов; представляет интерес в области очистки сточных вод)
- A0A011MFQ1 - содержащий SHSP-домен белок бактерии Candidatus Accumulibacter adjunctus (ещё один организм, принадлежащий группе полифосфатаккумулирующих организмов)
- A0A009S065 - белок семейства Hsp20-α кристаллинов бактерии Acinetobacter baumannii 99063 (патоген, вызывающий ряд заболеваний у людей с ослабленным иммунитетом; также известен как Iraqibacter)
На картинке №1 можно увидеть собственно попарные пространственные выравнивания A0A011NU96 (референсный) с A0A011MFQ1 и A0A009S065.
У первых двух организмов из таблицы №2 белки имеют больше сходств, чем первый и последний.
1-2: относительно небольшое отклонение RMSD в 1.33, немалое значение TM-score в 0.67, идентичность на 84% при выравнивании 98 а. о. второго белка с первым - всё указывает на гомологию как минимум на доменном уровне.
О паре 1-3 трудно сказть то же самое. Достаточно большое отклонение RMSD (3.12), значение идентичности всего 24%, а из 189 а. о. выравнены 102 - признаки гомологии (только) одного домена.
Небольшой обзор на программу множественного выравнивания MSAProbs
MSAProbs - алгоритм выравнивания нескольких последовательностей для белков. Принцип работы MSAProbs основан на комбинации парных скрытых марковских моделей и функций разбиения для вычисления апостериорных вероятностей. При оценке с использованием популярных тестов: BAliBASE, PREFAB, SABmark и OXBENCH, MSAProbs обеспечивает статистически значимое повышение точности по сравнению с существующими высокопроизводительными элайнерами, включая ClustalW, MAFFT, MUSCLE, ProbCons и Probalign. Кроме того, MSAProbs оптимизирован для процессоров с общей памятью за счет использования многопоточной конструкции и дополнительно распараллелен для систем с распределенной памятью, использующих MPI, чтобы преодолеть высокий барьер накладных расходов на память и достичь хорошей параллельности и масштабируемости по размеру данных.