Выравнивание геномов ВИЧ-1 и ВИЧ-2
Геном ВИЧ-1 - NC_001802
Геном ВИЧ-2 - NC_001722
Для задания выбрал два генома вирусов иммунодефицита человека 1 и 2. Произвёл выравнивание последовательностей MegaBLAST (рис.1), disdiscontiguous MegaBLAST (рис.2), BLASTn (рис.3) и tBLASTx (рис.4).
MegaBLAST
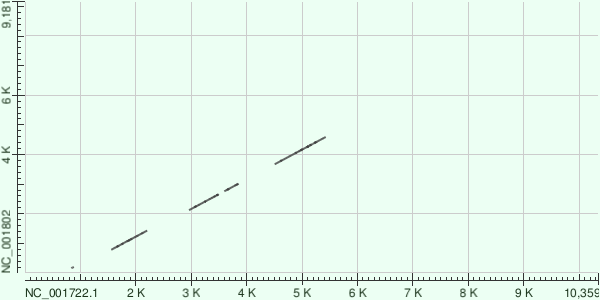

MegaBLAST со стандартными настройками ничего не находил, поэтому пришлось снизить длину слова до 16 (рис.1). На карте локального сходства видны 5 отрезков. При посмотре генома ВИЧ-1 на сайте NBCI выяснилось, что программой MegaBLAST выровнялись некоторые некодирующие области в начале и участки гена, который кодирует gag-pol (NCBI). Gag-pol белок-предшественник, из которого (после работы протеазы) получаются, например: из gag - нуклеопротеин (компактизация геномной РНК в вирусе), белки капсида и матрикса; pol - обратная транскриптаза (синтезирует вДНК на матрице геномной РНК), интеграза (встраивает вДНК в геном клетки хозяина) и протеаза (щепит белки-предшественники на функциональные).[1]
MegaBLAST нашел сходства в гене, кодирующем целое мн-во белков, но не обнаружил сходства между генами оболочки (env) и ряда вспомогательных белков.
dMegaBLAST дал результат несколько лучше. Он нашёл больше сходств между gag-pol ВИЧ-1 и -2 (рис.2).
BLASTn
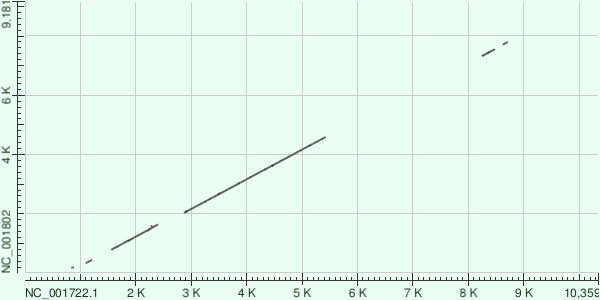
BLASTn обнаружил короткий отрезок (в промежутке 7-8 kbp по оси ординат) (рис.3), который соответствует генам env и asp (NCBI). У ретровирусов не является редкостью, когда один и тот же участок принадлежит сразу двум генам; рамки считывания у них несовпадают.[1]
tBLASTx
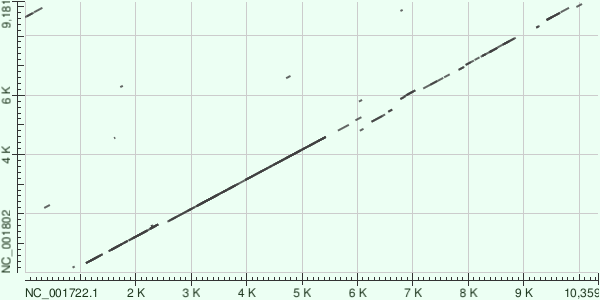
tBLASTx, наконец, находит практически все сходства данных геномов (рис.4), т.е. к предыдущему результату добавляются (почти все оставшиеся) гены остальных (вспомогательных, которые сильно отличаются между родами ретровирусов [1]) белков (и прочие учатски, пока не берусь уточнять).
Таким образом, tBLASTx дал наиболее информативный результат, в то время как MegaBLAST (в любом его обличии) из-за высокой длины слова находит только часть генов основных ретровирусных белков (gag и pol), а BLASTn хоть и лучше проявил себя, чем MegaBLAST, всё равно не нашел целого ряда сходств между мелкими генами вспомогательных белков.
Литература
[1] - статья by Stephen P. Goff в PDF формате (в том виде, в котором я получил её от своих научных руководителей; в будущем постараюсь найти онлайн версию этой статьи)