Подготовка данных
Избавимся от остатков адаптеров:
TrimmomaticSE SRR4240380.fastq.gz trim.out ILLUMINACLIP:adapters.fasta:2:7:7 2> err_trim.1.txt
Посчитаем число чтений. Для этого достаточно разделить число строк файла в формате fastqc на 4.
zcat SRR4240380.fastq.gz | wc -l # архивированный файл имеет меньшее число строк, чем разархивированная версия.
20869272
wc -l trim.out
20476576
Число чтений до удаления адаптеров - 5,217,318, после - 5,119,144. Т. о. около 1.88% чтений оказались остатками адаптеров.
Удалим с правых концов низкокачественные (<20) нуклеотиды и оставим чтения с длиной не менее 32 нуклеотид.
TrimmomaticSE SRR4240380.fastq.gz trim.2.out TRAILING:20 MINLEN:32 2> err_trim.2.txt
wc -l trim.2.out
19866668 trim.2.out
4,966,667 чтений стало. 152,477 чтений было удалено.
Файлы весят в разархивированном виде: до обработки - 525.3 Мб, после удаления адаптеров - 515.2 Мб, после последней обработки - 499.3 Мб.
Velvet
velveth seq 31 -short -fastq.gz trim.2.out 2>err_v.txt
kmvmax@kodomo:/mnt/scratch/NGS/kmvmax/pr14$ velvetg seq 2>err_vg.txt
...
Final graph has 392 nodes and n50 of 12382, max 25915, total 660240, using 0/4966667 reads
В выдаче команды velvetg есть значение N50 - 12382.
Запустим пайплайн для нахождения самых длинных контигов:
kmvmax@kodomo:/mnt/scratch/NGS/kmvmax/pr14$ grep '>' seq/contigs.fa | tr '_' '\t' | sort -k 4 -nr | head -3
>NODE 3 length 25915 cov 27.422071
>NODE 20 length 23850 cov 24.777359
>NODE 22 length 23807 cov 25.742975
Посчитаем, сколько всего было получено контигов. Зная эту информацию, легко найдем медианное значение в отсортированном по столбцу покрытий и потом пронумерованном списке контигов.
kmvmax@kodomo:/mnt/scratch/NGS/kmvmax/pr14$ grep '>' seq/contigs.fa | wc -l
163
kmvmax@kodomo:/mnt/scratch/NGS/kmvmax/pr14$ grep '>' seq/contigs.fa | tr '_' '\t' | sort -k 6 -nr | nl | grep ' 82'
82 >NODE 86 length 2452 cov 22.223898
Медианное покрытие 22.223898. Под определение аномально малых и больших покрытий попадают значения менее ~4,4446 и более ~111,115 соответственно.
kmvmax@kodomo:/mnt/scratch/NGS/kmvmax/pr14$ grep '>' seq/contigs.fa | tr '_' '\t' | sort -k 6 -nr | nl | head -3
1 >NODE 55 length 934 cov 130.479660
2 >NODE 11 length 2106 cov 126.008545 # нижняя граница аномально больших покрытий
3 >NODE 73 length 501 cov 86.676643
kmvmax@kodomo:/mnt/scratch/NGS/kmvmax/pr14$ grep '>' seq/contigs.fa | tr '_' '\t' | sort -k 6 -nr | nl | tail -12
152 >NODE 186 length 35 cov 4.571429
153 >NODE 252 length 62 cov 4.403226 # верхняя граница аномально маленьких покрытий
...
162 >NODE 106 length 80 cov 2.700000
163 >NODE 225 length 62 cov 2.419355
Под определение аномальных попадает 13 контигов. Самое большое покрытие наблюдается у контига с длиной 934 под номером 55, самое маленькое - длиной в 62 и под номером 225.
Анализ megablast
Образец пайплайна для получения последовательностей fasta самых больших контигов. x=⌈(y/60)+1⌉ (в каждой строке последовательности в fasta формате - 60 букв => y/60; заголовок тоже занимает строку => +1; т.к. строки бывают неполные, округляем полученное значение вверх)
grep -A x '>NODE_?_length_y' seq/contigs.fa > big?.out
Результаты
>NODE_3_length_25915_cov_27.422071
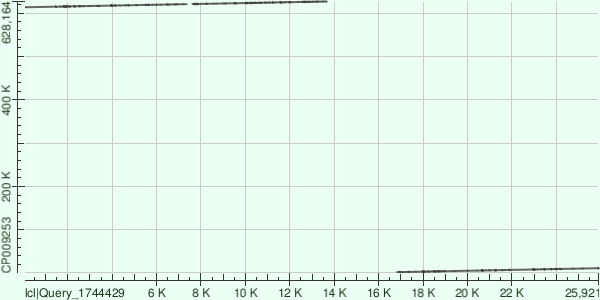
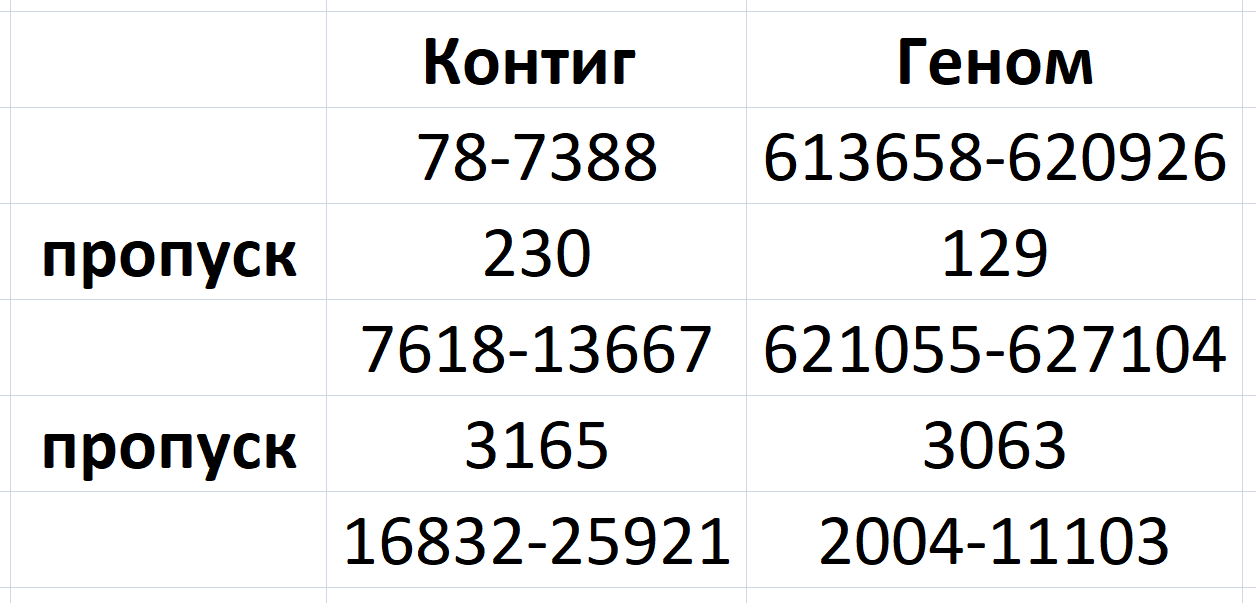
Пропуск между левым и правым выравниваниями около 3 тыс. п.н. (длина контига всего 25 тыс.)
Положение линий указывает, что одно из выравниваний расщепилось на два в силу линейности в ген. банке изначально кольцевой хромосомы.
Длина число пропущенный нуклеотид контига около 3400 п.н.
>NODE_20_length_23850_cov_24.777359
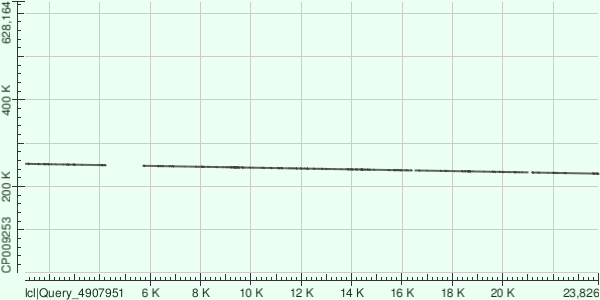
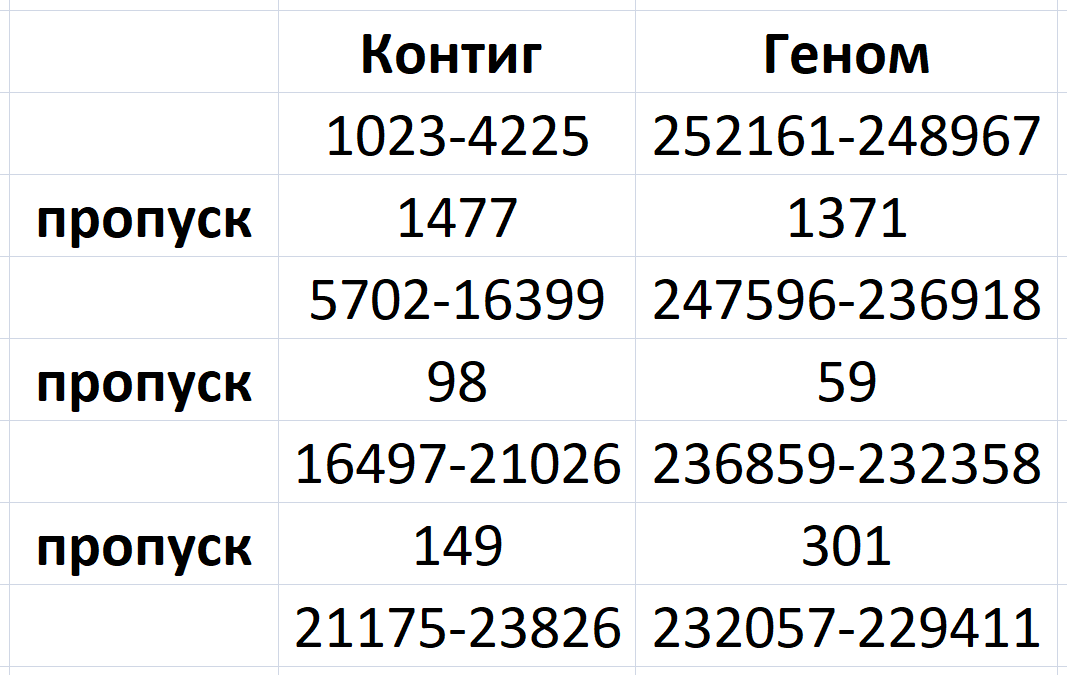
Все находки выравнялись +/- (контиг/геном). Длина пропущенных н.п. контига уже менее 1800 п.н.
>NODE_22_length_23807_cov_25.742975
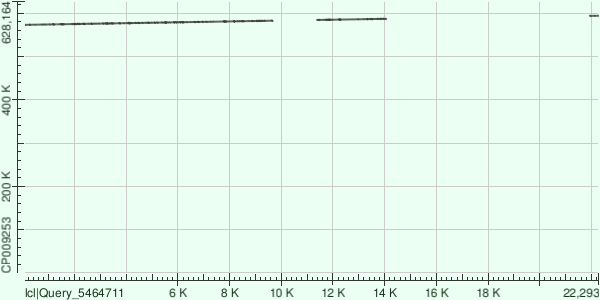
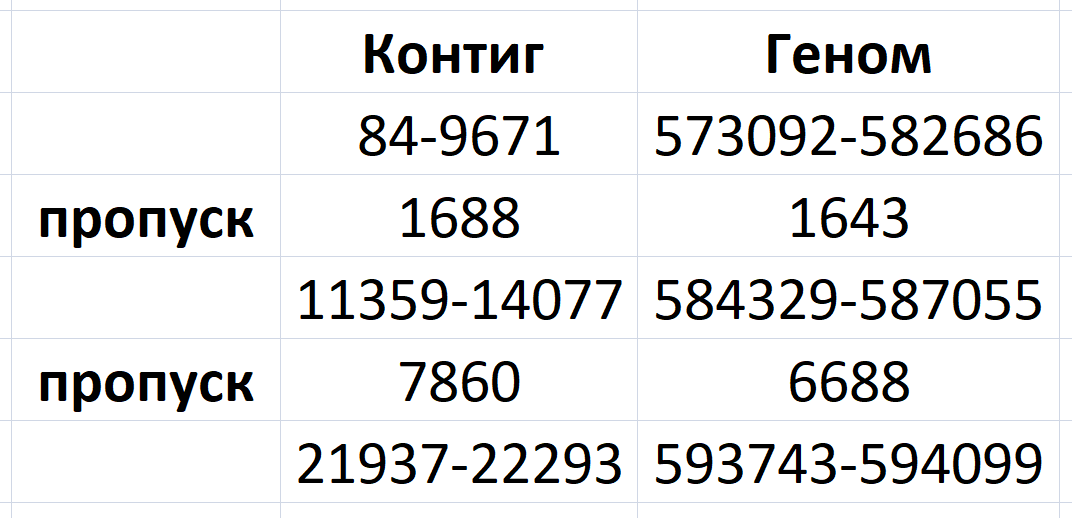
Большие пропуски. Правое выравнивание маленькое и находится на большом расстоянии от предыдущего - выглядит подозрительно.
Версии использованных программ