Мотивы в белках, паттерны и PSSM для их поиска
Консервативные мотивы в выравнивании
Выбран домен PF00161 или RIP - rest in peace ribosome inactivating protein. RIPs относятся к бактериальным и растительным токсинам, которые нарушают работу рибосом эукариотической клетки. RIPs представляют интерес как иммунотоксин в связке с моноклональными телами при лечении рака. Также один из представителей RIP обладает мощной активностью против заражённых ВИЧ-1 макрофагов и Т-клеток.
Информация взята из записи по PF00161.
Последовательностей выравнивания seed - 32. Белков, проверенных кураторами UniProtKB - 69.
Выравнивание (seed) доступно по ссылке.
Также в описании домена было указано, что рядом находящиеся остатки глутамата и аргинина непосредственно участвуют в катализе депуринизации аденина (4324) в 28S рРНК.
При окрашивании выравнивания seed методом Clustal с порогом идентичности 75% были найдены высоко консервативные остатки глутамата (97% - 31/32 совпадений) и аргинина (94% - 30/32 совпадений), находящиеся в seed в положениях 227 и 230 соответственно. По этим двум остаткам можно выделить мотив EAARF (консенсус для второго и последнего остатков - 78%; третий остаток - 41% - не окрасился), который принимает непосредственное участие в катализе данным доменом.
Произвел поиск по паттерну. Получил 21 последовательность во всём выравнивании. Все найденные по паттерну посл-ти принадлежат одному и тому же участку выравнивания - [227,231]
Произвёл поиск с помощью PROSITE - получил 266 совпадений в 265 последовательностях. Произвести выравнивание по всем последовательностям не удастся, поэтому придется от части данных отказаться. Отобрал рандомные 25 последовательностей с помощью скрипта. Последовательности, с которыми я буду работать - 25_fasta.txt
Произвел выравнивание с помощью программы muscle, получил следующее выравнивание.
Произвёл поиск по паттерну в полученном выравнивании. Получил 25 находок (в каждой посл-ти по одному). Выравненные друг с другом паттерны можно наблюдать в позициях выравнивания [1463, 1470] (5 белков), [2230, 2236] (9 белков), [3251, 3255] (3 белка). Остальные 8 найденных мотивов выравнялись отдельно друг от друга.
Наиболее интересный результат был получен для позиций [2230, 2236], где выравнялось 9 мотивов. Возможно, именно здесь находятся активные центры данных белков.
Мотив в кладе филогенетического дерева
Newick формула полученного дерева; на рис. 1 - изображение этого дерева.

Для посл-тей, выделенных зелёным цветом (судя по мнемоникам, это ветвь с бактериями) на рис. 1 нашёл наиболее консервативный и крупный мотив: LREQQEAER [591, 599].
Провёл поиск мотива LREQQEAER во всех 25 выравненных посл-тях: было найдено только 5 подпосл-тей + 2 очень близких (LREQQDAER и LREQAEAAR) в одних и тех же колоннах [1458, 1469]. Данный мотив характерен для выделенной клады из бактерий (CHLAA, KLEP7, SALTI, E. coli).
PSI-BLAST
Выбран O05886 - Ribosome hibernation promotion factor - небольшой (219 а. о.) белок бактерии Mycobacterium tuberculosis, функция которого заключается в димеризации активных 70S рибосом в 100S димеры неактивных рибосом (проще говоря, белок выключает рибосомы). Локализуется в цитоплазме
Таблица итераций. Результат не стабилизировался - с каждой итерацией белков выше порога становилось всё больше и больше. Считаю, что наилучший результат был достигнут после второй интерации - когда не было находок выше порога (> 0.005), получилось 27 находок, худшая из которых имела E-value порядком -11. В первой и во всех итерациях после второй шаг между худшим ниже порога (< 0.005) и лучшим выше порога (> 0.005) составлял в среднем 0.00336.
Поиск мотивов de novo
Взял все 69 белков из БД SwissProt в формате FASTA, в которых представлен домен PF00161.
Запустим поиск мотивов - будем искать 5 мотивов с длиной от 5 до 15 аминокислот:
Наиболее значимой (E-value = 2.3e-571) из пяти находок оказался мотив активного центра домена с рассматривавейся ранее подпосл-тью EAARF (которая содержит каталитически активные остатки глутамата и аргинина) (рис. 2); мотив представлен в 68 последовательностях из 69.

Наименее значимый мотив из найденных обладает значением E-value равным 5.1e-255 - данный мотив представлен на рисунке 3. Данный мотив привлёк моё внимание, т. к. содержит 3 высококонсервативные и информативные остатки аминокислот в положениях 1, 4 и 9 - "Y..F....R" (рис. 3).
Возможно, данный мотив участвует в связывании (за счёт полярности аргинина и/или стекинговых взаимодействий тирозина и фенилаланина с субстратом). Любопытно также, что в позициях 5, 8 и 12 выделяются остатки аллифатических аминокислот. Мотив наблюдался в 68 посл-тях из 69.

Представленность сайта GATC
Сгенерировал с помощью программы перестановки из G, A, T, C.
Выдача программы cbcalc.
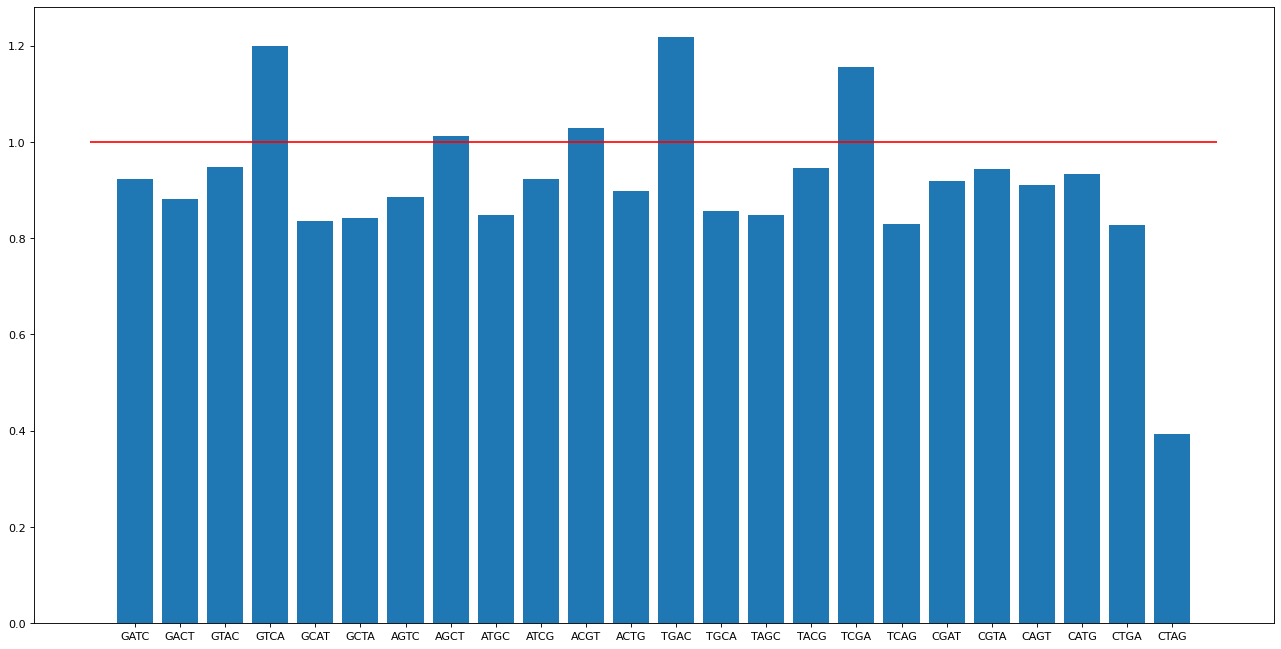
В моей бактерии (Aeromonas allosacharophila) (рис. 4) метилирование больше представлено для сайтов GTCA, AGCT, ACGT, TGAC и TCGA. Судя по графику, сайт GATC играет не слишком важную роль, т. к. уровень его метилирования меньше ожидаемого.