Т. к. далеко не у каждого вида, выбранного ранее, есть полностью отсеквенированный митохондриальный геном, пришлось подбирать другие близкородственные виды. Ко мнемоникам замещённых животных добавил знак "*", чтобы обозначить, что данный вид замещён близкородственным (в виду отсутствия полного митохондриального генома).
В качестве внешней группы выбрал представителя Лепидозавров - геккона (Thirakhupt's bent-toed gecko).
| Вид | Мнемоника | Таксономия |
| Francolinus pintadeanus (замещает Francolinus francolinus) | FRAFR* | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Sauropsida; Sauria; Archelosauria; Archosauria; Dinosauria; Saurischia; Theropoda; Coelurosauria; Aves; Neognathae; Galloanserae; Galliformes; Phasianidae; Perdicinae |
| Falco sparverius (замещает Falco femoralis) | FALFE* | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Sauropsida; Sauria; Archelosauria; Archosauria; Dinosauria; Saurischia; Theropoda; Coelurosauria; Aves; Neognathae; Neoaves; Telluraves; Australaves; Falconiformes; Falconidae |
| Aptenodytes forsteri (замещает Aptenodytes patagonicus) | APTPA* | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Sauropsida; Sauria; Archelosauria; Archosauria; Dinosauria; Saurischia; Theropoda; Coelurosauria; Aves; Neognathae; Neoaves; Aequornithes; Sphenisciformes; Spheniscidae |
| Oceanites oceanicus (замещает Fregetta tropica) | FRETR* | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Sauropsida; Sauria; Archelosauria; Archosauria; Dinosauria; Saurischia; Theropoda; Coelurosauria; Aves; Neognathae; Neoaves; Aequornithes; Procellariiformes; Hydrobatidae |
| Cyrtodactylus thirakhupti (внешняя группа) | CYRTH | Eumetazoa; Bilateria; Deuterostomia; Chordata; Craniata; Vertebrata; Gnathostomata; Teleostomi; Euteleostomi; Sarcopterygii; Dipnotetrapodomorpha; Tetrapoda; Amniota; Sauropsida; Sauria; Lepidosauria; Squamata; Bifurcata; Gekkota; Gekkonidae; Gekkoninae; Cyrtodactylus |
Реконструкция дерева по нуклеотидным последовательностям
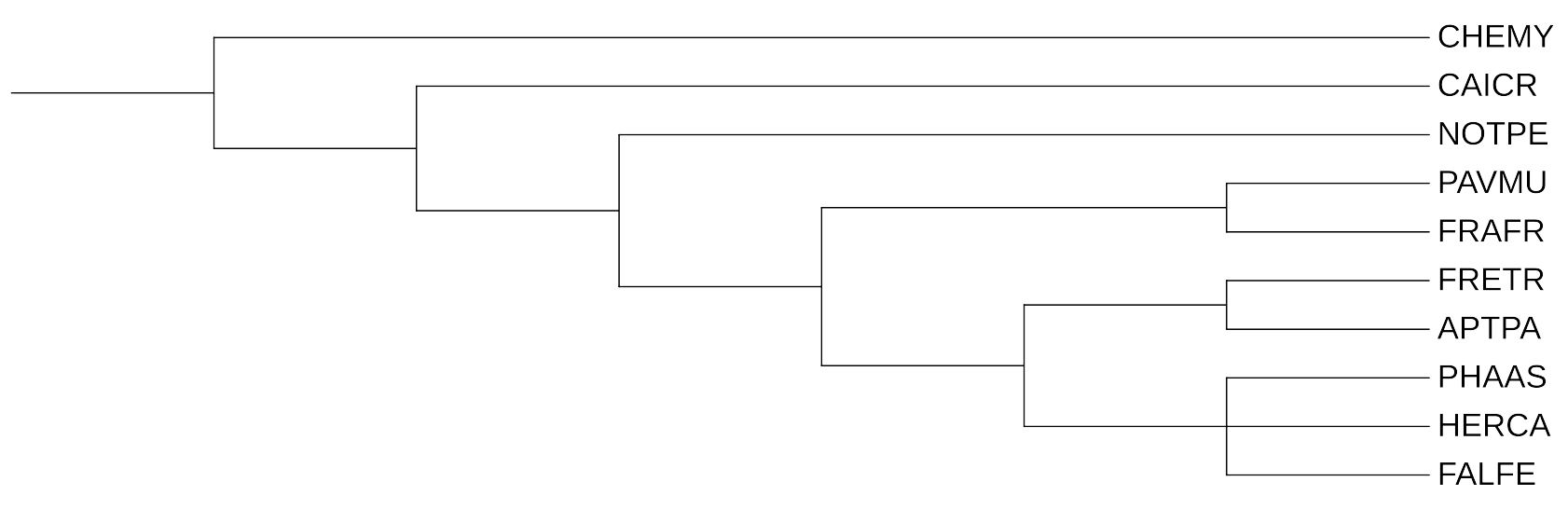

В филогенетическом дереве (рис.2), построенном с помощью fastme по последовательностям гена 12S рРНК митохондрии, правильно (в сравнении с референсным деревом - рис. 1) разделены CHEMY (черепаха) и клада Архозавров; Архозавры правильно разделены на CAICR (крокодил) и птиц (Dinosauria; Saurischia; Theropoda; Coelurosauria; Aves); птицы правильно разделены на NOTPE (бескилевая птица) и Новонёбные; в свою очередь Новонёбные правильно разделились на Фазановых (Galloanserae; Galliformes; Phasianidae - PAVMU и FRAFR*) и Neoaves.
На этом совпадения нового дерева с референсным заканчиваются. Oceanites oceanicus (замещающая Fregetta tropica) - FRETR* - и Aptenodytes forsteri (замещает Aptenodytes patagonicus) - APTPA* - правильно сгруппированы в кладу Aequornithes, однако эта группа неправильно расположена внутри клады Telluraves (т. е. вместе с HERCA). Две другие "теллуроптицы" правильно объединены вместе, но находится в соседстве с неправильно образованной группой HERCA-FRETR*-APTPA*.
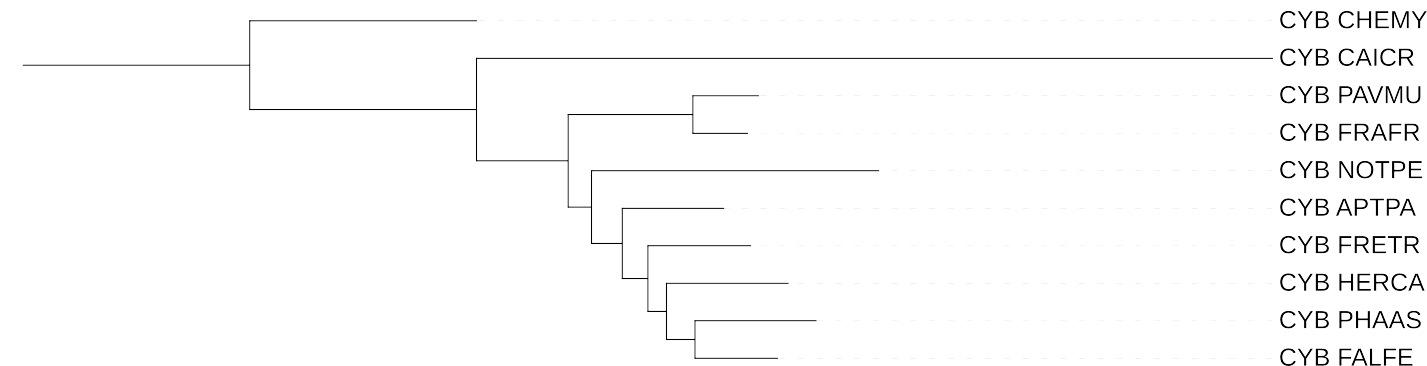
Сравним деревья (рис. 2 и рис. 3), построенные по разным последовательностям, но одной программой (fastme). Одинаково распределены CHEMY, CAICR. Птицы разделились по-разному - по 12S rRNA они разделились (как и полагается) на Бескилевых (NOTPE) и Новонёбных; в дереве с делением по цитохрому Б птицы сначала делятся Galloanserae (с лат. буквально "курогуси" - группа внутри Новонебных) и сборную группу из Бескилевых и Новонёбных. По 12S rRNA FRETR* и APTPA* верно сгруппированы в кладу Aequornithes, но неверно положение внутри клады Telluraves в соседстве с HERCA; по цитохрому аналогичные FRETR и APTPA не принадлежат одной кладе и более-менее правильно объединенные "теллуроптицы" находятся внутри Aequornithes.
Сравним деревья (рис. 2 и рис. 4), построенные разными программами и по разными последовательностям. Обе программы и по обоим последовательностям отличают крокодила, черепаху и птиц (одинаково распределены CHEMY, CAICR). iqtree по цитохрому также справилась с делением птиц хуже, чем fastme по 12S rRNA. fastme по 12S rRNA правильно поделила птиц на Бескилевых и Новонёбных, а iqtree по цитохрому (также, как и fastme по цитохрому) разделила птиц на "курогусей" и сборную группу. iqtree и со второй попытки не отличила бескилевую NOTPE, вместо этого разделив APTPE от остальных; fastme относительно успешно обособил Aequornithes. С третьей попытки iqtree, наконец, разделил остатки Новонёбных и одну Бескилевую птицу, разделив Новонёбных на Aequornithes в лице FRETR и "теллуроптиц"; fastme по 12S rRNA не справился с последними птицами, разделив "теллуроптиц" на две клады и расположив водных внутри одной из групп "теллуроптиц".
Выводы
fastme по 12S rRNA построил наилучшее дерево, перепутав только положение Aequornithes (FRETR* и APTPA*) относительно Telluraves.
fastme плохо справился с построением дерева по цитохрому Б: неправильно разделены Беескилевые и Новонёбные и ошибочно отделена Galloanserae от остальных Новонёбных; Aequornithes ошибочно принята за родительскую кладу для Telluraves и представители Aequornithes не объединены вместе.
iqtree по цитохрому Б построил самое далекое от референса дерево: iqtree повторил ошибку с ранним отделением "курогусей"; только после пингвина APTPA (APTPA оказалась совсем далеко от родственной FRETR) Бескилевая NOTPE отделилась от Новонёбных. Т. е. программа плохо отличила между собой примитивных и современных птиц, выделила "курогусей" (Galloanserae) отдельной кладой от остальных птиц и не обнаружила родства между представителями Aequornithes.
Укоренение во внешнюю группу

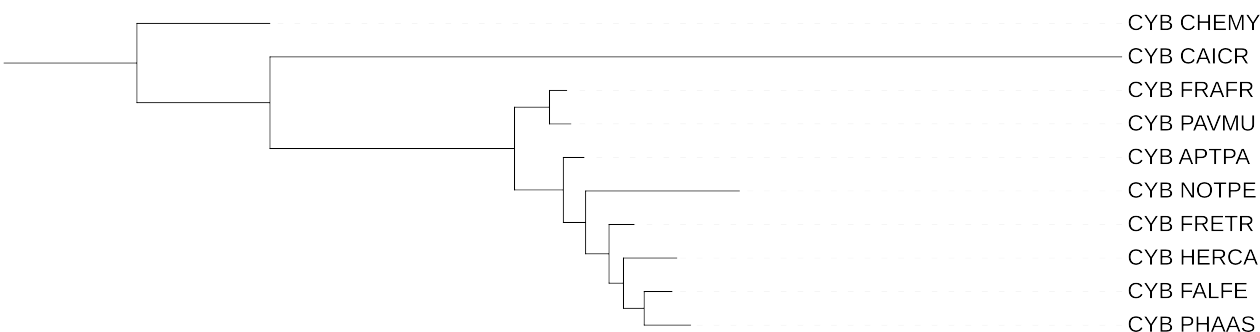
Использование геккона (рис. 5) в качестве внешней группы никак не изменило положение других животных и клад в дереве (рис. 2). Скорее всего, черепаха CHEMY в предыдущем построении дерева послужила внешней группой.
Бутстреп

В дереве (рис. 6) для черепахи и крокодила не подписаны числа поддержки - их положения гарантированны программой.
Программа подвергает сомнению птичью ветвь - только в 67 случаях из 100 Бескилевые правильно отделяются от Новонёбных. На предыдущих деревьях можно убедиться, что данная ветвь действительно проблемная для обеих программ.
Правильное разделение Новонебных птиц на Galloanserae и Neoaves встречается лишь в 52 репликах из 100. При выравнивании по цитохрому и fastme, и iqtree испытывали сложности при распределении "курогусей" - они неправильно распределяли Galloanserae вдали от других птиц.
В 91 из 100 реплик правильно установлен состав Galloanserae. С составом "курогусей" при построении дерева по цитохрому ни одна программа не ошибалась.
По какой-то причине ошибочная ветвь, разделяющаяя две "теллуроптицы" и "теллуроптицу" с водными, встречалась 87 раз в 100 репликах. Любопытно, что при выравнивании по цитохрому программы не разделяли на две части Telluraves и решали трихотомию, объединяя PHAAS и FALFE в подкладу. fastme при выравнивании по 12S rRNA не справился в этом моменте и при этом "считает" (судя по значению поддержки), что данной ветви можно доверять.
Значение поддержки для объедиения FALFE* и PHAAS - 92. Такое объединение действительно верно - обе птицы принадлежат кладе Telluraves.
47 для объединения HERCA (Telluraves) и Aequornithes. Значение поддержки подсказывает нам, что этой ветви доверять не стоит, хотя в действительности Telluraves и Aequornithes - сестринские клады.
Значение поддежрки для ветви Aequornithes (на мой взгляд) маловато - только 72, хоть FRETR* и APTPA* принадлежат обе Aequornithes
На этом дереве есть неправильно реконструированная ветвь и при этом высоко оценённая значением поддержки - это ветвь, которая разделяет (FALFE*, PHAAS) и (HERCA, (FRETR*, APTPA*)).
Вывод - бутстреп не всегда даёт правильную оценку достоверности ветви и может низко оценивать правильные ветви. Значения поддержки не гарантируют верность или неверность той или иной ветви.