Четвертый практикум
Я выбрал следующие белки: CLPX_THIDA, CLPX_AROAE, CLPX_BURMA, CLPX_AGRFC, CLPX_BARHE, CLPX_HAEIN, CLPX_ACICJ, CLPX_NEIMA.
Далее я собрал их в один файл, сделал среду для blastp, и воспользовался blastp, получив следующие результаты:
sp|Q3SI99|CLPX_THIDA ATP-dependent Clp protease ATP-binding subun... 642 0.0
sp|Q5P160|CLPX_AROAE ATP-dependent Clp protease ATP-binding subun... 619 0.0
sp|Q62JK8|CLPX_BURMA ATP-dependent Clp protease ATP-binding subun... 617 0.0
sp|Q8UFY5|CLPX_AGRFC ATP-dependent Clp protease ATP-binding subun... 596 0.0
sp|Q6G3Z2|CLPX_BARHE ATP-dependent Clp protease ATP-binding subun... 588 0.0
sp|P44838|CLPX_HAEIN ATP-dependent Clp protease ATP-binding subun... 587 0.0
sp|A5FX05|CLPX_ACICJ ATP-dependent Clp protease ATP-binding subun... 583 0.0
sp|Q9JTX8|CLPX_NEIMA ATP-dependent Clp protease ATP-binding subun... 557 0.0
sp|Q6G5G0|HSLU_BARHE ATP-dependent protease ATPase subunit HslU O... 97.1 4e-22
sp|Q5P503|HSLU_AROAE ATP-dependent protease ATPase subunit HslU O... 93.6 8e-21
sp|Q8UJ87|HSLU_AGRFC ATP-dependent protease ATPase subunit HslU O... 92.0 2e-20
tr|A5FYD7|A5FYD7_ACICJ ATP-dependent protease ATPase subunit HslU... 92.0 2e-20
sp|P43773|HSLU_HAEIN ATP-dependent protease ATPase subunit HslU O... 92.0 2e-20
tr|Q3SFW1|Q3SFW1_THIDA ATP-dependent protease ATPase subunit HslU... 86.7 2e-18
sp|Q62F00|HSLU_BURMA ATP-dependent protease ATPase subunit HslU O... 82.4 4e-17
tr|A0A0H3LXZ4|A0A0H3LXZ4_BARHE ATP-dependent zinc metalloprotease... 45.4 3e-05
tr|Q7CT50|Q7CT50_AGRFC ATP-dependent zinc metalloprotease FtsH OS... 45.4 4e-05
tr|A5FVF9|A5FVF9_ACICJ ATP-dependent zinc metalloprotease FtsH OS... 44.7 6e-05
tr|A0A0H2WJ72|A0A0H2WJ72_BURMA ATP-dependent zinc metalloprotease... 43.9 9e-05
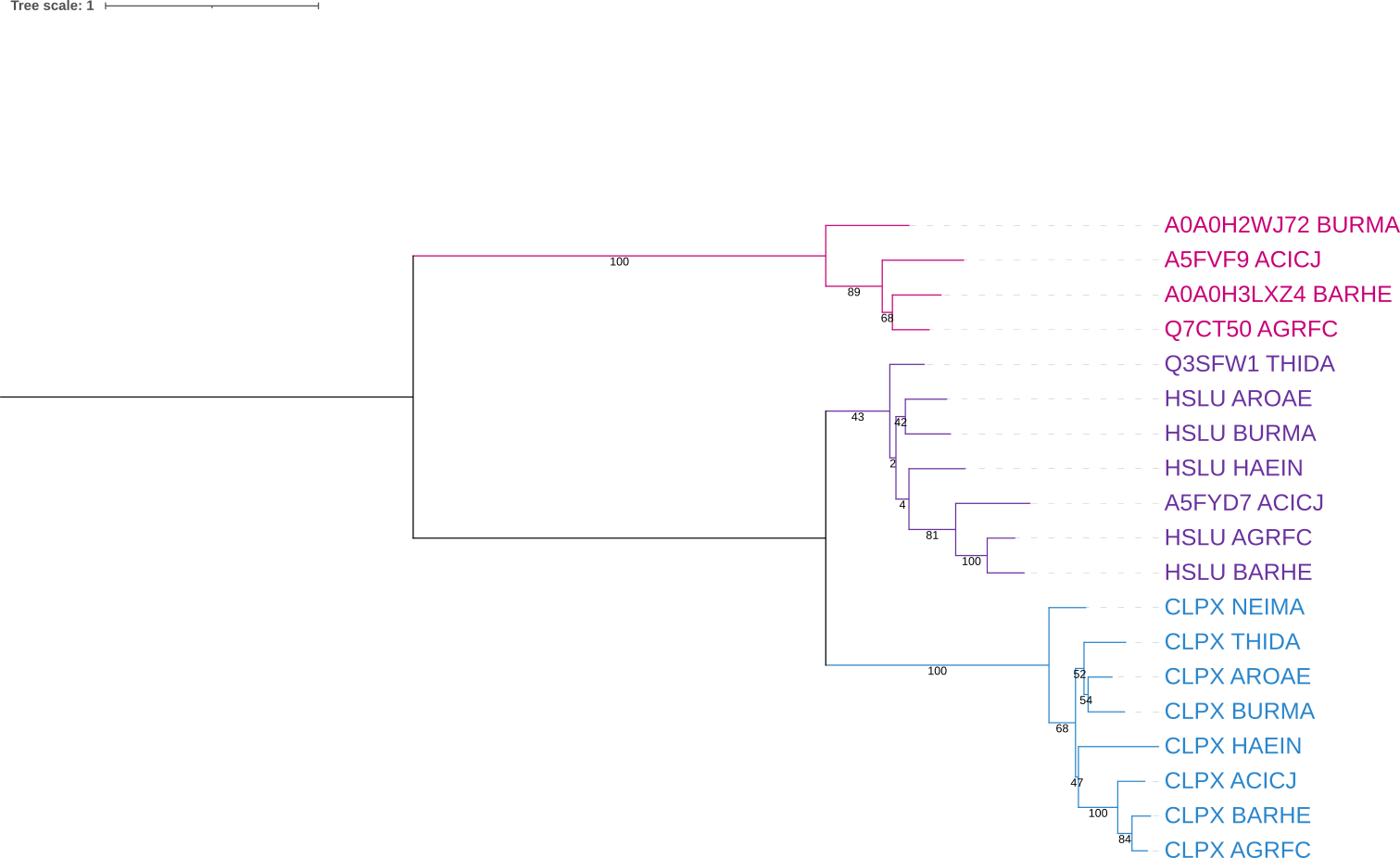
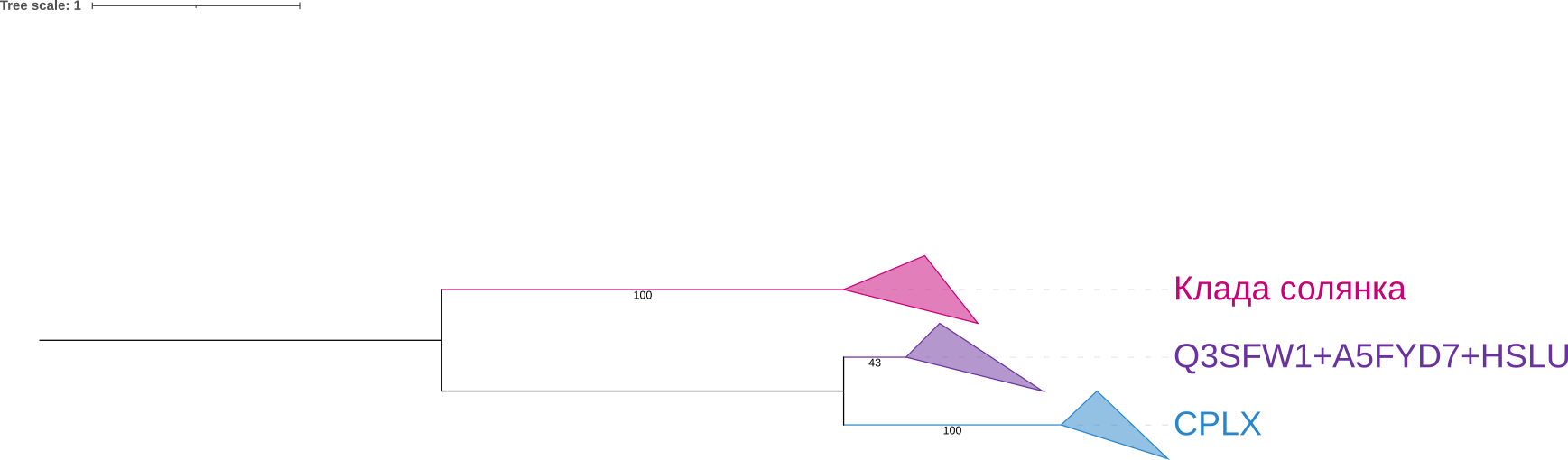
ФайликДалее я выровнял последовательности с помощью muscle, перевел в формат phylyp и подал в fastme, используя алгоритм LG (я воспользовался сайтом, ибо на кодомо мне выдавало ошибку о несоответствии длинн в выравнивании)
Сначала семье CPLX: ACICJ, BARHE и AHRFC совпадают с филогенией самих бактерий, AROAE и THIDA должны быть в одной кладе, но вместо этого AROAE ближе к BURMA. К кладе AROAE, THIDA и BURMA должна быть ближе NEIMA, но вместо этого ближе к кладе ACICJ, BARHE и AHRFC.
Для семьи HSLU: единтсвенное отличие от изначального дерева - HAEIN должно быть ближе к кладе AROAE, BURMA. Для солянки: реконструированно верно.
Белки в солнке являются белком АТФ-зависимой цинковой металлопротеазой FtsH.
Парологи: HSLU_BURMA и CPLX_BURMA, HSLU_AGRFC и CPLX_AGRFC и HSLU_HAEIN и CLPX_HAEIN
Парологи: HSLU_AGRFC и HSLU_BURMA, HSLU_BARHE и HSLU_HAEIN и CLPX_ACICJ и CLPX_NEIMA