Девятый практикум
Моя бактерия с первого семестра: Nitratireductor kimnyeongensis. GFF файл и FASTA файл
Сначала я воспользовался программой operon mapper, далее подал выход на код старшекурсника, получив тренировочную выборку, тестовую выборку и негативный контроль.
Код берет 100 нуклеотидов до оперона (промотр), и дальше сортирует все находки по группам.
Дальше я подал все на MEME, используя следующую команду:
meme term4v2-pr9-housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50
-dna показывает, что на вход дана ДНК, -nmotifs 3 ищет 3 мотива, -minw и -maxw ограничивают их минимальную и максимальную длинну.

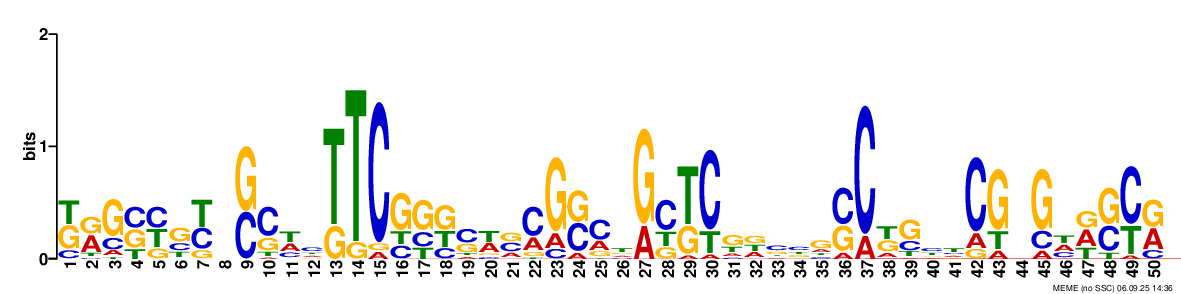

Я выбрал мотиф ATATTDGCATACDVDMTGTATGTCAAKWTATATA как самый короткий с самым маленьким pvalue. В итоге выход был подан на fimo
fimo --o term4v2-pr9-fimo-test --motif ATATTDGCATACDVDMTGTATGTCAAKWTATATA --thresh 0.05 meme_out/meme.txt term4v2-pr9-promotors.fasta
fimo --o term4v2-pr9-fimo-control --motif ATATTDGCATACDVDMTGTATGTCAAKWTATATA --thresh 0.05 meme_out/meme.txt term4v2-pr9-negative.fasta
Как видно, в контроле гораздо меньше (тестовая - 10572, контроль - 551), то есть последовательность и в правду чаще встречается в промоторе, чем в случайной части генома.