Мини-обзор генома и протеома бактерии Blattabacterium clevelandi
Кудрин Константин Александрович
Факультет Биоинженерии и Биоинформатики, Московский Государственный университет, Москва, Россия.
Контактные данные: kkudrin2006@yandex.ru
Аннотация
В настоящей работе было проведено исследование генома и протеома бактерии Blattabacterium clevelandi посредством функций электронных таблиц и языка программирования Python.
Ключевые слова: Blattabacterium clevelandi, мини-обзор.
Введение
| Домен | Bacteria |
|---|---|
| Тип | Bacteroidota |
| Класс | Flavobacteriia |
| Отряд | Flavobacteriales |
| Семейство | Blattabacteriaceae |
| Род | Blattabacterium |
| Вид | Blattabacterium clevelandi |
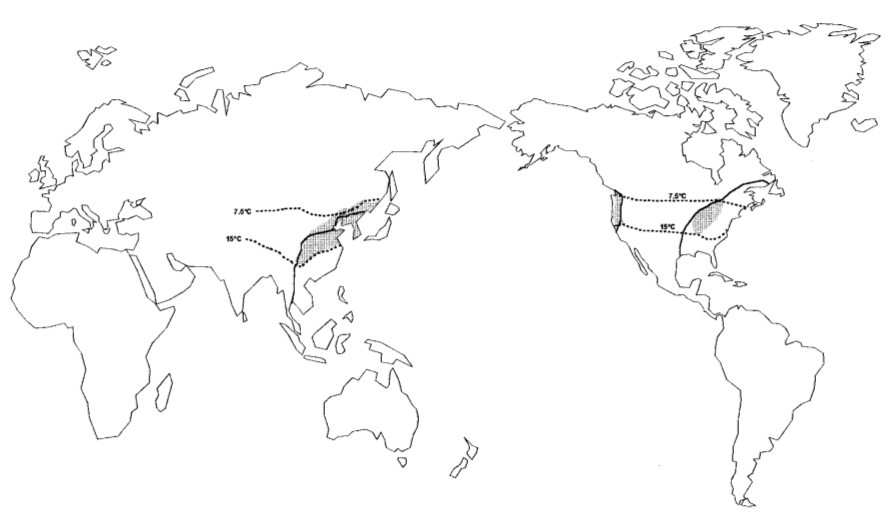
Blattabacterium clevelandi — грамотрицательные, слегка изогнутые и прямые палочки, являющиеся внутриклеточными симбионтами тараканов вида Cryptocercus cleveland, обитающих на территории, отмеченной на рисунке 1. Эти эндосимбиотические бактерии обнаруживаются преимущественно в специализированных клетках жировых тел насекомых. Они играют роль в превращении метаболитов мочевой кислоты в полезные азотистые соединения в анаэробных условиях. Эндосимбионты передаются вертикально от матери к потомству. Они обладают сильно редуцированными геномами, размер генома которых оценивается примерно в 638 килобаз. Было предложено выделить эту бактерию в отдельный вид в 2003 году. Расположение бактерии в систематике представлено в таблице 1 [1][2]. В данной работе исследуется геном и протеом бактерии Blattabacterium clevelandi: была составлена гистограмма длин белков, гистограмма GC% по CDS, и многое другое.
Материалы и методы
Для изучения генома и протеома бактерии мы использовали таблицы feature table и CDS(приведённые в сопроводительных материалах), которые мы анализировали с использованием функционала гугл таблиц и языка программирования Python.
3.1 Использование гугл таблиц
При помощи гугл таблиц были составлены гистограммы и диаграммы, приведённые в работе. Также таблицы были использованы для изоляции необходимых данных и подсчёта искомых элементов(использовались такие функции как max,min,countifs,sum,median,).
3.2 Использование Python
При помощи Python были подсчитаны количества нуклеотидов и разных старт/стоп кодонов.
Результаты
4.1 Гистограмма длин белков
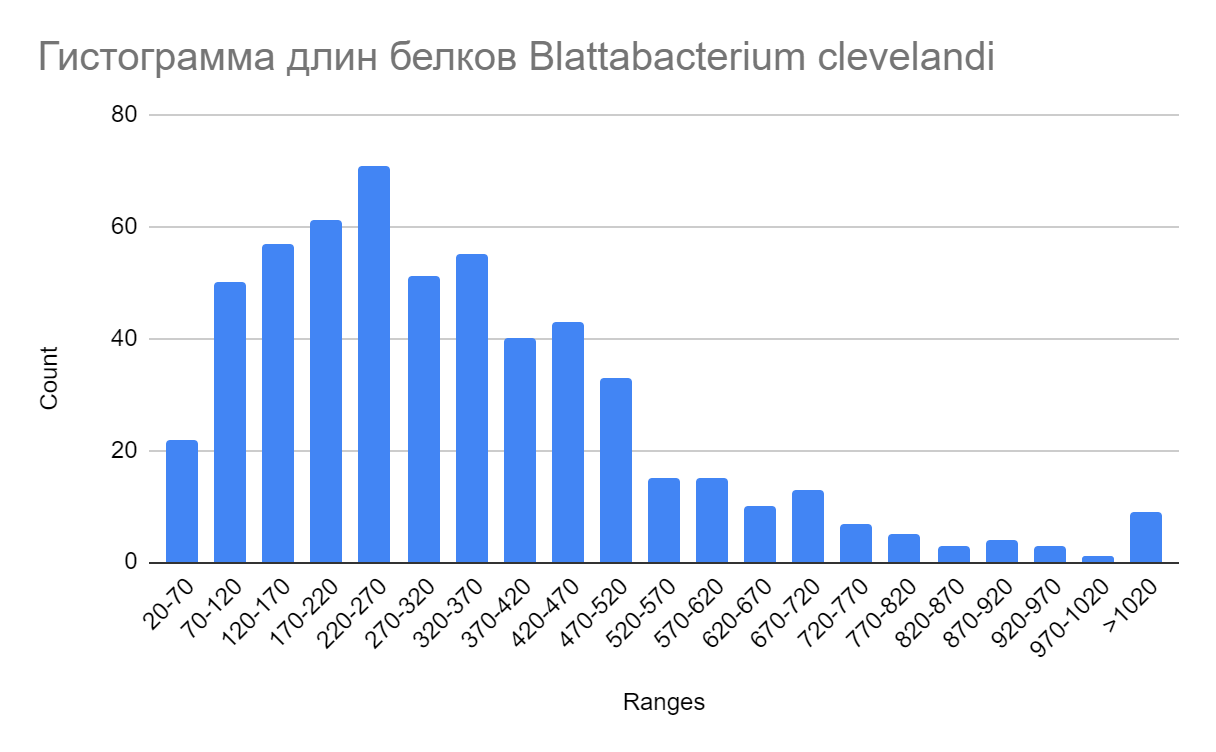
На полученной гистограмме(рисунок 2) представлено количество белков в зависимости от их размера. Наибольшее число белков имеет длину 220 — 270 аминокислотных остатков, в то время как наибольший из встретившихся — 1432, а наименьший — 31. Медианная длина — 290 ( см.таблицу в сопроводительных материалах )
4.2 Количество нуклеотидов
| Количество A | 232111 |
|---|---|
| Количество T | 236237 |
| Количество G | 78828 |
| Количество C | 73595 |
| Итого | 620771 нуклеотидов |
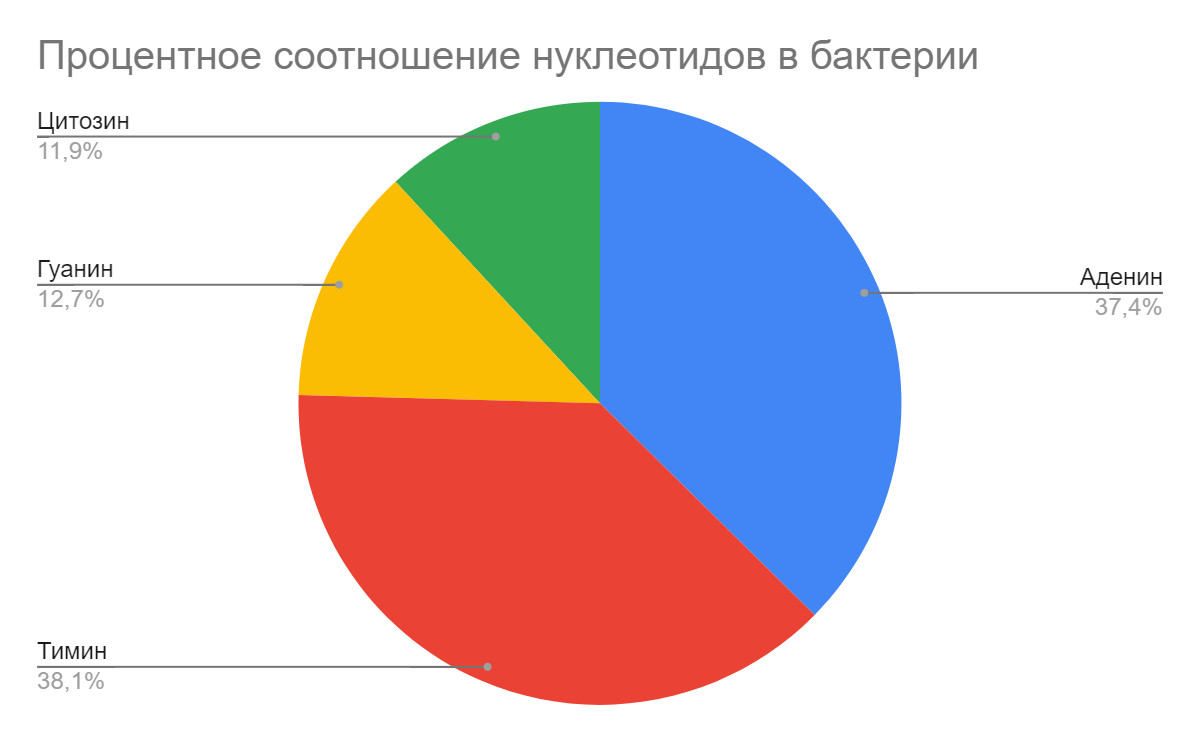
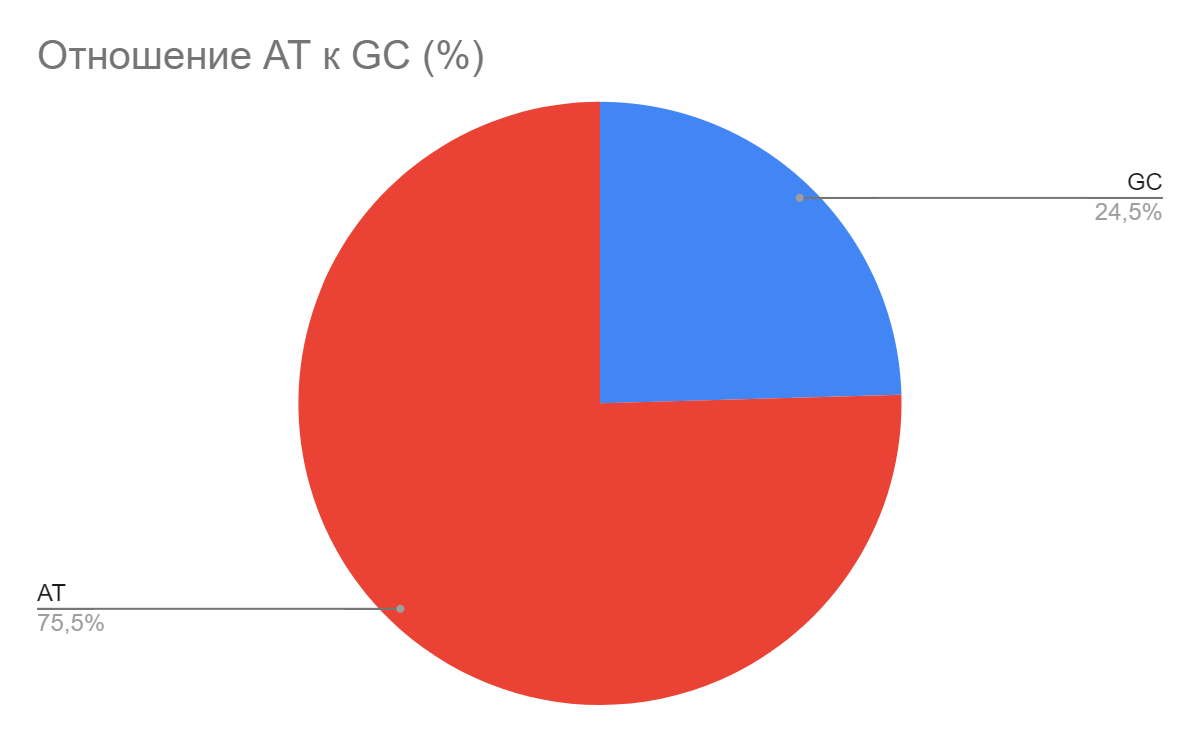
Даже без дополнительного анализа, мы можем увидеть по диаграмме(рисунок 3) насколько сильно AT преобладает над GC в геноме нашей бактерии. Теперь, зная количество нуклеотидов, мы можем узнать много других вещей( вроде GC-skew/CGC-skew/Z-curve)
4.3 GC-состав
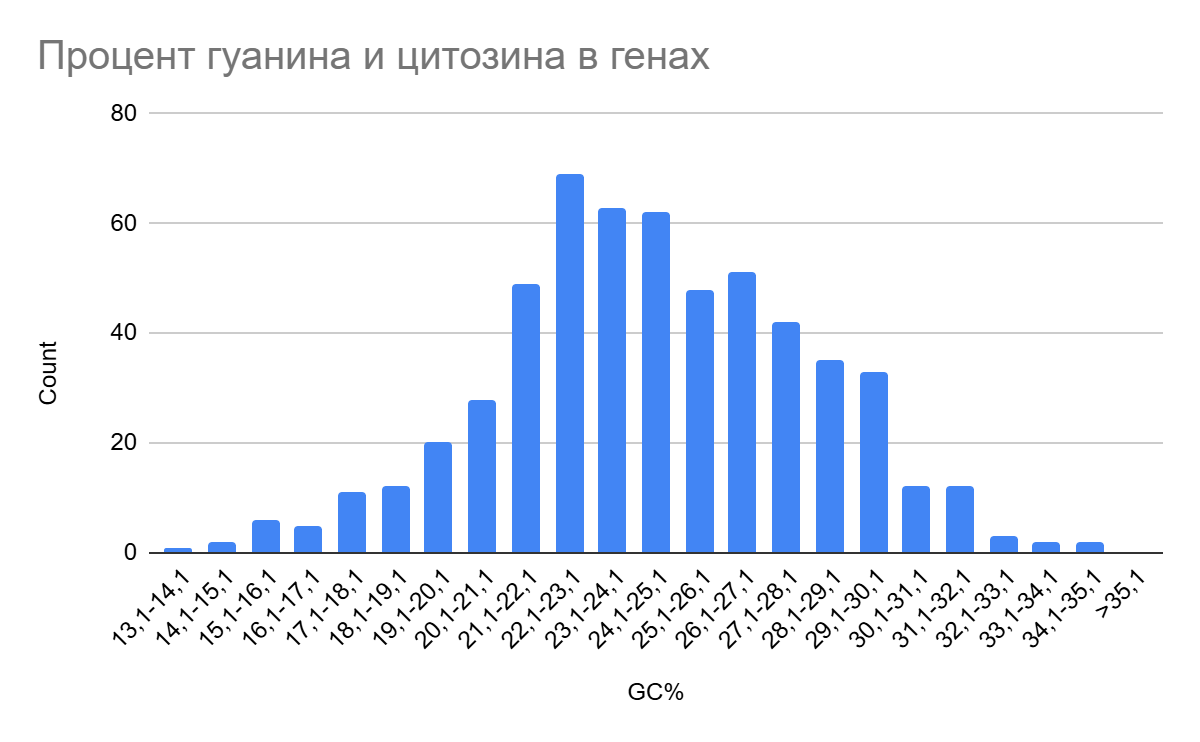
Таким образом, в белках Blattabacterium clevelandi преобладают AT нуклеотиды, при наибольшем проценте содержания GC = 35,04%, и наибольшим числом генов(69) содержащих 22,1 — 23,1%(рисунок 4). Медианный процент = 24.42.
Соответственно мы можем составить диаграмму(рисунок 5) отношения AT к GC [4]:
4.4 GC skew/AT deviation
GC skew, или по-другому GC перекос — это когда имеется избыток или ,напротив, недостаток гуанина/цитозина на участке ДНК/РНК. Есть мнение, что подобный перекос возникает в результате разной частоты мутаций в разных цепочках [6].
Для нахождения отклонения от [C] = [G] воспользуемся формулой:
GC deviation = (G - C)/(G + C)
Т.е., GC deviation = (78828-73595)/(78828+73595) = 0,03433208898
То же можно проделать и с AT:
AT deviation = (A - T)/(A + T)
Т.е., AT deviation = (232111-236237)/(232111+236237) = −0,00880968852
4.5 Количество старт-кодонов и стоп-кодонов
| TAA | 23881 |
|---|---|
| TAG | 10253 |
| TGA | 10014 |
| Всего: | 44148 |
| ATG | 10430 |
|---|---|
| TTG | 10476 |
| GTG | 2105 |
| Всего: | 23011 |
Мы можем объяснить подобное количество старт и стоп кодонов(таблица 3, таблица 4) содержанием AT и GC нуклеотидов. Так как AT встречаются чаще, то неудивительно и то, что TAA встречается более чем в два раза чаще чем TGA или TAG.
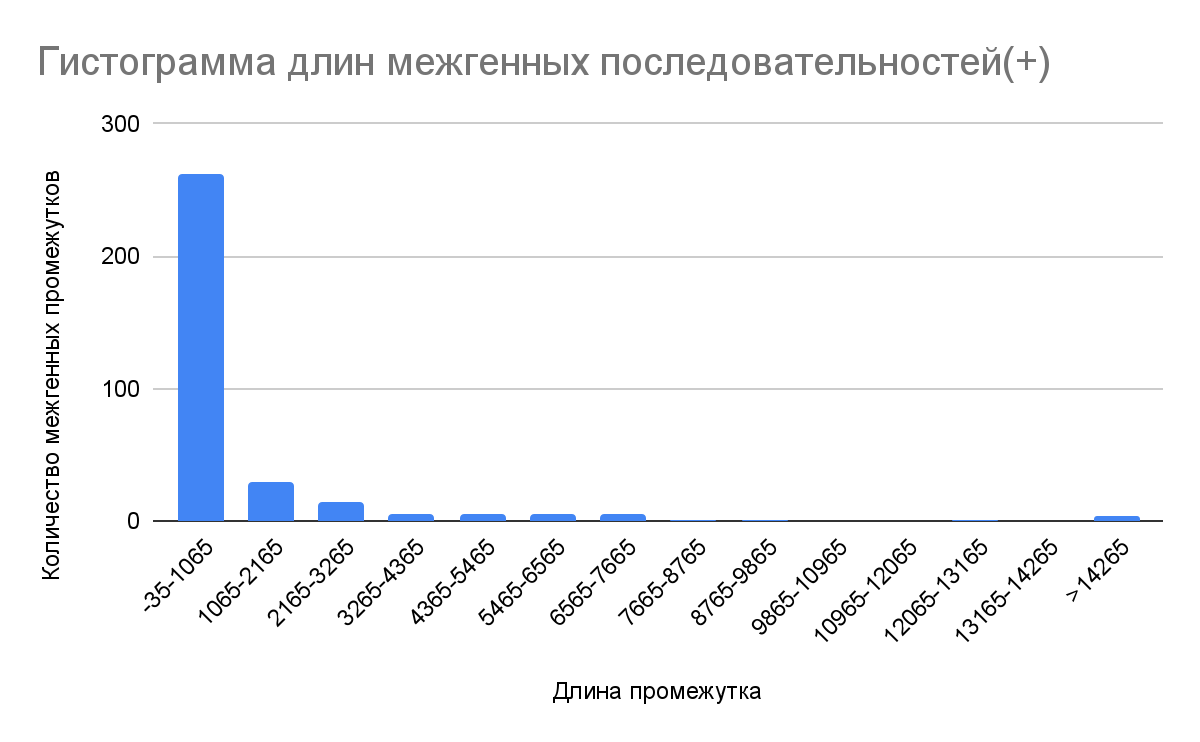
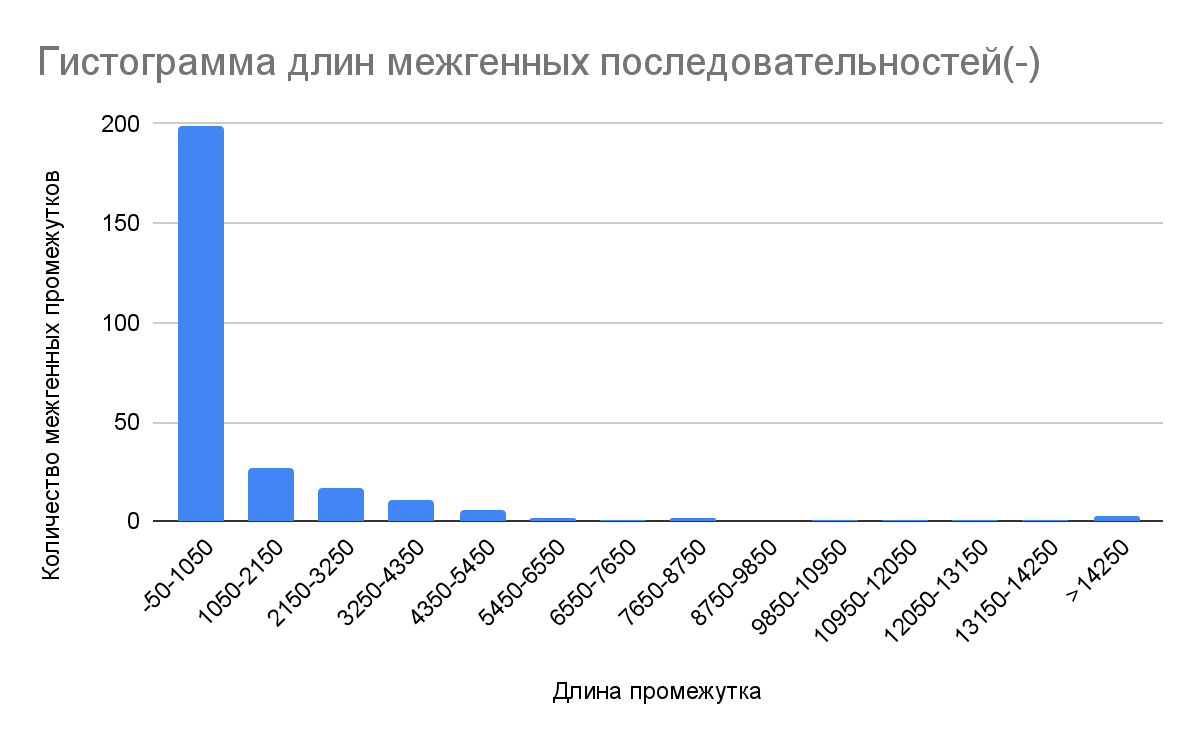
4.6 Гистограммы длин промежутков между кодирующими последовательностями в хромосоме
Межгенные промежутки — это участки, не кодирующие гены и не обладающие какой-то информационной ценностью. Гистограммы длин промежутков на прямой и комплементарной цепях днк представлены ниже(рисунок 6, рисунок 7). Также, глядя на диаграммы, мы можем заметить такое интересное явление, как перекрывание генов(это можно понять по отрицательным значениям длины межгенных последовательностей). Перекрывающиеся гены — это гены, имеющие общие участки днк. Существует мнение, что подобное припособление помогает делать код более компактным, а также участвует в регуляции экспрессии генов [7].
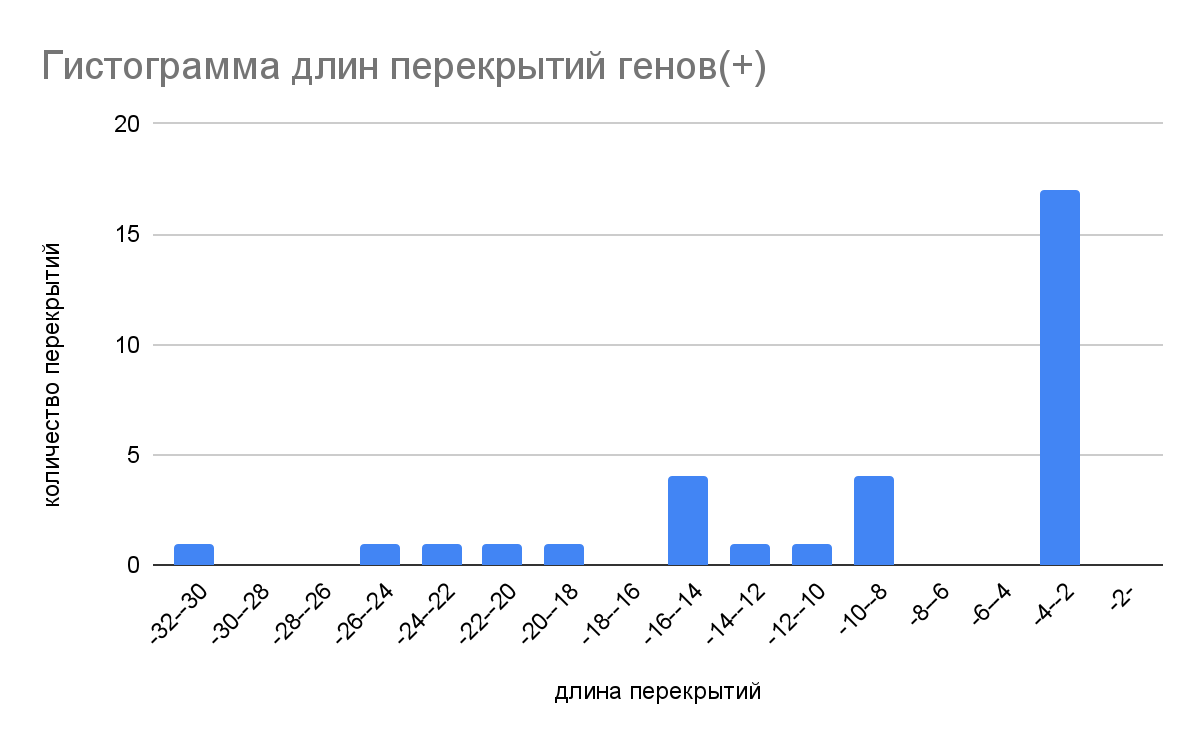
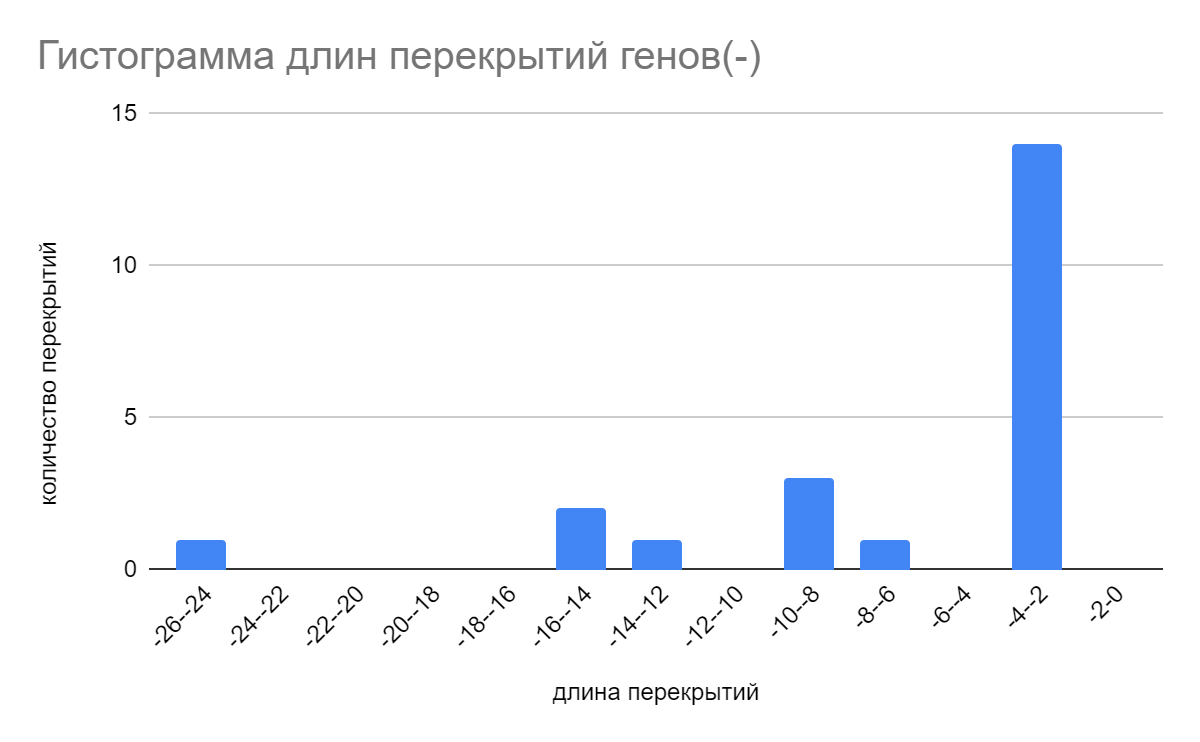
4.7 Гистограммы длин перекрываний между генами в хромосоме
В результате анализа перекрытий, выяснилось что на основной цепи всего 32 перекрытия, а на комплементарной только 28. Наибольшая часть перекрытий на основной цепи(17) имеют длину в 3 нуклеотида(рисунок 8). То же верно и для комплементарной последовательности, в которой уже 14 перекрытий имеют длину в 3 нуклеотида(рисунок 9).
4.8 Анализ РНК
При рассмотрении РНК в Feature table, мы обнаружили, что среди всех представленных в бактерии типов РНК, тРНК кодируются наибольшим количеством последовательностей, а некодирующая и транспортно-матричная РНК представлены только одной последовательностью(таблица 5).
| tRNA | 32 |
|---|---|
| rRNA | 3 |
| ncRNA | 1 |
| tmRNA | 1 |
Возможные дальнейшие исследования
В дальнейшем можно исследовать геном бактерии на предмет повторов, которые могут свидетельствовать о наличии МГЭ - мобильных генетических элементов. Согласно современным представлениям, многочисленному семейству МГЭ («прокариотическому мобилому») отводится ключевая роль в эволюции всех живых организмов, включая прокариот и архей. Эти подвижные генетические элементы являются важным звеном, опосредующим быструю фенотипическую адаптацию бактерий к изменяющимся условиям среды обитания и приводящему к их диверсификации. Семейство прокариотического мобилома включает чрезвычайно пёстрый спектр элементов с различной склонностью к осёдлости и подвижности, которые широко распространены в различных таксонах бактерий. Все МГЭ, включая плазмиды, инсерционные последовательности (insertion sequences, IS), транспозоны (Тn), интегроны (In), фаги и другие представители этих генетических «путешественников» в процессе переноса способны вызвать значительные изменения в геноме. Например, через них бактерии могут получить несколько факторов вирулентности, именуемых островками патогенности, изменить пути метаболизма, а также приобрести гены ABR [8].
Для реализации этого можно использовать команду на Python, которая создаст окно длинною в примерно 10 нуклеотидов, и будет сдвигать его, сравнивая с предыдущим выводом.
Также можно заметить, что тРНК располагаются небольшими кластерами, посмотреть, сколько тРНК в каждом кластере, и какие антикодоны они переносят.
Сопроводительные материалы
- Электронная таблица, c данными для гистограммы длин белков и числа генов: ссылка
- Таблицы с геномными и протеомными данными Blattabacterium clevelandi: ссылка
- Код на питон:
- количество нуклеотидов: Nuc_count.ipynb
- количество старт.стоп кодонов: Stop/start-codons.ipynb
Список литературы
- Kambhampati S (2010). "Family II. Blattabacteriaceae fam. nov.". In Krieg NR, Staley JT, Brown DR, Hedlund BP, Paster BJ, Ward NL, Ludwig W, Whitman WB (eds.). Bergey's Manual of Systematic Bacteriology. Vol. 4 (2nd ed.). New York, NY: Springer. p. 315.
- Clark JW, Kambhampati S. Phylogenetic analysis of Blattabacterium, endosymbiotic bacteria from the wood roach, Cryptocercus (Blattodea: Cryptocercidae), including a description of three new species. Mol Phylogenet Evol. 2003 Jan;26(1):82-8. doi: 10.1016/s1055-7903(02)00330-5. PMID: 12470940.
- Grandcolas, Philippe. (1999). Systematics, Endosymbiosis, and Biogeography of Cryptocercus clevelandi and C. punctulatus (Blattaria: Polyphagidae) from North America: A Phylogenetic Perspective. Annals of the Entomological Society of America. 92. 285-291.
- Данные по содержанию GC были взяты с сайта: ссылка
- Yakovchuk P., Protozanova E., Frank-Kamenetskii M. D. Base-stacking and base-pairing contributions into thermal stability of the DNA double helix (англ.) // Nucleic Acids Res. : journal. — 2006. — Vol. 34, no. 2. — P. 564—574.
- Lobry JR. Asymmetric substitution patterns in the two DNA strands of bacteria. Mol Biol Evol. 1996 May;13(5):660-5. doi: 10.1093/oxfordjournals.molbev.a025626. PMID: 8676740.
- David C. Krakauer, STABILITY AND EVOLUTION OF OVERLAPPING GENES, Evolution, Volume 54, Issue 3, 1 April 2000, Pages 731–739, ссылка.
- Andryukov B. G., Besednova N. N., Zaporozhets T. S. Mobile genetic elements of prokaryotes and their role in the formation of antibiotic resistance in pathogenic bacteria. Antibiotiki i Khimioter = Antibiotics and Chemotherapy. 2022; 67: 1–2: 62–74. doi: 10.37489/0235-2990-2022-67-1-2-62-74.