Глобальное и локальное выравнивание аминокислотных последовательностей.
На главную>Второй
семестр>Выравнивание аминокислотных последовательностей
1. Матрица переходов.
Вес совпадения = 2, вес замены = -1, штраф за делецию = -2.
 |
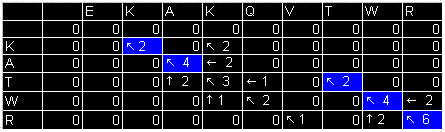 |
Глобальное выравнивание
Seq1:
L L A A G
Seq2:
L A A
LLAAG
Выравнивание: |||
-LAA-
Вес выравнивания (S(A)) = 2 |
Локальное выравнивание
Seq1: EKAKQVTWR
Seq2: KATWR
EKAKQVTWR
Выравнивание: || |||
-KA---TWR
Оптимальный путь S(A)=6
Субоптимальный путь S(A)=4 |
2. Поиск участков локальной гомологии
Seq1:
QVTWRLLAAGFMGDKPTLMS
Seq2:
MEKAKQVTWRLLAAGVCLLTVSSVARADSLDEQRSRYAQIKQAWDNRQMDVVEQMMPGLK
DYPLYPYLEYRQITDDLMNQPAVTVTNFVRANPTLPPARTLQSRFVNELARREDWRGLLA
FSPEKPGTTEAQCNYYYAKWNTGQSEEAWQGAKELWLTGKSQPNACDKLFSVWRASGKQD
PLAYLERIRLAMKAGNTGLVTVLAGQMPADYQTIASAIISLANNPNTVLTFARTTGATDF
TRQMAAVAFASVARQDAENARLMIPSLAQAQQLNEDQIQELRDIVAWRLMGNDVTDEQAK
WRDDAIMRSQSTSLIERRVRMALGTGDRRGLNTWLARLPMEAKEKDEWRYWQADLLLERG
REAEAKEILHQLMQQRGFYPMVAAQRIGEEYELKIDKAPQNVDSALTQGPEMARVRELMY
WNLDNTARSEWANLVKSKSKTEQAQLARYAFNNQWWDLSVQATIAGKLWDHLEERFPLAY
NDLFKRYTSGKEIPQSYAMAIARQESAWNPKVKSPVGASGLMQIMPGTATHTVKMFSIPG
YSSPGQLLDPETNINIGTSYLQYVYQQFGNNRIFSSAAYNAGPGRVRTWLGNSAGRIDAV
AFVESIPFSETRGYVKNVLAYDAYYRYFMGDKPTLMSATEWGRRY
Участки наилучшего выравнивания полученные при помощи matcher.
630
SLT_ECOLI FMGDKPTLMS
::::::::::
seq3 FMGDKPTLMS
20
10
SLT_ECOLI QVTWRLLAAG
::::::::::
seq3 QVTWRLLAAG
10
SLT_ECOLI VTWRLL
::::::
seq3 VTWRLL
290
3. Влияние параметров на глобальное выравнивание
Заметно, что при высокой цене GO гэпы в последовательности seq3 отсутствуют, а при GO=1 уже присутствует разбиение, следовательно цена на открытие и продолжение делеции существенно влияет на вид выравнивания.
| GO=10 GE=1 BLOSUM62 |
GO=1 Ge=1 BLOSUM62 |
1 MEKAKQVTWRLLAAGVCLLTVSSVARADSLDEQRSRYAQIKQAWDNRQMD 50
1 0
51 VVEQMMPGLKDYPLYPYLEYRQITDDLMNQPAVTVTNFVRANPTLPPART 100
1 0
101 LQSRFVNELARREDWRGLLAFSPEKPGTTEAQCNYYYAKWNTGQSEEAWQ 150
1 0
151 GAKELWLTGKSQPNACDKLFSVWRASGKQDPLAYLERIRLAMKAGNTGLV 200
1 0
201 TVLAGQMPADYQTIASAIISLANNPNTVLTFARTTGATDFTRQMAAVAFA 250
1 0
251 SVARQDAENARLMIPSLAQAQQLNEDQIQELRDIVAWRLMGNDVTDEQAK 300
1 0
301 WRDDAIMRSQSTSLIERRVRMALGTGDRRGLNTWLARLPMEAKEKDEWRY 350
1 0
351 WQADLLLERGREAEAKEILHQLMQQRGFYPMVAAQRIGEEYELKIDKAPQ 400
1 0
401 NVDSALTQGPEMARVRELMYWNLDNTARSEWANLVKSKSKTEQAQLARYA 450
1 0
451 FNNQWWDLSVQATIAGKLWDHLEERFPLAYNDLFKRYTSGKEIPQSYAMA 500
1 0
501 IARQESAWNPKVKSPVGASGLMQIMPGTATHTVKMFSIPGYSSPGQLLDP 550
1 0
551 ETNINIGTSYLQYVYQQFGNNRIFSSAAYNAGPGRVRTWLGNSAGRIDAV 600
1 QVTWRLLAAGFMGDKPTLMS 20
.:.:......||||||||||
601 AFVESIPFSETRGYVKNVLAYDAYYRYFMGDKPTLMSATEWGRRY 645
|
1 MEKAKQVTWRLLAAGVCLLTVSSVARADSLDEQRSRYAQIKQAWDNRQMD 50
1 0
51 VVEQMMPGLKDYPLYPYLEYRQITDDLMNQPAVTVTNFVRANPTLPPART 100
1 0
101 LQSRFVNELARREDWRGLLAFSPEKPGTTEAQCNYYYAKWNTGQSEEAWQ 150
1 0
151 GAKELWLTGKSQPNACDKLFSVWRASGKQDPLAYLERIRLAMKAGNTGLV 200
1 0
201 TVLAGQMPADYQTIASAIISLANNPNTVLTFARTTGATDFTRQMAAVAFA 250
1 0
251 SVARQDAENARLMIPSLAQAQQLNEDQIQELRDIVAWRLMGNDVTDEQAK 300
1 0
301 WRDDAIMRSQSTSLIERRVRMALGTGDRRGLNTWLARLPMEAKEKDEWRY 350
1 0
351 WQADLLLERGREAEAKEILHQLMQQRGFYPMVAAQRIGEEYELKIDKAPQ 400
1 0
401 NVDSALTQGPEMARVRELMYWNLDNTARSEWANLVKSKSKTEQAQLARYA 450
1 0
451 FNNQWWDLSVQATIAGKLWDHLEERFPLAYNDLFKRYTSGKEIPQSYAMA 500
1 0
501 IARQESAWNPKVKSPVGASGLMQIMPGTATHTVKMFSIPGYSSPGQLLDP 550
1 0
551 ETNINIGTSYLQYVYQQFGNNRIFSSAAYNAGPGRVRTWLGNSAGRIDAV 600
1 QVT--W-R-LLA--A--G-FMGDKPTLMS 20
: | : : :|| | . ||||||||||
601 AFVESIPFSE-TRGYVKNVLAYDAYYRYFMGDKPTLMSATEWGRRY 645 |

