На главную>Третий семестр> Эволюционная модель
Данное задание было заключалось в том, что самостоятльно мутировали нуклеотидную последовательность, и поэтому мы знали точну структуру дерева, промутировав и получив некоторый набор последовательностей, приступили к реконструкции деревьев, а также размышляли о возможности использования данных методов в качестве инструментов рекнострукции.
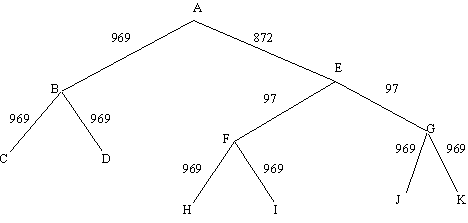
Изначально у нас была нуклеотидная последовательность белка SLT_ECOLI и в качестве вводной информации нам предоставили скобочную структуру дерева - ((A:50,B:50):50,((C:50,D:50):5,(E:50,F:50):5):45), число мутаций в котором дано на 100 нуклеотидов. Для построения эволюционной модели нам необходимо было рассчитать расстояние для нашей последовательности, длина которой составляет 1938 нуклеотидов, поэтому расстояние мы определяли домножая расстояние указанное в скобочной структуре на 19,38 и округляя до целого числа. В результате получили дерево со следующими расстояниями:

Затем используя программу msbar из пакета EMBOSS получили мутантные последовательности, для упрощения решения этой задачи - был сделан скрипт:
msbar ecoli.fasta b.fasta -point 4 -count 969 -auto msbar ecoli.fasta e.fasta -point 4 -count 872 -auto msbar b.fasta c.fasta -point 4 -count 969 -auto msbar b.fasta d.fasta -point 4 -count 969 -auto msbar e.fasta f.fasta -point 4 -count 97 -auto msbar e.fasta g.fasta -point 4 -count 97 -auto msbar f.fasta h.fasta -point 4 -count 969 -auto msbar f.fasta i.fasta -point 4 -count 969 -auto msbar g.fasta j.fasta -point 4 -count 969 -auto msbar g.fasta k.fasta -point 4 -count 969 -auto
Анализ методов реконструкции деревьев