Выравнивание последовательностей
Глобальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| Acetyl-coenzyme A carboxylase carboxyl transferase subunit beta | ACCD_ECOLI | ACCD_BACSU | 597 | 41,3% | 58,4% | 36 | 7 |
| Purine nucleoside phosphorylase DeoD-type | DEOD_ECOLI | DEOD_BACSU | 692 | 56,1% | 73,2% | 6 | 3 |
| Deferrochelatase | EFEB_ECOLI | EFEB_BACSU | 661,5 | 36,4% | 50,9% | 45 | 15 |
Локальное парное выравнивание гомологичных белков
| Protein Name | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Acetyl-coenzyme A carboxylase carboxyl transferase subunit beta | ACCD_ECOLI | ACCD_BACSU | 614 | 48,5% | 66,5% | 5 | 3 | 85,2% | 88,3% |
| Purine nucleoside phosphorylase DeoD-type | DEOD_ECOLI | DEOD_BACSU | 698 | 58,5% | 76,0% | 1 | 1 | 95,8% | 97,9% |
| Deferrochelatase | EFEB_ECOLI | EFEB_BACSU | 665,5 | 37,1% | 52,1% | 42 | 13 | 96,5% | 95,0% |
Локальное парное выравнивание неродственных белков
| Выравнивание | ID 1 | ID 2 | Score | % Identity | % Similarity | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Needle | BLC_ECOLI | BSAA_BACSU | 25 | 6,8% | 11,2% | 219 | 7 | - | - |
| Water | BLC_ECOLI | BSAA_BACSU | 35,5 | 27,3% | 42,4% | 13 | 4 | 37,3% | 31,9% |
Множественное выравнивание белков и импорт в Jalview
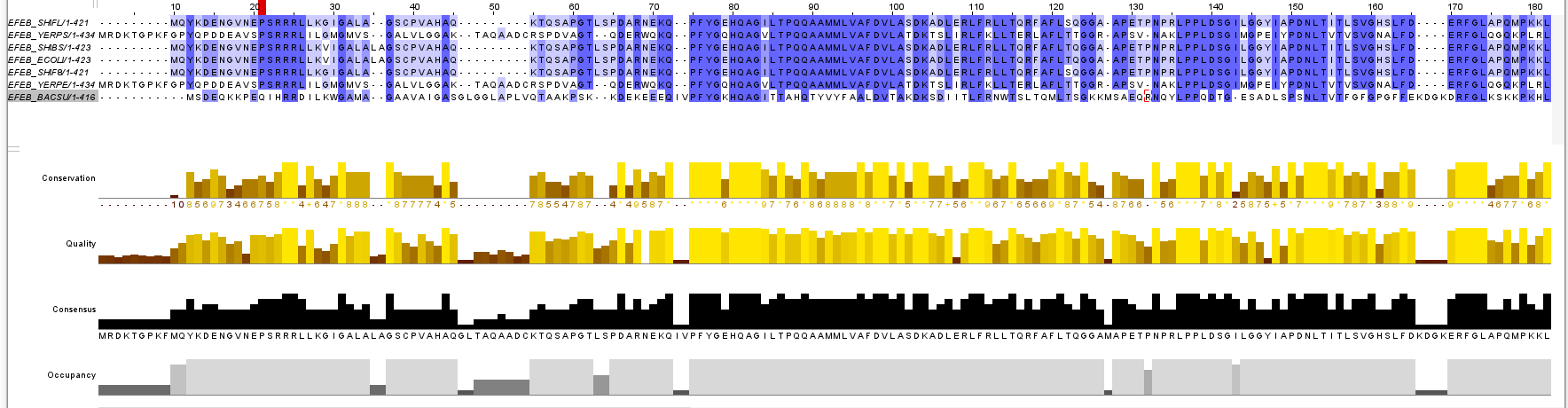
Для данного задания были найдены белки с мнемоникой "EFEB", полное имя белков - "Deferrochelatase". Всего таких оказалось 14 штук. Далее были взяты : EFEB_ECOLI, EFEB_YERPS, EFEB_SHIFL, EFEB_SHIF8, EFEB_YERPE, EFEB_SHIBS.
С помощью программы Jalview было произведено выравнивание последовательностей белков (Web service -> Alignment -> Muscle with Defaults). Для упрощения восприятия была применена окраска Percentage Identity.
Хорошо выравнялись и являются гомологичными все белки, кроме EFEB_BACSU, т.к у них много длинных консервативных участков, и практически все промежутки между ними занимаются менее консервативными участками.
Ссылка на проект