Мотивы в доменах белков
Поиск консервативного мотива в seed
Я взяла домен PF01954. В seed у него 41 последовательность,
в Swiss-Prot 20 белков (графа reviewed в proteins).
Затем скачала выранивание seed и открыла в Jalview. Изменить на msf?
Попробовала удалить одинаковые последовательности - на 95% из 40 последовательностей остались все 40.
При покраске Above identity threshold на 95% проявилась первая колонка, на 90% - выделилось 4 колонки рядом (ну почти).
При снижении порога до 85% подсветилось 7 колонок в этом же участке

Я не нашла, что что конкретно отвечает мотив YE.GV[LF]KPL, но этот домен в целом antitoxin components of the a type II toxin-antitoxin (TA) system
Данный мотив нашелся в 24 последовательностях из 40, все в нужном месте. Т.е. этот мотив достаточно консервативен и специфичен. В формате Prosite данный мотив выглядит так : Y-E-x-G-V-[LF]-K-P-L . В ScanProsite нашлось 15 последовательностей. страница выдачи сканпросайт Putative antitoxin VapB4. Archaeoglobus fulgidus итп Putative antitoxin APE_0279a.1. Aeropyrum pernix Putative antitoxin PYRAB11980. Pyrococcus abyssi итп Putative antitoxin MJECL31. Methanocaldococcus jannaschii Много находок у организма Archaeoglobus fulgidus, что было ожидаемо, т.к AF2212 характерен имеенно для этой бактерии. Также были найдены данный мотивы и в других бактериях, причем все они отвечают за utative antitoxin
Мотив, специфичный для одной клады филогенетического дерева
По выраниваниям seed было построено дерево с помощью NJ в Jalview.
Calculate-Calculate tree-NJ-выбрать кладу-перенести последовательности в новый файл-найти мотив-искать мотив в исходном выравнивании
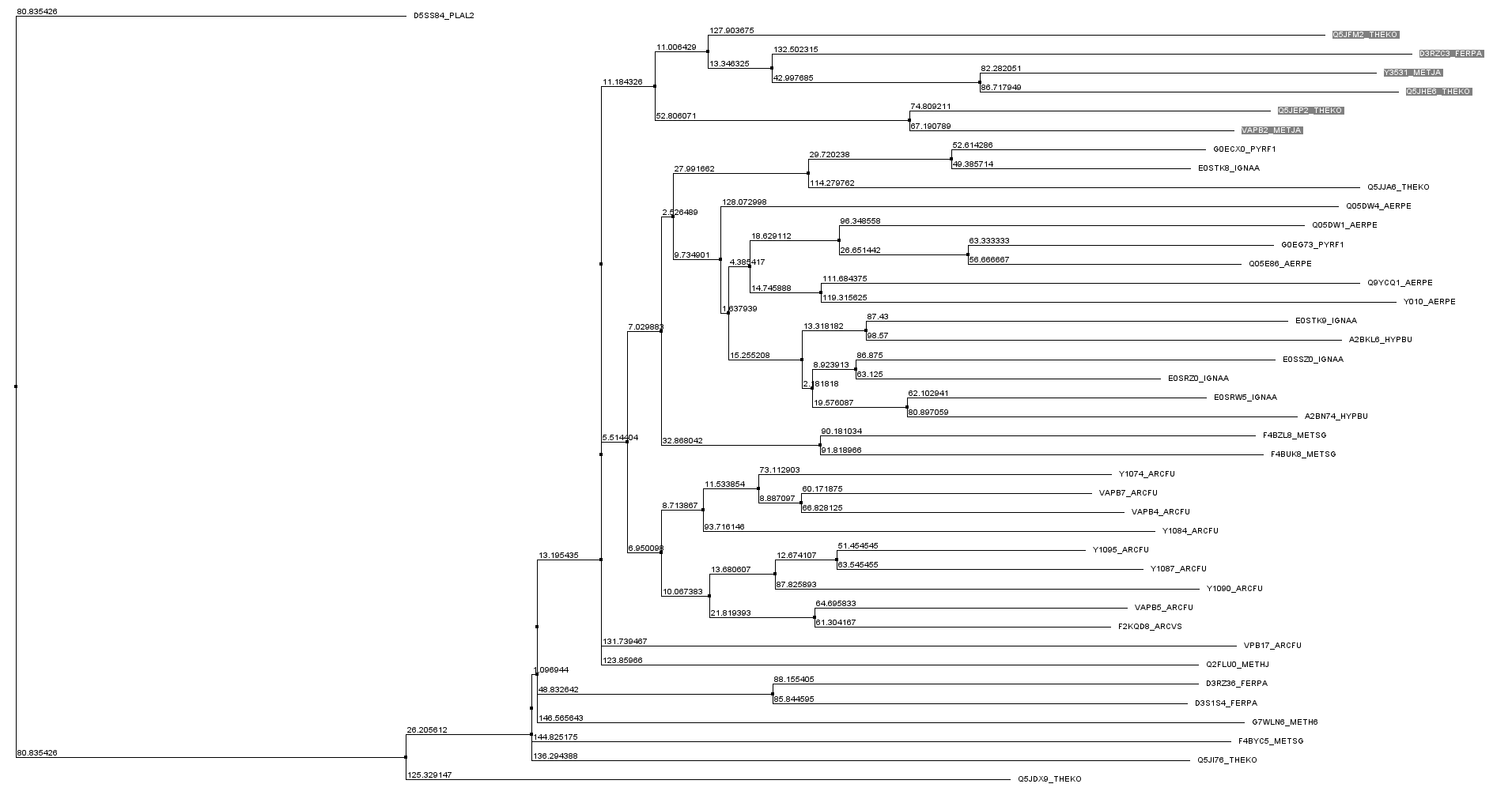
Далее я решила найи уникальный консервативный мотив для верхней клады из 6 последовательностей.
Получился паттерн YE.GVL (100%) - он нашелся в 18 последовательностях, которые в одну ветвь не собираются. Значит, либо данный мотив не
является характерным для какой-либо одной клады (что очень вероятно, т.к. выравнивание короткое и данный мотив является частью большого
консервативного мотива для всех последовательнстей), либо дерево было построено неверно.
PSI-BLAST
В данном задании составляется семейство гомологов для белка Q67XL4 с помощью программы
PSI-BLAST
Это хлоропластный белок с неизвестной функцией, содержащий домен CRM At3g25440
За 4 итерации PSI-BLAST список находок стабилизировался - 21 последовательность найдена (список находок выше порога не поменялся по сравнению с предыдущей итерацией).
Т.к. разница между худшей находкой выше порога и лучшей находкой ниже порога большая, то можно сделать
вывод, что найдено семейство гомологичных белков.
Судя по схожему описанию находок, семейство было определено верно.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 18 | Q9FFU1.1 | 0.004 | Q9SL79.2 | 0.012 |
| 2 | 20 | Q9LDA9.1 | 2,00E-29 | - | - |
| 3 | 21 | P54454.1 | 2,00E-05 | - | - |
| 4 | 21 | P54454.1 | 8,00E-20 | B0RGZ2.1 | 0.012 |
В выборке поледовательностей с доменом из SwissProt найдите de novo мотивы с помощью MEME
Я решила рассмотреть род Thermococcus, у него для данного домена есть 187 белков в 48 видах. Я скачала их последовательности
в формате fasta
Запустила meme :
meme thermo.fasta -minw 4 -maxw 12 -nmotifs 3
В результате мы получили 3 мотива . В самой лучшей находке с низким E-value виден консервативный мотив YEnGVLKP,
ранее найденный в seed. Раз мы его нашли с отличной точностью, то он явно является важным для домена PF01954.
meme.html

Далее я проверила, сколько раз встречается мотив IEAVYENGVLKP :
fimo -motif IEAVYENGVLKP -thresh 0.001 meme_out/meme.txt thermo.fasta
Он нашелся в 223 местах в 187 последовательностях. Мотив немного перепредставлен, но, возможно, могут встречаться два одинаковых
домена в одной архитектуре.
fimo.html
Оценка представленности сайта GATC в геноме бактерии
Для оценки представленности была исользована программа cbcalc на kodomo для вычисления контрастов
cbcalc -s sites.txt -M -o res.tsv GCF_019668465.1_ASM1966846v1_genomic.fna
Для ее запуска был создан список перестановок без повторения G, A, T, C - sites.txt
Затем по результатам res.tsv я построила гистограмму :
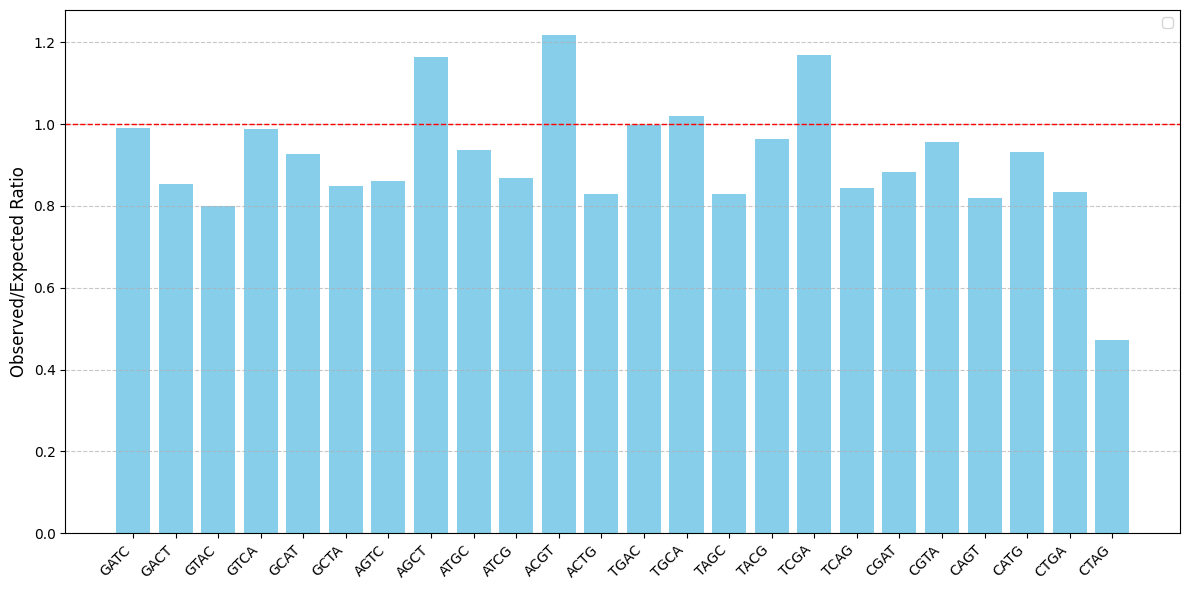
Как видно по рис. 2, сайт метилирования GATC имеет представленность, практически равную 1. Значит, этот сайт важен для метилирования, но существуют и другие сайты с большей представленностью. Возможно, они играют большую роль в метилировании.