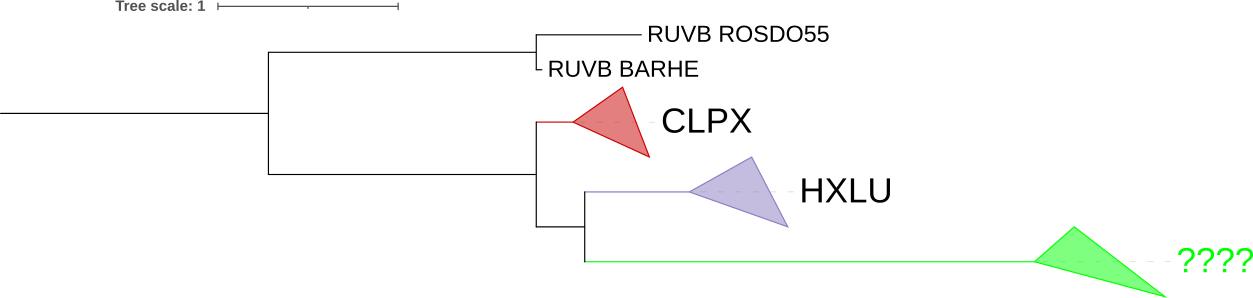
Ортологи и паралоги
Отобранные гомологи CLPX_ECOLI
Из директории на kodomo я отобрала 7 бактерий.
- ACICJ
- BARHE
- HAEIN
- NEIMA
- POLAQ
- ROSDO
- THIDA
cat /P/y22/term4/Proteomes/ACICJ.fasta /P/y22/term4/Proteomes/BARHE.fasta /P/y22/term4/Proteomes/HAEIN.fasta /P/y22/term4/Proteomes/NEIMA.fasta /P/y22/term4/Proteomes/POLAQ.fasta /P/y22/term4/Proteomes/ROSDO.fasta /P/y22/term4/Proteomes/THIDA.fasta > pr5/pr_db.fasta
У этих бактерий я нашла белки, гомологичные белку CLPX_ECOLI
nano CLPX_ECOLI.fasta - создала файл с последовательностью исходного белка
makeblastdb -in pr_db.fasta -dbtype prot
blastp -task blastp -query CLPX_ECOLI.fasta -db pr_db.fasta -out blast.out -evalue 0.0001
В результате получилас список находок в blast.out
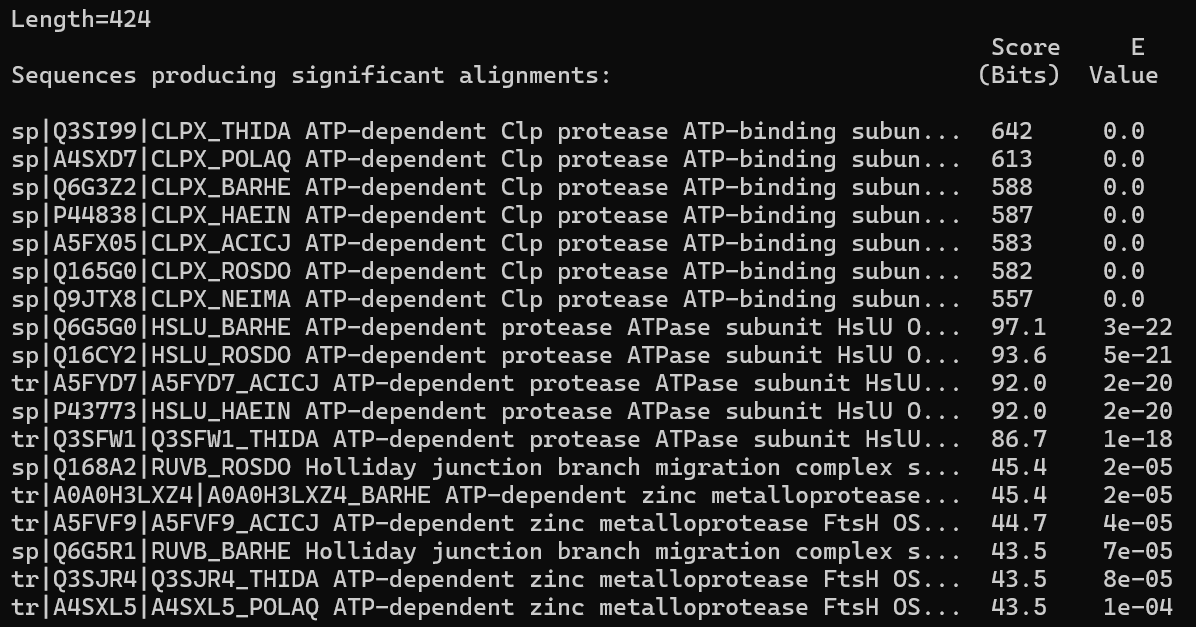
В файл clpx_mid.list записала sw находок
Применила code.txt в командной строке и получила формулу дерева
Реконструкция и визуализация
Формула дерева в формате Newick : clpxm.tre
Последовательности в формате fasta : clpxm.fasta
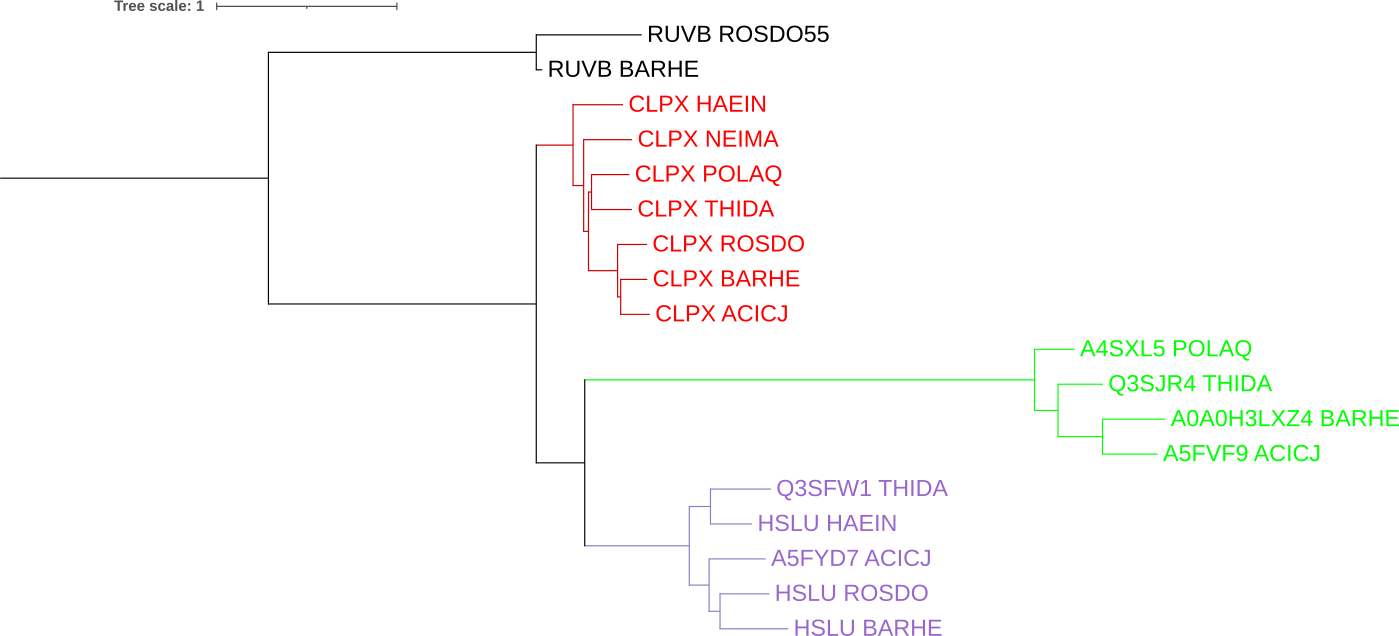
В программе iTol я построила дерево и с помощью label_pr5.txt покрасила разные ортологические группы
Ортологи - гомологичные белки из разных организмов, разделившихся в результате видообразования.
Паралоги - гомологичные белки из одного организма.
В данном случае ортологи выделены цветами, а паралогами являются :
- CLPX HAENI & HSLU HAENI
- CLPX THIDA & Q3SFW1 THIDA & Q3SJR4 THIDA
- CLPX ROSDO & HSLU ROSDO
- CLPX BARHE & HSLU BARHE & A0A0H3LXZ4 BARHE
- CLPX ACICJ & A5FVF9 ACICJ & A5FYD7 ACICJ
- CLPX POLAQ & A4SXL5 POLAQ
На следующей картинке ортологические группы схлопнуты - клады были названы по наиболее часто встречающемуся белку, т.е
CLPX, HSLU и клада с разными белками.
Далее я проанализировала соответсвие филогении белков филогенетическому дереву бактерий
- CLPX :
- должно было быть разделение на ROSDO, BARHE, ACICJ и всех остальных (укоренение произошло не туда)
- должно было быть разделение на ROSDO, BARHE и ACICJ (поменять ROSDO и ACICJ местами)
- должен был быть общий предок у NEIMA, THIDA, POLAQ и HAENI
- ??? :
- должны быть клады POLAQ, THIDA и BARHE, ACICJ (укоренение не туда)
- HSLU :
- все верно