Базы Данных
Для последующего поиска по базам данных взаимосвязей между генами мне были даны 8 генов человека,
вот их ID.
Названия этих генов мне ничего не говорят (хорошо хоть, буквенные), поэтому, чтобы понимать что вообще нужно найти, я решила
посмотреть их описание в Uniprot. Выяснилось, что их основными функциями являются:
- катаболизм лейцина, изолейцина
- биосинтез жирных кислот (карбоксилирование ацетил-КоА)
- катаболизм жирных кислот
- не функция, но часто используется биотин как кофермент для карбоксилирования
- и просто участие в метаболизме
GO
База данных GO представляет собой граф биологических терминов, соединенных различными отношениями (узлы - термины GO, ребра - отношения между терминами)
- В GO есть три группы терминов:
- молекулярные функции - специфическая активность генного продукта на молекулярном уровне
- биологические процессы - сложные явления, необходимые для жизнедеятельности организмов и происходящие благодаря осуществлению последовательности молекулярных функций
- клеточные компоненты - части клетки или внеклеточного пространства, где осуществляется функция генного продукта
Таким образом, GO позволяет установить связь между генами.
Еще в GO есть система классификации PANTHER - она хранит информацию об эволюции семейств генов, кодирующих белки , в частности о филогении белков, функциях и генетических вариациях, влияющих на эти функции. С помощью нее можно проводить анализ обогащения терминами и статистические тесты.
Просто ссылка на базу данных GO
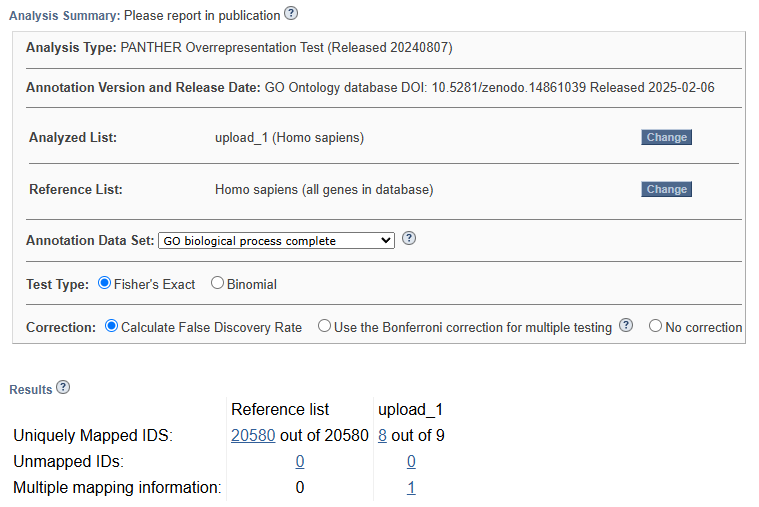
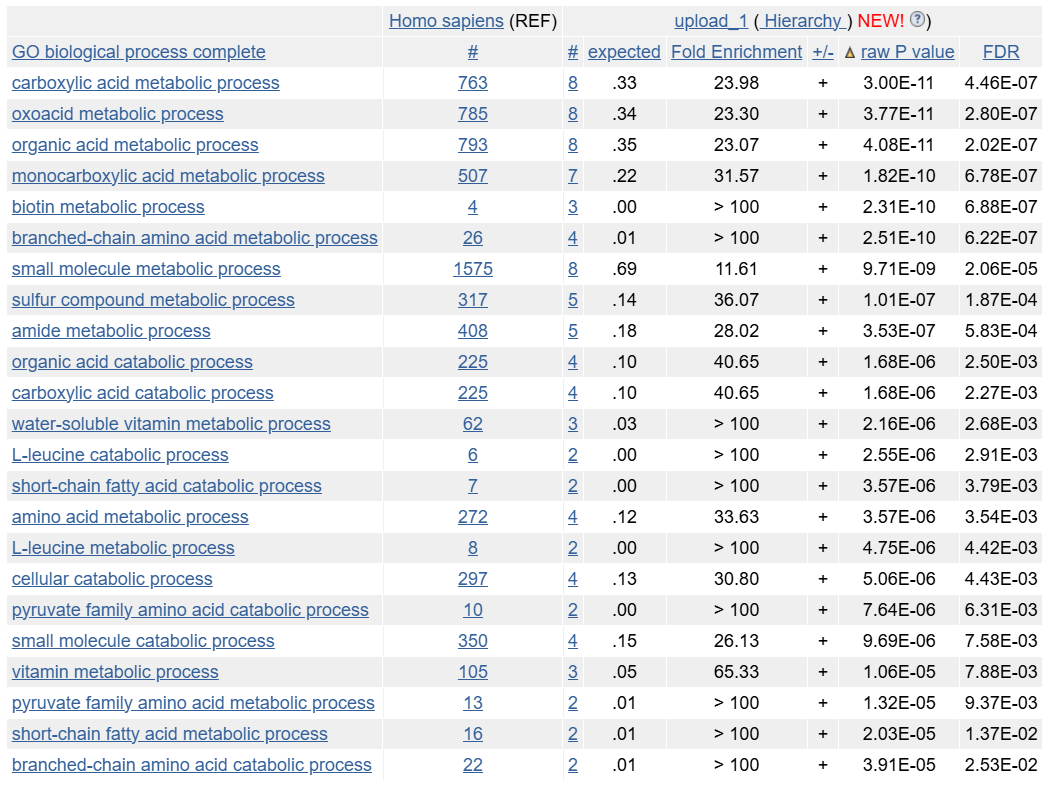
Полная выдача поиска по базе данных GO: biological_process.txt
Я решила провести анализ обогащения терминами по биологическим процессам.
Как видно из результатов выдачи, мои белки являются участниками процессов метаболизма биотина, метаболизма карбоновых кислот и карбоксилирования, катаболизма лейцина и пируватов.
Наиболее значимые результаты можно найти, посмотрев на графу Fold Enrichment - чем больше значение, тем больше
наши гены представлены в данном процесее (Homo sapiens REF # - скольго генов всего для этого процесса,
следующая графа # - сколько наших генов участвуют)
Также нужно смотреть на p-value - оно отображает значимость находки (а вдруг мы случайно нашли не то...) и
делать поправку на множественное тестирование (FDR).
Результаты GO впадают с функциями генов, указанными в Uniprot, а значит, анализ был проведен правильно и данная база данных выдает релевантную классификацию генов по их функциям.
KEGG
KEGG - база знаний по систематическому анализу функций генов.
- В ней содержится 16 баз данных, разделенных на четыре категории:
- системная информация - данные о метаболических путях, модулях генов и т. д.
- геномная информация - данные об отдельных генах, геномах различных существ, ортологичных группах
- химическая информация - данные о химических реакциях, происходящих в живых организмах
- информация, связанная со здоровьем человека - данные о человеческих болезнях, лекарствах и т. д.
Базы данных KEGG представляют данные в виде графических диаграмм, включающих большинство метаболических путей и некоторые из наиболее известных регуляторных путей. Кроме того, информация о путях представлена в виде таблиц ортологов, которые содержат как гены-ортологи, так и паралоги из различных организмов
В дальнейшем я буду использовать PATHWAY - база данных, содержащая данные о генных продуктах, связанных в сети белок-белковых взаимодействий, сети ферментов и регуляторов генов.
Осуществляя анализ генов и их функций, я уже примерно представляла, что они делают и где используются -
поэтому в PATHWAY нашла схему Fatty acid biosynthesis и с помощью нее искала другие пути, в которых участвуют мои гены.
Впрочем, поиск можно осуществлять по отдельным генам - в результате мы получим схемы, в которых данные гены присутсвуют и играют какую-то роль.
Просто ссылка на базу данных KEGG
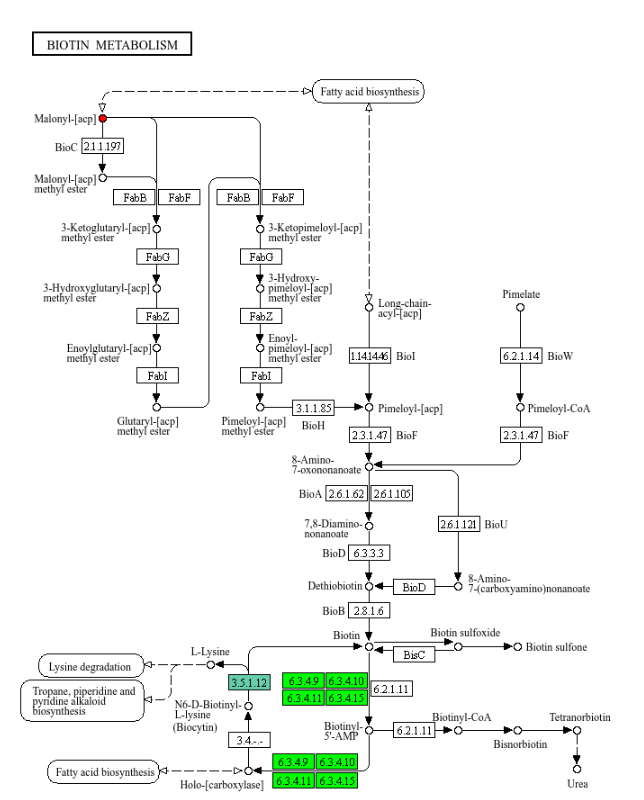
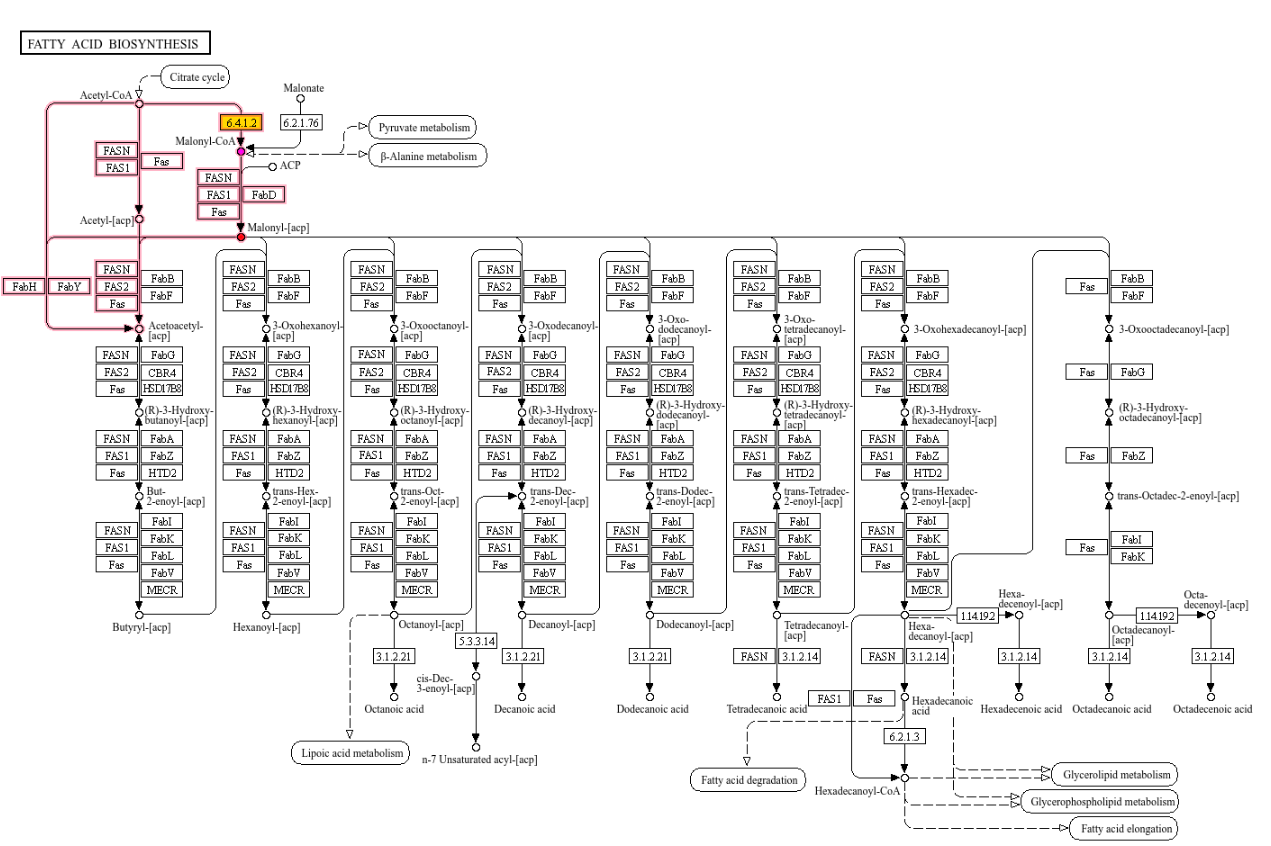
В самом начале цикла используется Ацетил-КоА, который может образовываться в цикле деградации лейцина (рис. 7)
Малонил-КоА образуется при участии АСАСА и нужен для начала метаболизма пирувата либо перехода от метаболизма пирувата к жирным кислотам.

РС участвует в пути глюконеогенеза (метаболический путь, приводящий к образованию глюкозы из неуглеводных соединений).
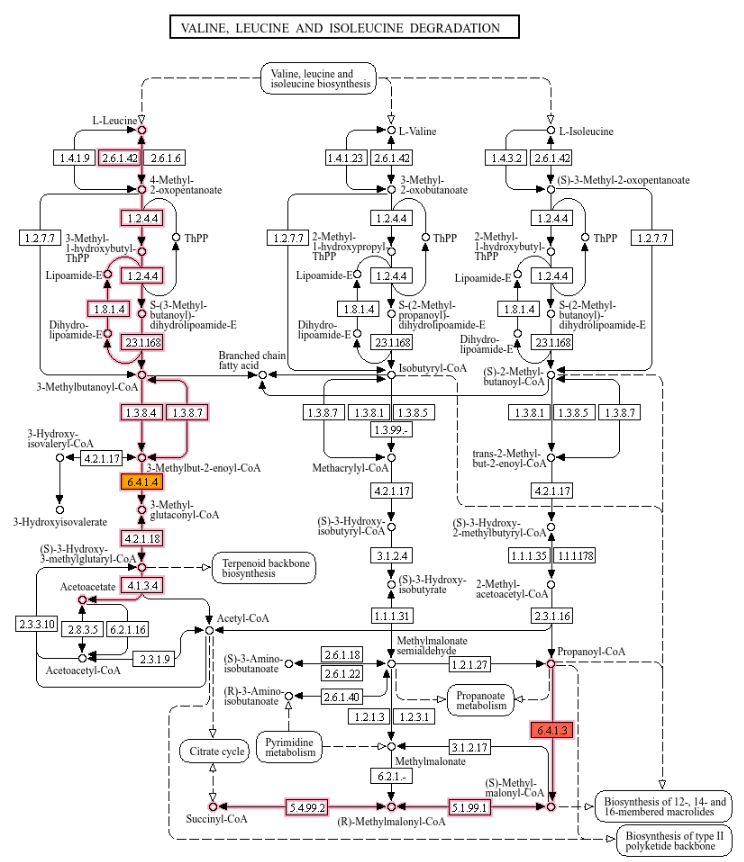
MCC1 и МСС2 напрямую задействованы в пути деградации лейцина, в то время как ЗССА и ЗССВ участвуют в углеводном обмене (метаболизм пропаноил-КоА).
Цикл деградации лейцина ведет к образованию Ацетил-КоА, который нужен для биосинтеза жирных кислот.
Выводы
Все картинки расположены в порядке перехода от одной схемы к другой (в овальчиках указаны процессы, к которым можно перейти - ищем нужный). Единственный отсутсвующий переход - от
пирувата к деградации лейцина. Должна быть связь пируват-синтез лейцина-деградация лейцина, но в синтезе лейцина не участвует ни один ген из исследуемых мной, поэтому данная схема была опущена.
Следовательно, раз я смогла найти связки между протекающими процессами метаболизма, то данные гены связаны функционально