Поиск de novo сигналов в ДНК Mycobacterium senriense
Подготовка данных
В данном практикуме я решила найти последовательность сайты посадки сигма-субъединицы РНК-полимеразы в геноме Mycobacterium senriense.
Для этого из файла генома GCF_019668465.1_ASM1966846v1_genomic.fna были
отобраны нуклеотиды, где точно должна быть сайт посадки - промоторы перед оперонами (100 нуклеотидов)
promotors.fasta, и где ее точно не должно быть (100 случайных нуклеотидов) -
negative.fasta
Список оперонов я получила, загрузив последовательность генома и список генов GCF_019668465.1_ASM1966846v1_genomic.gff в https://biocomputo.ibt.unam.mx/operon_mapper/
Для обучения взяла гены домашнего хозяйства housekeeping.fasta
Поиск сигнала с помощью MEME
meme promotors.fasta -dna -nmotifs 3 -minw 15 -maxw 35
Известно, что у сайта посадки сигма-субъединицы рибосомы консервативны участки -10 и -35, поэтому я ищу мотив похожей длины (15-35), чтобы попасть на консервативный участок
В результате получила
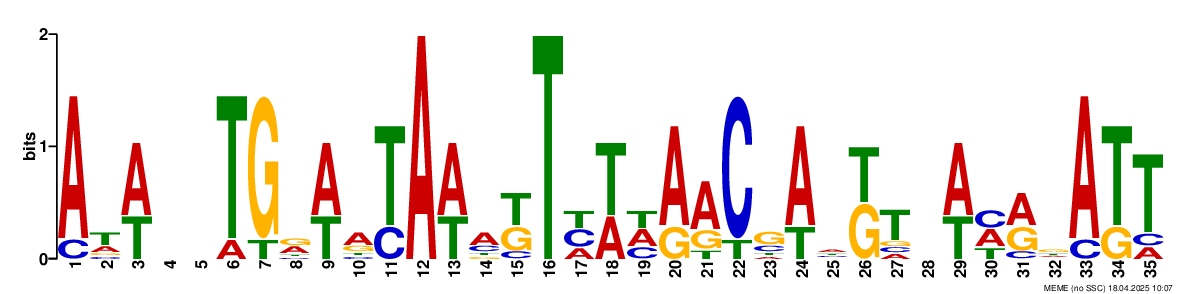
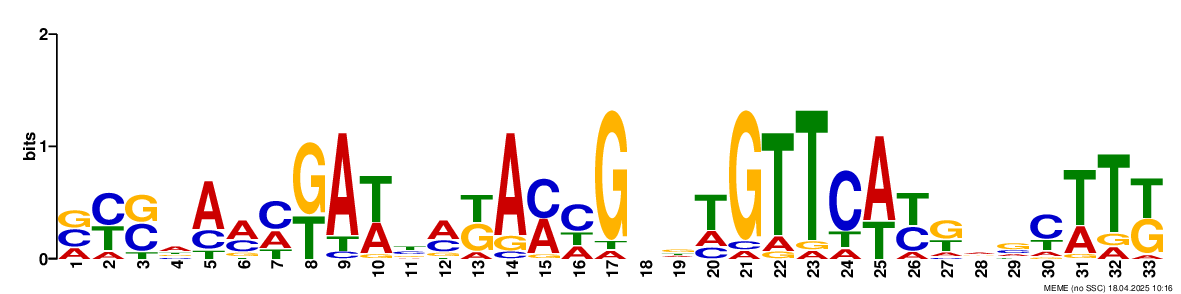
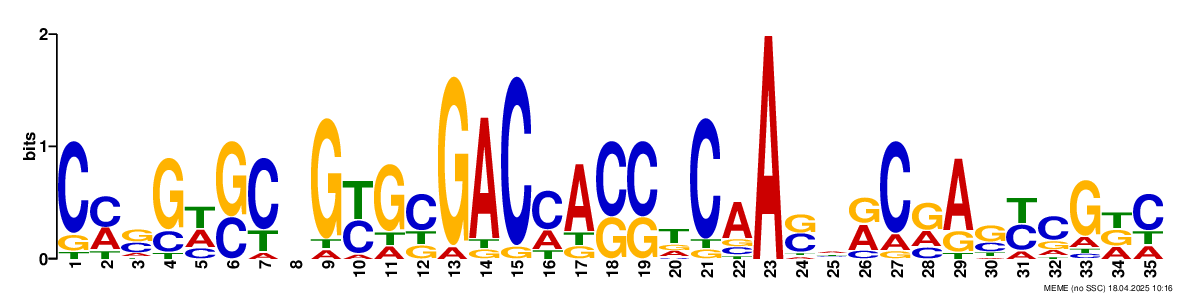
housekeeping.txt
Меня немного смутило, что длина достоверного по e-value участка граничная (35), поэтому я попробовала расширить поиск
и установить границы 5-50 по длине. Результат мне не понравился - все мотивы с очень высоким e-value.
Для дальнейшего поиска с fimo я использую logo1.
Оно больше остальных похоже на то, что нужно.
Поиск сигнала с помощью FIMO
Далее я тестирую fimo - посмотреть, у скольких последовательностей есть этот мотив, и может ли это быть случайной находкой - отрицательный контроль
fimo --norc -motif AWWNNTGRWRTAWMKTHWHAACSAHKTNWHRDATT -thresh 0.001 meme_out/meme.txt promotors.fasta
fimo --norc -motif AWWNNTGRWRTAWMKTHWHAACSAHKTNWHRDATT -thresh 0.001 meme_out/meme.txt negative.fasta
Получилось, что там, где эта последовательность должна быть - 57 из 5447, а в отрицательном контроле - 2 из 406 (столько генов домашнего хозяйтсва взяли, т.е. все). Значит, наш мотив естьв 1% "хороших" последовательностей и в 0.5% "плохих". Я бы сказала, что с такими результатами данный сигнал является слабым и незначительным в геноме Mycobacterium senriense