Мотивы в белках
Консервативные мотивы в выравнивании
В базе данных PFAM нужно найти подходящий для анализа домен. В нем должно быть не слишком много белков, при этом выравнивание seed должно содержать достаточно последовательностей для дальнейшего анализа. В итоге я остановился на гелиородопсине, AC в PFAM - PF18761.
Скачав выравнивание, я стал искать консервативные мотивы. В целом можно сказать, что в последовательностях достаточно много мотивов. Это ожидаемо, т.к. анализ литературы показал, что гелиородопсины содержат много консервативных позиций, которые необходимы для правильного улавливания света и последующей регуляции внутриклеточных процессов (хотя подробно функции этих белков еще не были изучены). Я выбрал крупный мотив ближе к С-концу белка. Jalview-кодировка этого мотива: LSLVAK[S,T][L,A,V]LAW. Он нашелся в 25 из 46 сиквенсов, везде только один раз. Это ожидаемо для такого большого и строго заданного мотива. С-конец этих белков находится снаружи клетки, вероятно высокая консервативность этого участка объясняется тем, что именно через него гелиородопсины регулируют некоторые внеклеточные белки (опять же об их функциях мало что известно, поэтому остается спекулировать)
Далее я решил найти этот паттерн в других белках. Для этого я воспользовался Prosite, который может по заданному паттерну производить поиск в базах данных. Сначала перевел свой мотив в формат prosite, получилось L-S-L-V-A-K-[ST]-[LAV]-L-A-W, а потом провел поиск по Swiss-Prot. Т.к. мой домен довольно редкий, а мотив довольно жестко задан, хитов не было. Я решил провести аналогичный поиск но уже по базе TrEMBL. Нашлось 111 белков, я сохранил их последовательности в формате FASTA, выровнял и нашел исходный мотив. Результаты представлены на рис. 1.
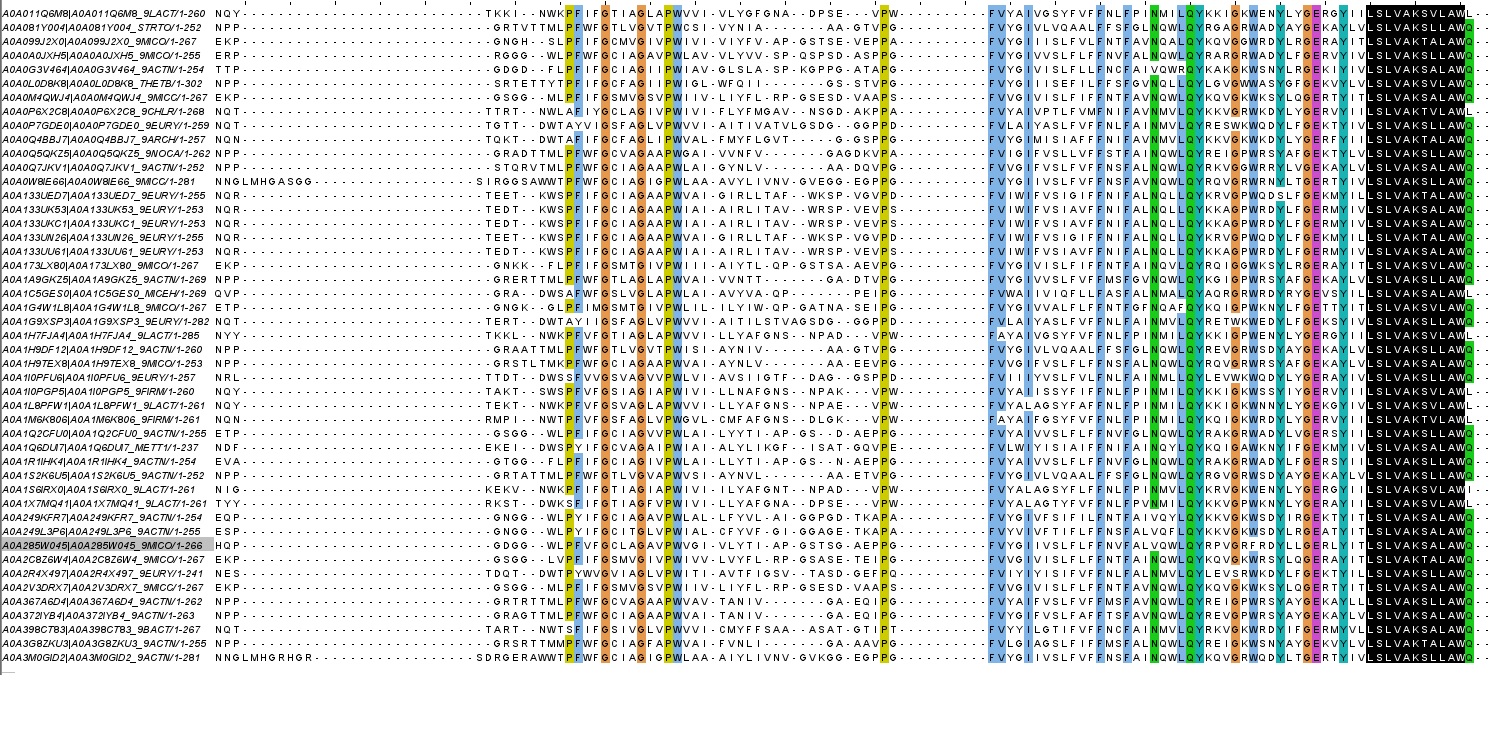
Ожидаемо мотивы в найденных последовательностях выровнялись друг на друга (опять же из-за того, что мотив большой и задан строго). Более того, у найденных последовательностей есть и другие консервативные участки, предшествующие описанному мотиву, которые совпадают с таковыми в исходном seed. Это позволяет с уверенностью говорить, что мы нашли близкие гомологи наших белков, вероятно это тоже гелиородопсины
В том же выравнивании найдите мотив, специфичный для одной клады филогенетического дерева
По полученному в предыдущем задании выравнивании я построил дерево методом NJ. В полученном дереве (которое я здесь показывать не буду из-за его громоздкости), я выбрал одну кладу, содержащую в себе 17 белков. Дерево этих 17ти белков вы можете увидеть по этой ссылке. К сожалению, когда я вывел выравнивание этих белков в отдельное окно, оказалось, что в ней мотив такой же, как и во всех последовательностях: LSLVAK[S,T][L,A,V]LAW. Картинку с выравниванием вы можете увидеть по этой ссылке.
Таким образом, мотив в этой кладе не специфичен для нее, а совпадает с мотивом всех выбранных ранее белков :(.
Поиск гомологов PSI-BLAST
Теперь составим семейство гомологов белковой последовательности, используя PSI-BLAST. Для этого я выбрал случайный АС- P74518. Это фактор активации гибернации рибосом из цианобактерии Synechocystis sp. Он усиливает димеризацию рибосом, переводя их в неактивное состояние, нужен для регуляции трансляции. Я провел поиск гомологов этого белка с помощью PSI-BLAST, всего потребовалось 5 итераций: после 4й результаты не поменялись, а 5ю я провел для верности. Таблицу, содержащую информацию о каждой итерации, вы можете видеть ниже.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 24 | P33987.1 | 3,00E-05 | - | - |
| 2 | 28 | P9WMA8.1 | 4,00E-06 | - | - |
| 3 | 28 | P24694.1 | 2,00E-20 | - | - |
| 4 | 28 | P24694.1 | 2,00E-20 | - | - |
| 5 | 28 | P24694.1 | 3,00E-20 | - | - |
По этой ссылке вы можете найти выдачу PSI-BLAST после последней итерации. Давайте сделаем вывод из полученных данных. Все найденные белки имеют одинаковые имена (или такие, что по ним понятно, что делают они одно и то же), это хороший знак. После 3й итерации результаты сошлись (то есть в следующих итерациях не менялись), это хороший знак и дает нам понять, что скоре всего то, что мы нашли, действительно является белковым семейством. Еще один критерий качества найденного семейства- разница E-value между худшей "правильной" находкой и лучшей "неправильной". Так получилось, что для моего семейство не было белков, со значением E-value выше порогового 0.005. Это значит, что выбранная группа белков очень сильно отличается от всех остальных и, вероятнее всего, действительно составляет семейство.
Таким образом, можно с уверенностью говорить, что найденная группа образует семейство гомологичных белков.