Домены и профили
HMMER2
Построение HMM-профиля и поиск
Для анализа я выбрал двухдоменную архитекутуру из PFAM (AC- Q8GCX7), первый домен- это обратная транскриптаза (AC- IPR000477), а второй- HNH-эндонуклеаза (АС- IPR002711). Белков в этом подсемействе 195 штуки. Я скачал их в формате fasta и стал подготавливать к созданию HMM-профиля.
Для начала я выровнял их полные последователеьности, результат вы можете скачать по ссылке: full.fasta.
Далее по координатам из PFAM я нашел N-конец первого домена и C-конец второго домена и вывел эту область в отдельный файл и выровнял еще раз. Результат находится в этом файле: выравнивание двух доменов.
После этого из полученного выравнивания я удалил несколько последовательностей, которые сильно отличались от других (дают большие гэпы во многих местах выравнивания) и затем удалил последовательности, похожие на 93% или более. Полученную совокупность последовательностей я перевыровнял, результаты находится в этом файле: выравнивание двух доменов после чистки.
По последнему полученному файлу мы строим HMM-профиль и калибруем его:
- hmm2build hmmout subfamily_without_redundancy_align.fasta
- hmm2calibrate hmmout
Теперь выполним поиск:
- hmm2search --cpu=1 hmmout subfamily_full.fasta > hmm_results.txt
Вот что получили в выдаче:
Анализ HMM-профиля
Для дальнейшей обработки пришлось немного переписать таблицу с выдачей и убрать все ненужное, используемые далее файлы:
- AC из выравнивания full
- AC белков, используемых для построения HMM-профиля
- AC белков доменной архитектурой
- Исправленная таблица выдачи HMM поиска
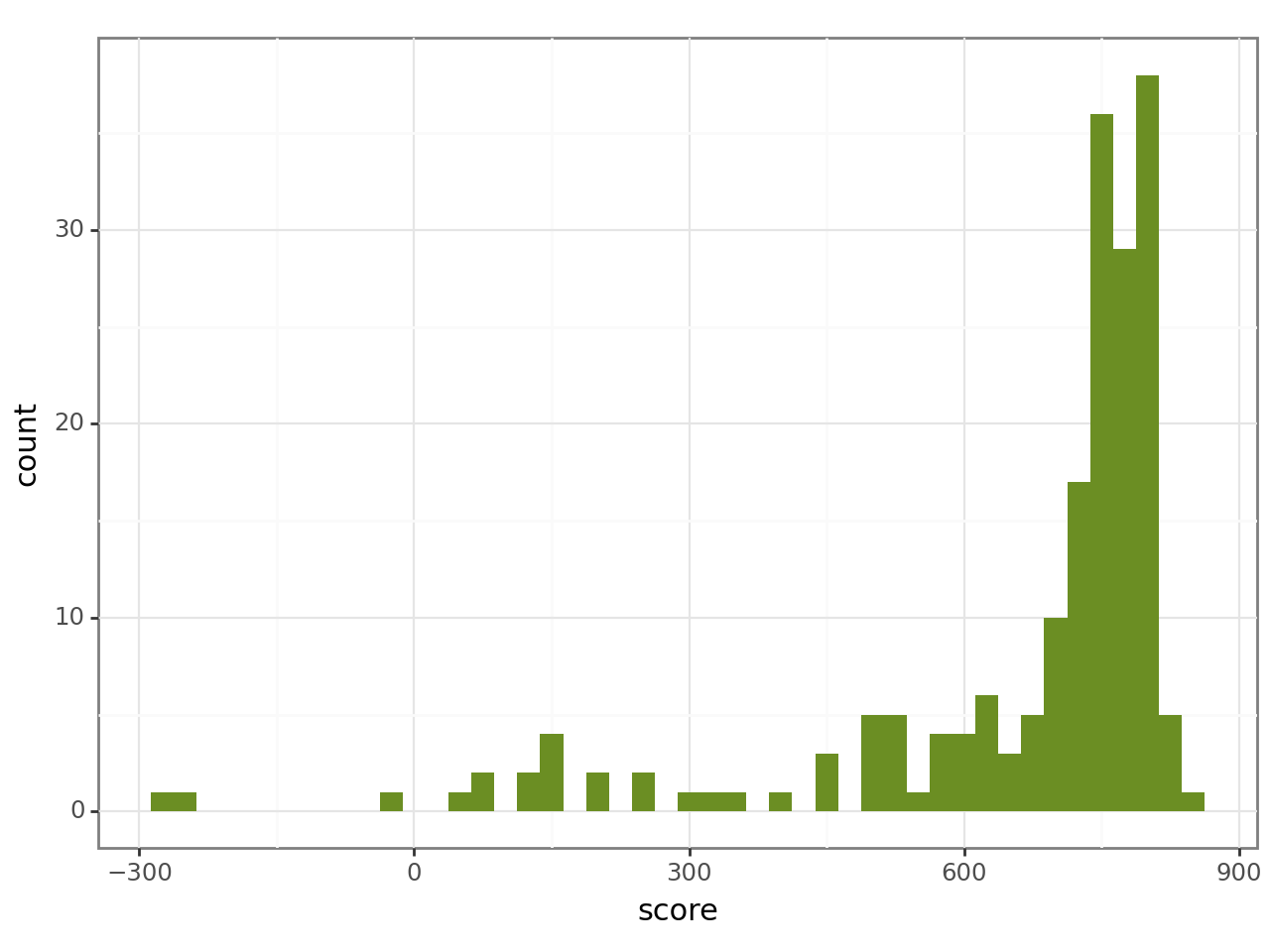
Рис. 1 Гистограмма весов белков, видно, что за порог можно взять значение около 600
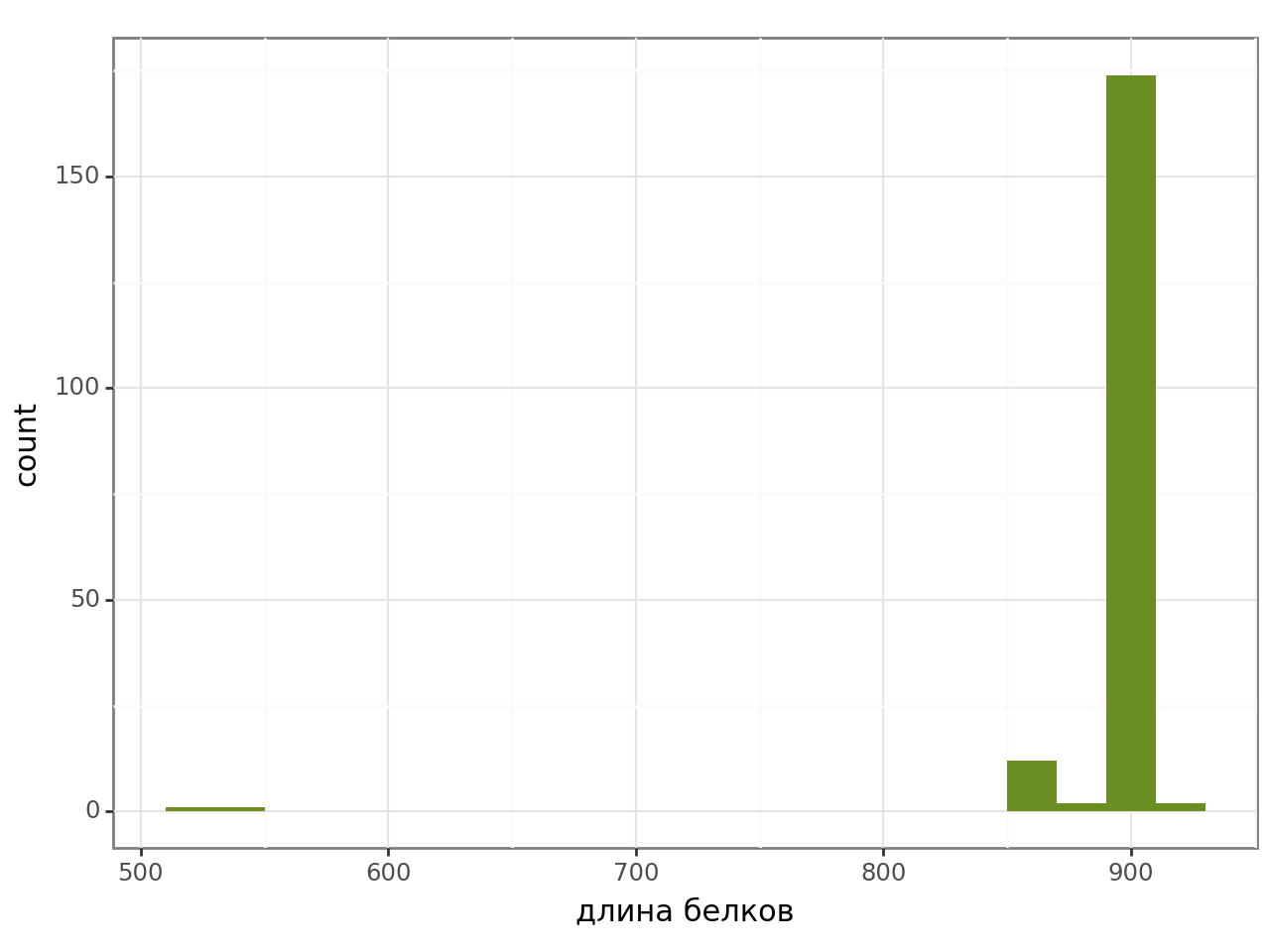
Рис. 2 Гистограмма длин белков, почти все белки имеют одинаковую длину
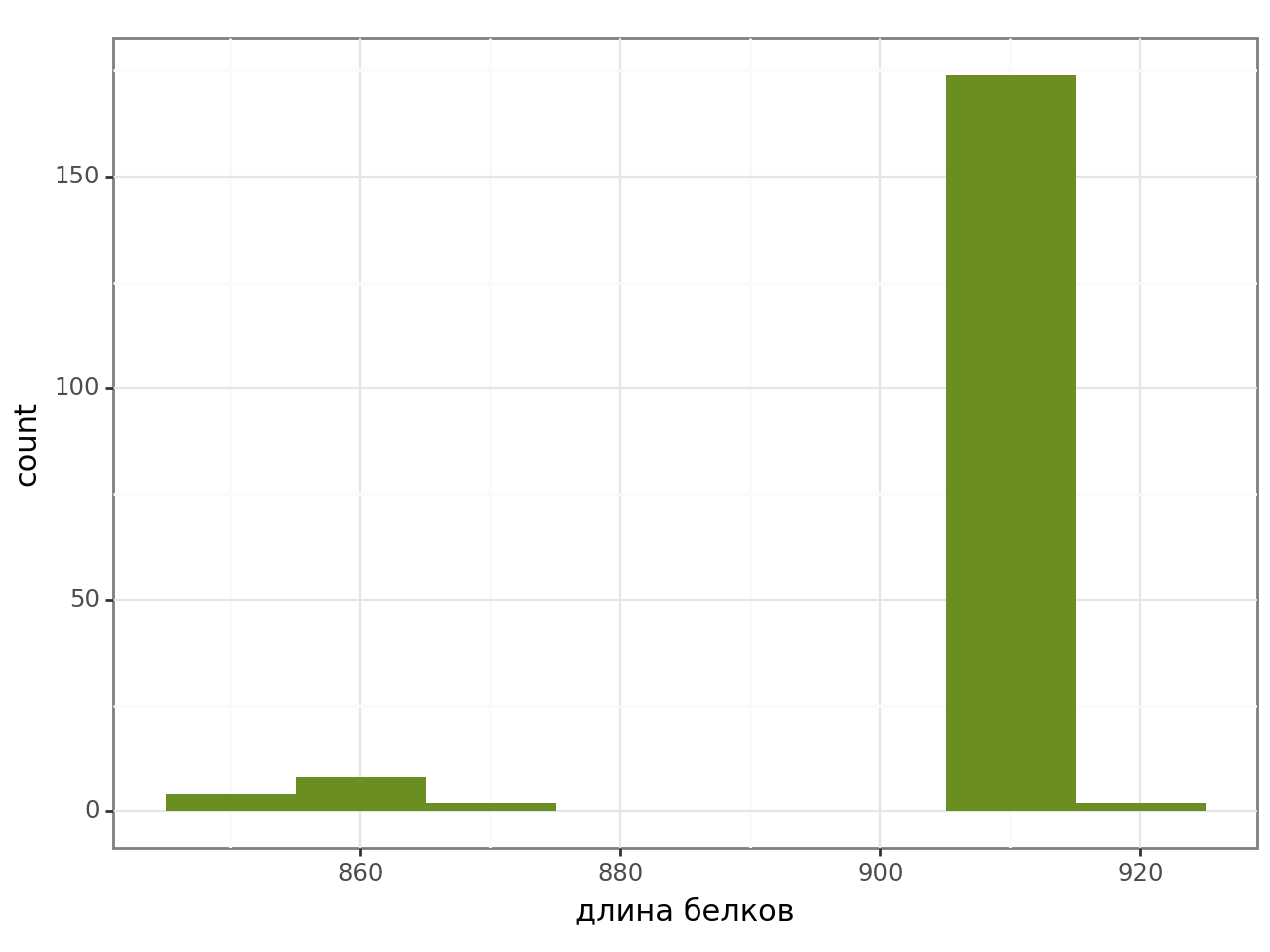
Рис. 3 Гистограмма длин белков, удалил белки с длиной меньше 600, чтобы лучше было видно остальное распределение