Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для анализа необходимо было выбрать белок с трансмембранными бета-слоями из библиотеки OPM. Просмотр случайных белков не дал результата, т.к. у всех трансмембранной областью являлась альфа-спираль. Поэтому я ввел в поиск запрос beta-barrel и нашел белок который буду далее анализировать- BamA бета-бочонок в комплексе с даробактином. Название на английском- BamA beta-barrel with darobactin, идентификатор PDB- 7nre, идентификатор UniProt- P0A940, белок выделен из E.Coli. Этот белок является частью белкового комплекса, располагающегося во внешней мембране; основная функция этого комплекса (согласно UniProt)- встраивание в мембрану белков, имеющих в своей структуре бета-бочонок.
У моего белка 15 трансмембранных участков, вот их координаты:
- 425- 431
- 439- 446
- 454- 463
- 468- 473
- 484- 492
- 509- 517
- 525- 532
- 569- 579
- 591- 599
- 608- 619
- 630- 638
- 711- 720
- 735- 742
- 768- 776
- 783- 788
Далее я использовал DeepTMHMM для предсказания трансмембранных доменов моего белка по первичной последовательности. Первичную последовательность в формате FASTA я получил из UniProt. Результаты работы DeepTMHMM представлены на Рис. 1.
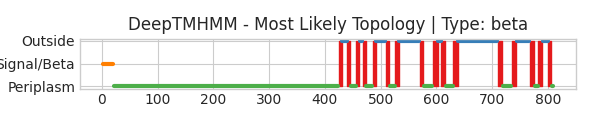
Разберемся подробнее с тем, что представлено на Рис. 1. Зеленая линия указывает на часть белка, находящаяся в периплазме, красным- трансмембранная часть, синяя- часть белка снаружи клетки. DeepTMHMM также выдает предсказанные координаты трансмембранных доменов:
- 425- 430
- 440- 445
- 456- 462
- 468- 473
- 486- 491
- 509- 515
- 526- 532
- 570- 576
- 593- 602
- 609- 616
- 630- 638
- 710- 718
- 735- 743
- 768- 775
- 783- 790
- 801- 806
Полную выдачу в формате gff3 вы можете скачать по ссылке. Мы видим, что координаты предсказанных двумя способами трансмембранных доменов почти полностью совпадают (+- пара аминокислот), за исключением наличия 'лишнего' трансмембранного домена на С-конце в предсказании DeepTMHMM. Отличия в границах трансмембранных доменах может быть объяснена тем, что включение или не включение крайних аминокислот в структуру трансмембранного домена не сильно сказывается на энергии (или разные модели по разному ее рассчитывают), из-за чего мы видим отличия в границах.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для анализа мне был выдан белок HemX (видимо на русский его можно перевести как ГемХ), идентификатор в Swiss-Prot HEMX_BREBE, название организма- Brevibacillus brevis. Функция- участие в синтезе HemL (в общем участие в биосинтезе порфирина).
Предскажем по аминокислотной последовательности белка трансмембранные домены используя DeepTMHMM. Результаты представлены на рис. 2.
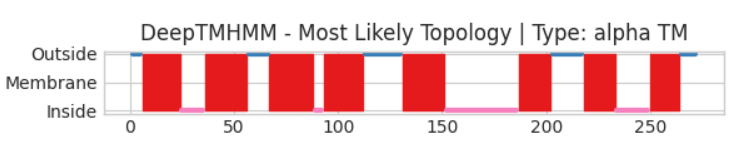
То, как интерпретировать выдачу DeepTMHMM описывалось ранее, тут все аналогично. Посмотрим на получившиеся трансмембранные домены, их 8 штук:
- 6- 24
- 36- 56
- 67- 88
- 93- 112
- 131- 151
- 187- 202
- 218- 233
- 250- 264
Полную выдачу в формате gff3 вы можете скачать по ссылке.
Далее предскажем трансмембранные с помощью PPM 3.0. Параметры: тип мембраны- внутренняя мембрана грамположительных бактерий (к ним относится Brevibacillus brevis), allow curvature- да (видимо если выставить этот параметр на 'да', мы позволяем программе немного передвигать атомы и связи, разумно поставить параметр на 'да', т.к. изначально pbd файл был получен просто предсказанием структуры с помощтю AlphaFold, а в нем могут быть ошибки), топология N-конца- снаружи (просто надо указать, снаружи или внутри клетки находится N-конец белка, согласно выдаче DeepTMHMM N-конец снаружи, поэтому так и будем считать дальше). Получились следующие трансмембранные домены:
- 4- 27
- 32- 56
- 69- 89
- 93- 111
- 127- 152
- 180- 202
- 215- 237
- 244- 263
Полную выдачу в формате pdb вы можете скачать по ссылке. Результаты отличаются значительнее, чем при сравнении белка с бета-листами, количество трансмембранных доменов все еще совпадает, но границы доменов в некоторых случаях отличаются уже на 3-5 аминокислот. Если посмотреть на предсказание AlphaFold, то можно увидеть, что границы трансмембранных доменов имеют меньшую точность предсказания, что объясняет различия в длине трансмембранных доменов, предсказанных двумя способами.