Базы данных STRING и Reactome
STRING
Для анализа мне был выдан список из 40 ID генов, полный список вы можете увидеть по ссылке ссылке. Пробежавшись по нему глазками, я узнал только два белка- MAOA и MAOB, это ферменты моноаминоксидазы, осуществляющие окислительное дезаминирование моноаминов.
Для начала воспользуемся датабазой STRING. Она позволяет найти некоторые взаимосвязи между белками (используя информацию из других баз данных) и выражает их в форме графа. Загрузим наши ID в STRING и посмотрим что он выдаст интересного. Исходный граф не очень хорошо понимается из-за избытка представленной информации, поэтому я сразу попробовал разбить его на кластеры используя встроенный функционал STRING. Я использовал алгоритм MCL clustering, т.к. судя по описанию, он должен находить оптимальное число кластеров и по ним распределять все белки. У него есть один параметр- inflation density. Он как-то связан с числом получающихся кластеров, чем больше этот параметр, тем больше кластеров в итоге получится. Я его покрутил и остановился на значении 3, т.к. при нем не образовывалось кластеров, с одним белком в составе (по идее такие кластеры не очень информативные, поэтому я старался их количесто минимизировать). Полученный граф представлен на рис. 1.

Посмотрим что содержится в образовавшихся кластерах:
- В зеленом кластере 5 белков, 4 из них отдалены от красного кластера, а пятый лежит прямо на его границе. Поэтому можно предположить, что среди этих пяти белков, 4 белка, лежащих в стороне, будут более похожи друг на друга, чем на пятый белок, лежащий возле красного кластера. Так и получается- MAOA, MAOB, AOC2 и AOC3 являются аминоксидазами, а лежащая в стороне ALDH7A1- окисляет полуальдегиды; но все вместе они ответственны за метаболизм аминокислот.
- Синий кластер составляют белки, работающие с фосфорилированием- 4 мутазы (PGAM1, PGAM2, PGAM4, BPGAM) и одна киназа (GLYCTK); помимо этого они все принимают участие в метаболизме некоторых аминокислот.
- Красный кластер самый большой, в нем 30 аминокислот. Почти все они являются цитоплазматическими ферментами, которые ответственны за метаболизм аминокислот. Стоит отметить, что при повышении параметра inflation density, в первую очередь из этого кластера отсоединяются одиночные белки в самостоятельные кластеры. Это в очередной раз свидетельствует о большом разнообразии белков, представленных в нем.
Ну вроде из сильно интересного все, можно было попробовать другие способы кластеризации, но там +- похожая картина получается, только кластеры отсоединяются в разном порядке (ну и всегда много кластеров с одним представителем, что не есть хорошо).
Reactome
Далее я решил изучить свой набор белков с помощью базы данных Reactome, т.к. в ней легче понимать функциональное разнообразие белков, чем смотря на граф. Полная выдача представлена на рис. 2.
Мы видим, что (как и ожидалось), наши белки разбросаны по большому количеству групп. Более детальная информация представлена на рис. 3-6.
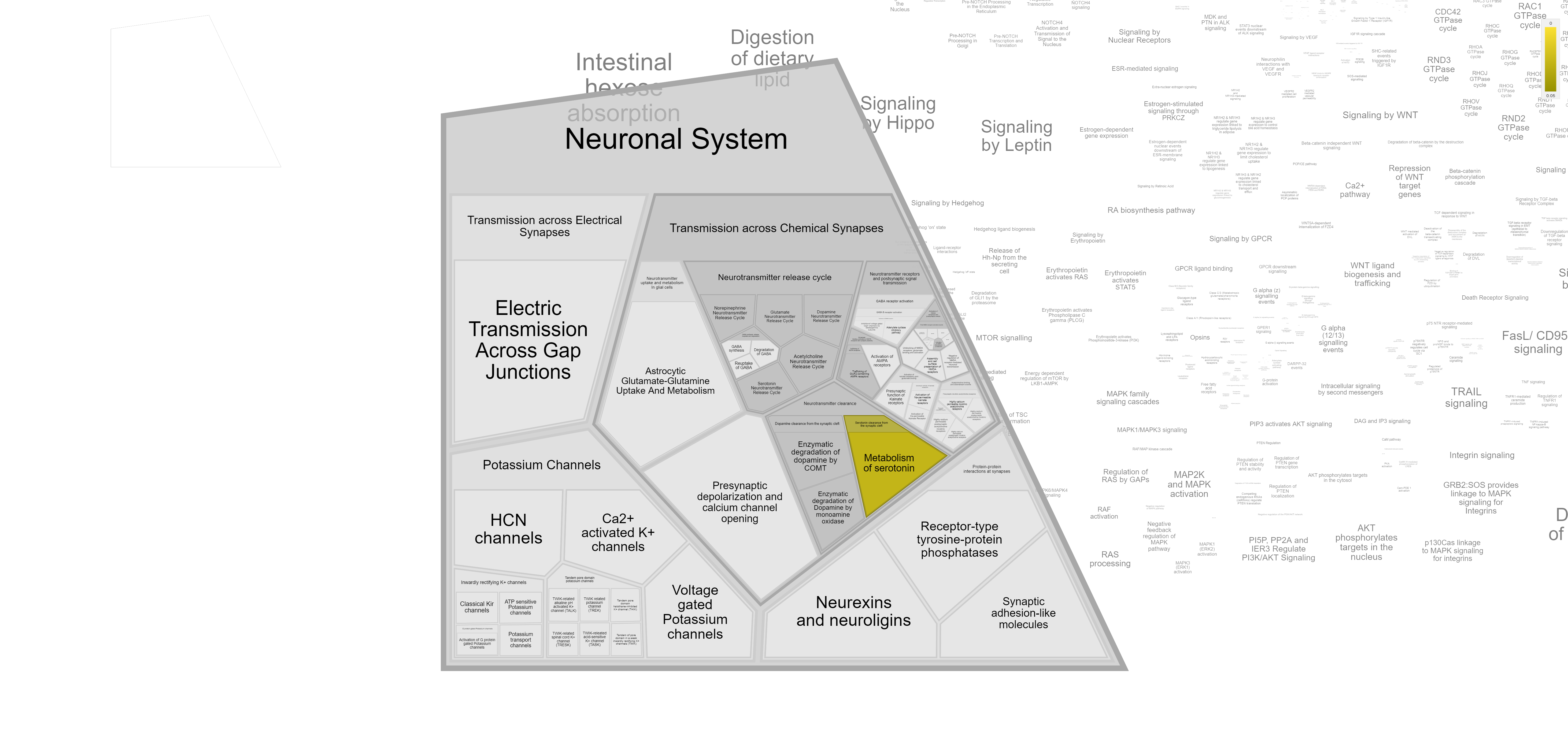
Здесь представлены белки, ответственные за метаболизм серотонина. Если быть точнее, то здесь находится уже упоминавшаяся в начале работы моноаминоксидаза.
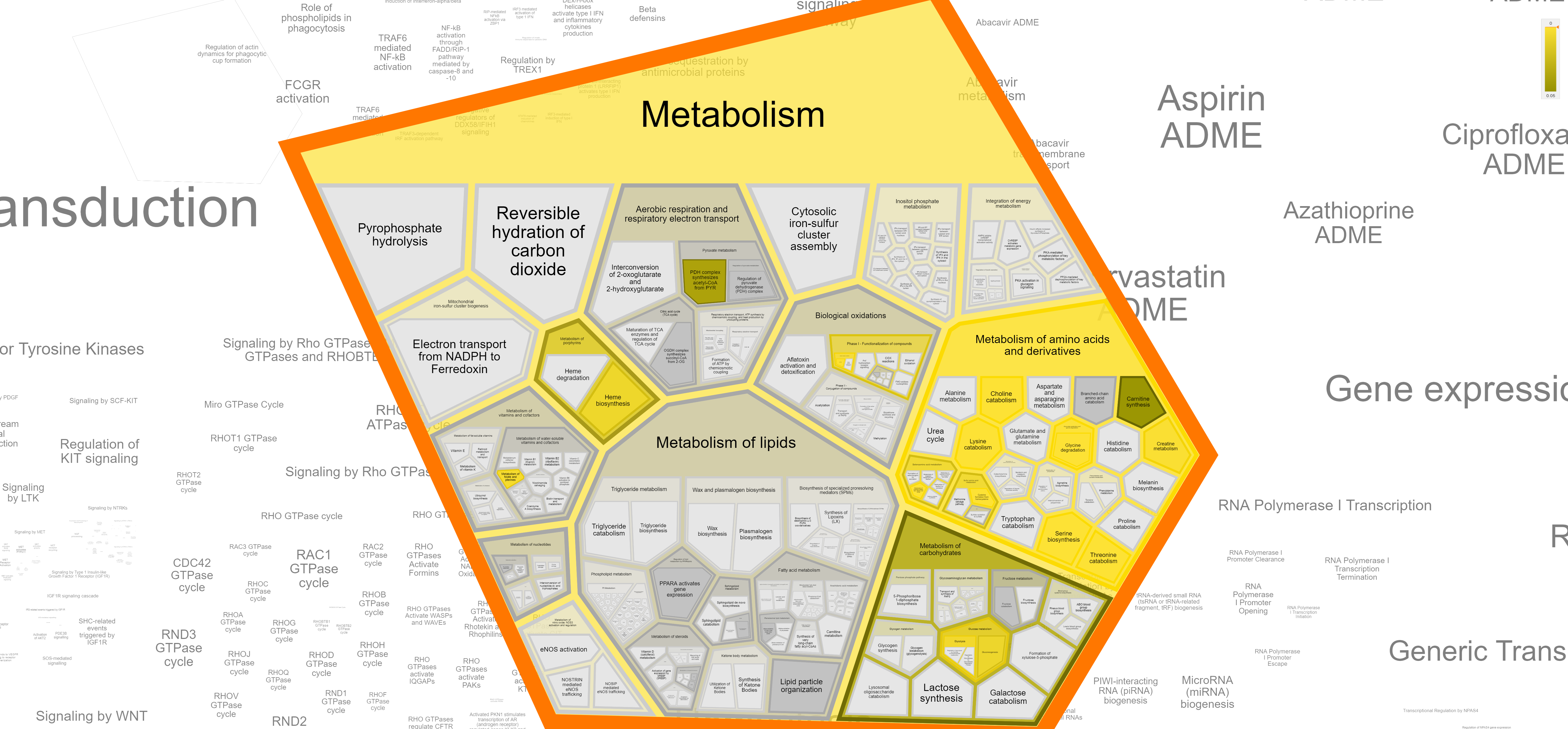
Здесь находится самое большое количество белков с очень разнообразными функциями, включая ферменты ответственные за метаболизм углеводов, аминокислот, некоторых витаминов и гема, подробно говорить про каждую группу здесь мне кажется не имеет смысла.
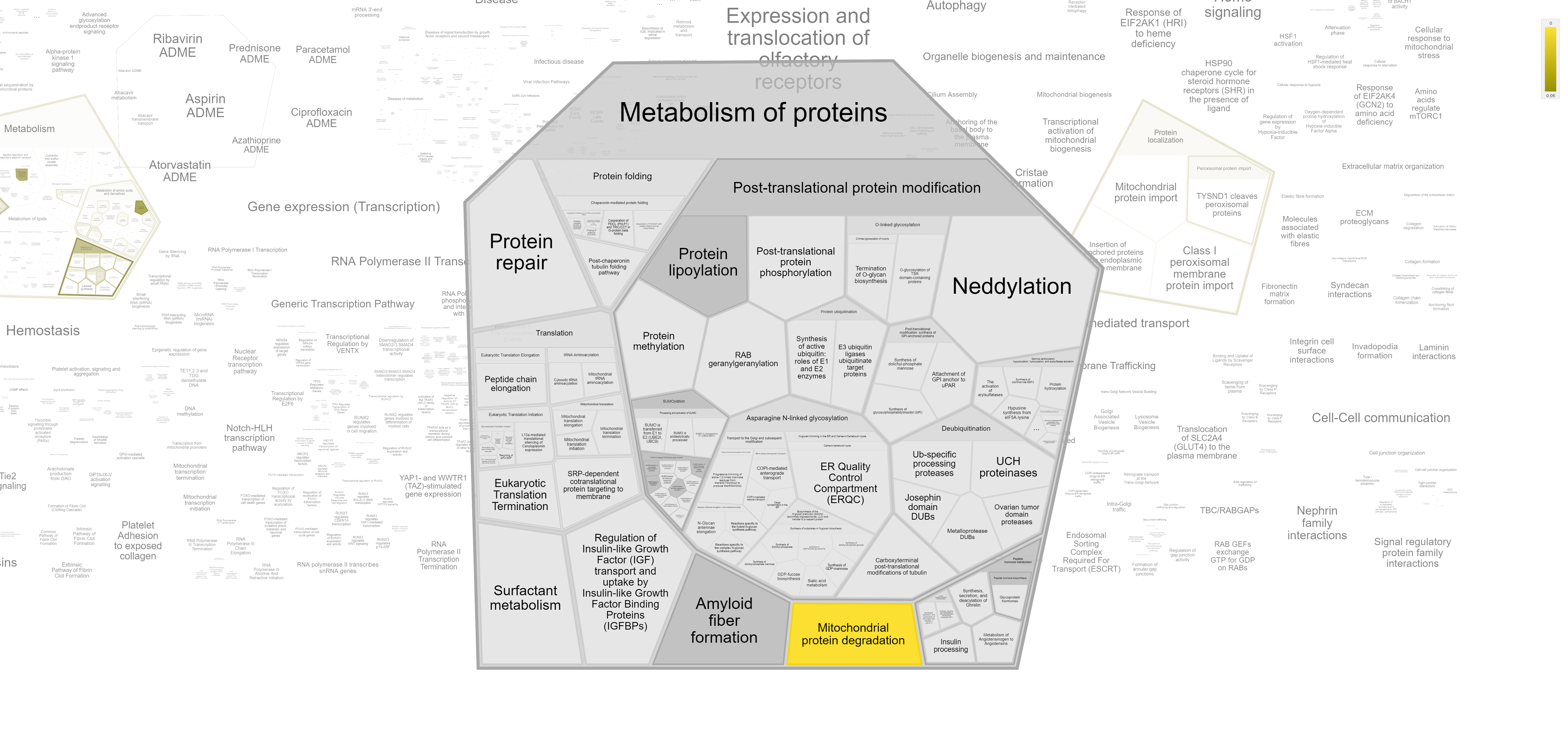
Здесь есть только пара белков, ответственных за деградацию митохондриальных белков
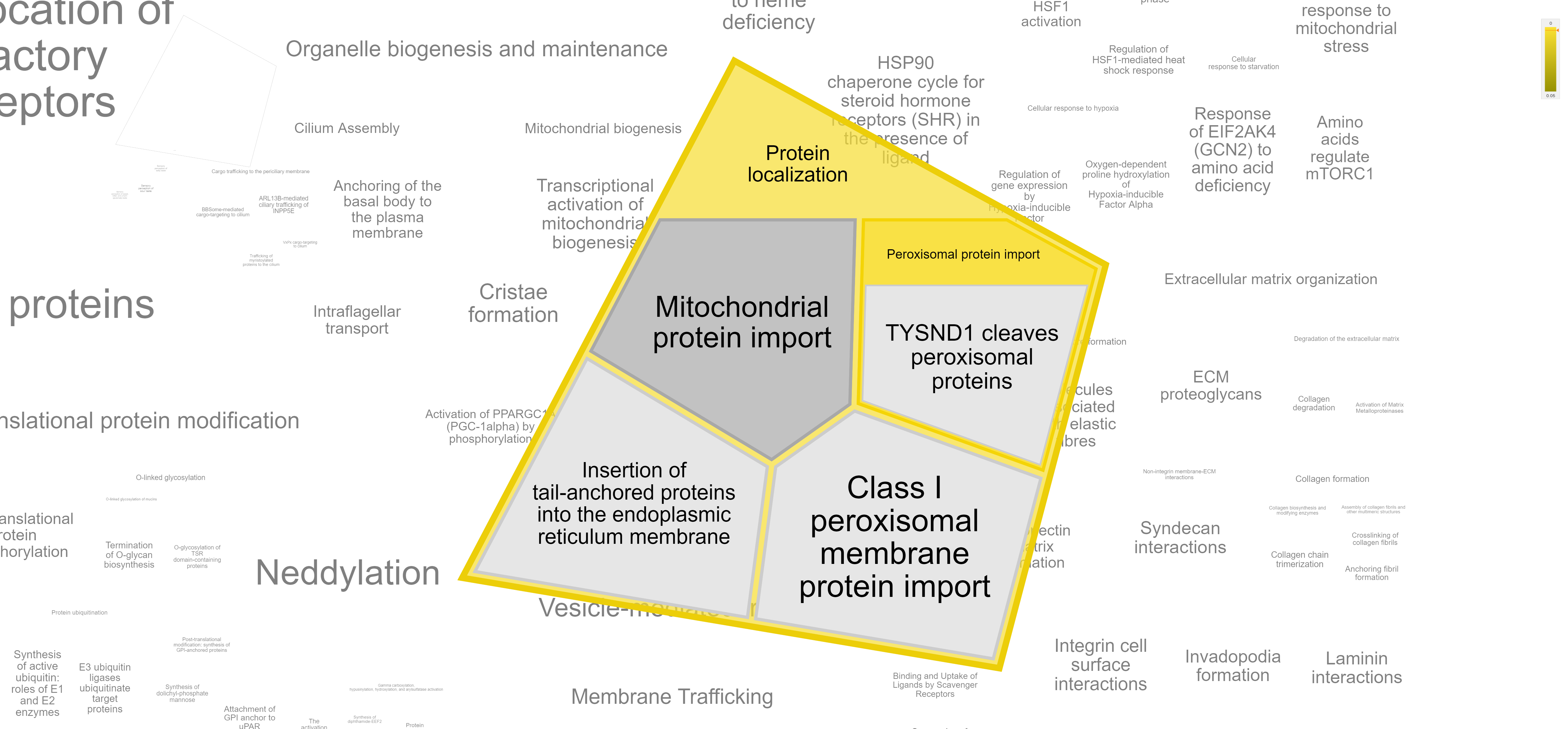
Здесь мы видим белки, которые ответственны за импорт в пероксисомы
В целом, результаты совпали с анализом при помощи STRING- у наших белков множество непохожих функций.