Сигналы в геноме
Задание 1. Rho-зависимая терминация транскрипции у бактерий
Для этого задания я выбрал геном бактерии E. coli, в котором я буду искать сайты Rho-зависимой терминации транскрипции. Название- сайт связывания ро-фактора (по-другому называется ро-утилизационным (rut) сайтом). По своей сути сигнал- это одноцепочечный регион (около 70 нуклеотидов) с высоким C/G соотношением (то есть он богат цитозином и беден гуанином), он находится синтезируемым в РНК, выше (upstream) фактической последовательности терминатора. Адресован он Ро-фактору, который, связываясь с ним, догоняет и 'выталкивает' РНК-полимеразу, терминируя транскрипцию. Сила данного сигнала, как мне кажется, должна быть довольно высока, т.к. в обратном случае будут образовываться слишком длинные тринскрипты, что неоптимально для клеток.
Для поиска примеров сигнала я воспользовался кодом из рекомендованной нам статьи за авторством Di Salvo et al. Вот ссылка на статью и на код. Он находит в геноме последовательность с высоким соотношением C/G (больше 1), находит в этой области окно длиной 78 нуклеотидов с максимальным соотношением C/G и особым паттерном расположения цитозинов, далее в окне 150 нуклеотидов после этой последовательности пытается найти шпильку (путем поиска палиндромов) или консенсусную последовательность, которые бы тормозили РНК-полимеразу. Запустив код на выбранном мною геноме E. coli я получил список предполагаемых последовательностей rut-сайтов (и не только, но интересует нас сейчас только это), вот некоторые из них:
- ACAGATAAAAATTACAGAGTACACAACATCCATGAAACGCATTAGCACCACCATTACCACCACCATCACCATTACCAC
- CGGTCACAACGTTACTGTTATCGATCCGGTCGAAAAACTGCTGGCAGTGGGGCATTACCTCGAATCTACCGTCGATAT
- TGTCCTACCAGGAAGCGATGGAGCTTTCCTACTTCGGCGCTAAAGTTCTTCACCCCCGCACCATTACCCCCATCGCCC
- TCACGCGCCCGTATTTCCGTGGTGCTGATTACGCAATCATCTTCCGAATACAGCATCAGTTTCTGCGTTCCACAAAGC
- TTCTTTGCCGCACTGGCCCGCGCCAATATCAACATTGTCGCCATTGCTCAGGGATCTTCTGAACGCTCAATCTCTGTC
- AGAATAAACATATCGACTTACGTGTCTGCGGTGTTGCCAACTCGAAGGCTCTGCTCACCAATGTACATGGCCTTAATC
- CGCGAAGGTTTCCACGTTGTCACGCCGAACAAAAAGGCCAACACCTCGTCGATGGATTACTACCATCAGTTGCGTTAT
- CCTGTGCTGCCCGCAGAGTTTAACGCCGAGGGTGATGTTGCCGCTTTTATGGCGAATCTGTCACAACTCGACGATCTC
- CAGAGACATTCAGTCTCAACAACCTCGGACGCTTTGCCGATAAGCTGCCGTCAGAACCACGGGAAAATATCGTTTATC
Полную выдачу вы можете найти по ссылке.
Задание 2. PWM и поиск новых сайтов по PWM
Для анализа в этом задании я выбрал последовательность Козак в геноме человека. Я воспользовался кодом, любезно предоставленный мне старшекурсниками, который принимает на вход таблицу human-genes.tsv и выдает следующие файлы:
- kozak-learn.fasta, kozak-test.fasta и pseudokozak1.fasta – файлы с последовательностями для обучения, тестирования и негативного контроля соответственно
- result.csv – позиционная весовая матрица с псевдоттсчетами (PWM), построенная на материале обучения
- ic.csv – матрица информационного содержания IC(b,j) для выравнивания из материала обучения
- hist.svg и hist.png – изображения гистограммы весов
- check.csv – таблица результатов проверки
В результате работы скрипта получено по 100 последовательностей для обучения, тестирования и отрицательного контроля. На материале обучения построена следующая позиционная весовая матрица с псевдоотсчетами (PWM):
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.35 | -0.47 | -0.46 | -0.21 | 0.42 | -0.03 | -0.50 | 1.22 | -6.60 | -6.60 | -0.27 | -0.18 | -0.54 |
| T | -0.37 | -0.34 | -0.34 | -0.76 | -1.25 | -0.59 | -1.38 | -6.60 | 1.22 | -6.60 | -0.68 | -0.40 | -0.37 |
| G | 0.30 | 0.57 | 0.43 | 0.12 | 0.46 | 0.10 | 0.33 | -6.24 | -6.24 | 1.58 | 0.81 | -0.03 | 0.52 |
| C | 0.42 | 0.17 | 0.34 | 0.65 | -0.38 | 0.46 | 0.82 | -6.24 | -6.24 | -6.24 | -0.23 | 0.56 | 0.31 |
На основе данной таблицы были посчитаны веса всех последовательностей и построена гисторгамма распределения (Рис. 1).

Исходя из результатов, представленной на гистограмме, порогом правильной находки был выбран вес равный 4, и с ним составлена таблица результатов проверки (Таблица 2).
| Обучение | Положительный контроль | Отрицательный контроль | |
|---|---|---|---|
| Cигнал(+) | 368 (73.6%) | 369 (73.8%) | 154 (30.8%) |
| Сигнал(-) | 132 (26.4%) | 131 (26.2%) | 346 (69.2%) |
Веса последовательностей из материала обучения и материала тестирования имеют сходные распределения, в то время как веса последовательностей негативного контроля заметно сдвинуты влево. Однако эти распеделения в значительно перекрываются, поэтому ошибки первого и второго рода при тестировании велики (30.4% и 23.6% соответственно).
Далее была получена матрица информационного содержания (Таблица 3).
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.10 | -0.13 | -0.12 | -0.07 | 0.27 | -0.01 | -0.13 | 1.76 | 0.00 | 0.00 | -0.09 | -0.06 | -0.13 |
| T | -0.11 | -0.10 | -0.10 | -0.15 | -0.15 | -0.14 | -0.15 | 0.00 | 1.76 | 0.00 | -0.15 | -0.11 | -0.11 |
| G | 0.12 | 0.30 | 0.20 | 0.04 | 0.22 | 0.03 | 0.13 | 0.00 | 0.00 | 2.29 | 0.54 | -0.01 | 0.26 |
| C | 0.19 | 0.06 | 0.14 | 0.37 | -0.08 | 0.21 | 0.55 | 0.00 | 0.00 | 0.00 | -0.06 | 0.29 | 0.13 |
| IC(j) | 0.09 | 0.13 | 0.11 | 0.18 | 0.26 | 0.09 | 0.40 | 1.76 | 1.76 | 2.29 | 0.25 | 0.10 | 0.1 |
И наконец, с использованием сервиса WebLogo 3 была построена LOGO-диаграмма (Рис. 2) для последовательности Козак.
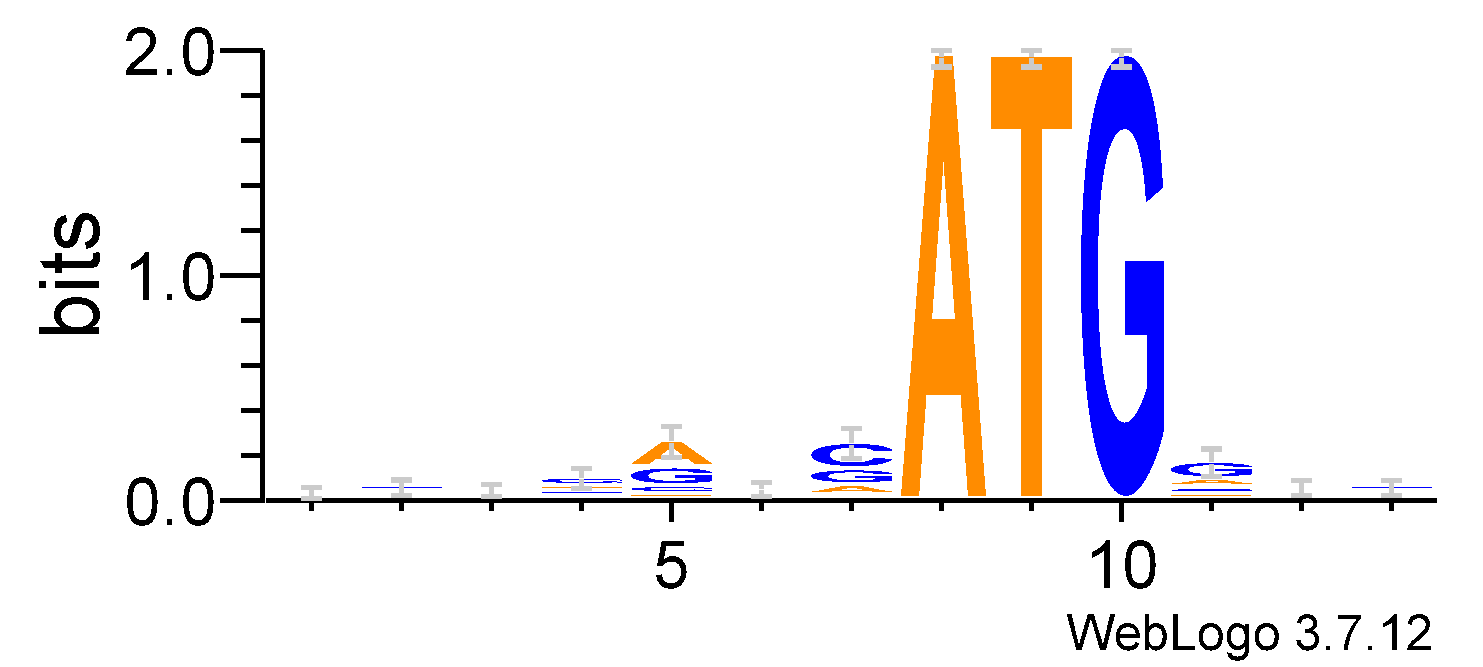
По результатам проведенных экспериментов, можно сделать вывод, что последовательность Козак обладает довольно слабым, но тем не менее консенсусом.