Анализ и очистка чтений
Была проанализировнна 13-ая хромосома.
Было получено 12155 чтений длинной 39-100 нуклеотидов.
Как видно из Рис.1 качество чтений до отчистки оставляет желать лучшего, особенно хвосты последних ридов. Работа программы Trimmomatic, которая убрала нуклеотиды качеством ниже 20 и чтения длиной меньше 50 нуклеотидов, помогла исправить "отросшие" усы у боксов, оставив после себя 11933 рида. Результат ее работы можно видеть на Рис.2.
GC-content также показывает качество ридов, в случае с нашей хромосомой он получился далек от нормального распределения, поэтому были использованы разные параметры для работы Trimmomatic в целях исправить эту проблему - не помогло: график становился более гладким, но нормального распределения достичь не удалось.
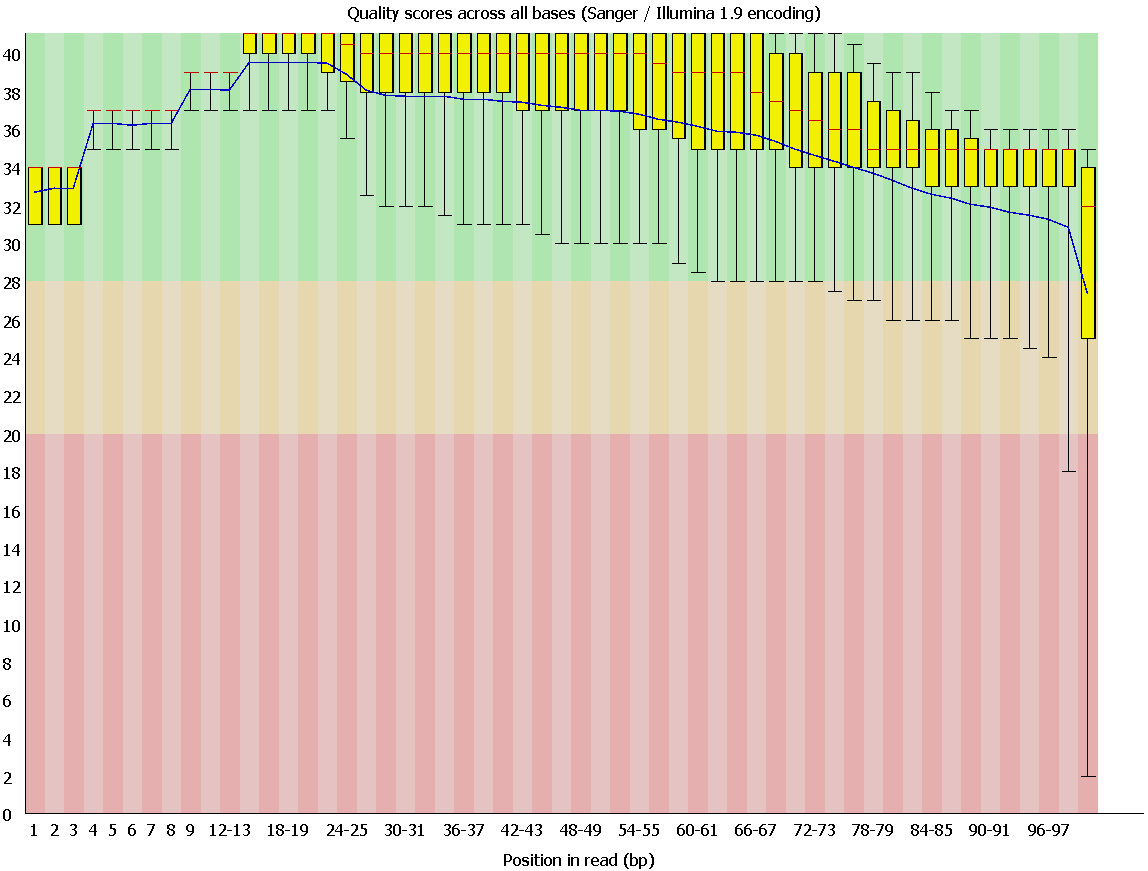
Рис. 1 Качество чтений до отчистки
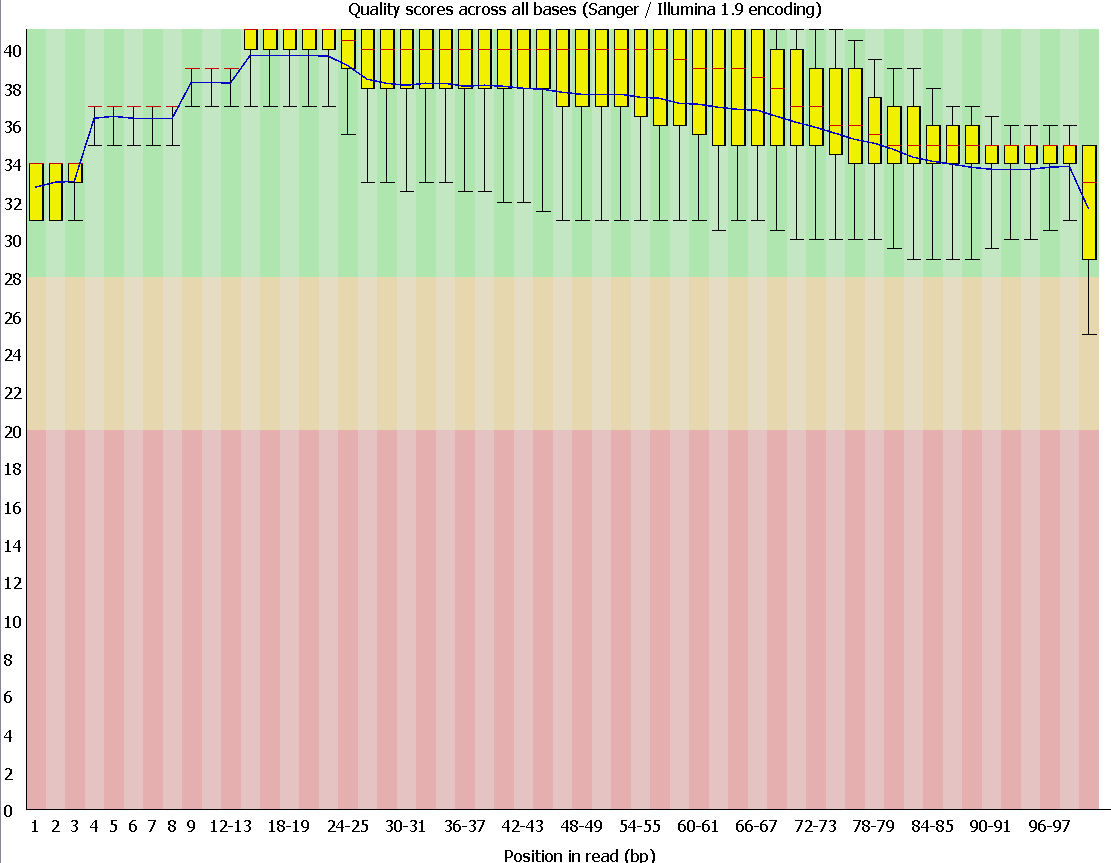
Рис. 2 Качество чтений после отчистки